wgcna专题
R语言绘制动态网络图Network教程WGCNA
今天分享的笔记是使用NetworkD3对WGCNA的共表达网络进行可视化,创建交互式动态网络图,展示基因之间的相互关系,可以用于转录组或者其他调控网络展示。 加权基因共表达网络分析 (WGCNA, Weighted correlation network analysis)是用来描述不同样品之间基因关联模式的系统生物学方法,可以用来鉴定高度协同变化的基因集,并根据基因集的内连性和基因集与表型
7+非肿瘤+WGCNA+机器学习+诊断模型,构思巧妙且操作简单
今天给同学们分享一篇生信文章“Platelets-related signature based diagnostic model in rheumatoid arthritis using WGCNA and machine learning”,这篇文章发表在Front Immunol期刊上,影响因子为7.3。 结果解读: DEGs和血小板相关基因的鉴定 作者通过R的“GEOquery
孟德尔随机化+WGCNA+预后模型,7+轻松get
今天给同学们分享一篇生信文章“Exploring the causality and pathogenesis of systemic lupus erythematosus in breast cancer based on Mendelian randomization and transcriptome data analyses”,这篇文章发表在Front Immunol期刊上,影响因子为
7+WGCNA+机器学习+实验+泛癌分析,多要素干湿结合
今天给同学们分享一篇生信文章“Analysis and Experimental Validation of Rheumatoid Arthritis Innate Immunity Gene CYFIP2 and Pan-Cancer”,这篇文章发表在Front Immunol期刊上,影响因子为7.3。 结果解读: DEG筛选和数据预处理 数据在箱线图中进行了标准化,不同的颜色代表不同
卷王必看。非肿瘤+WGCNA+单细胞,快来抄作业
今天给同学们分享一篇生信文章“m6A Regulators Is Differently Expressed and Correlated With Immune Response of Esophageal Cancer”,这篇文章发表在Dis Markers期刊上,影响因子为3.464。 结果解读: 食道癌中差异表达基因(DEGs)的鉴定和功能注释 作者进行了整体分析,并绘制了箱
3+单细胞+代谢+WGCNA+机器学习
今天给同学们分享一篇生信文章“Identification of new co-diagnostic genes for sepsis and metabolic syndrome using single-cell data analysis and machine learning algorithms”,这篇文章发表Front Genet.期刊上,影响因子为3.7。 结果解读:
WGCNA如何挖掘潜在的共表达基因
欢迎关注微信公众号《生信修炼手册》! 共表达基因指的是表达量具有协同变化趋势的基因集合,通常认为这些基因参与相同的生物学过程,比如参与同一个代谢通路,正是由于功能上的协同作用,导致表达量呈现出高度相关性。 在WGCNA中,对传统的相关系数进行乘方运算,用最终得到的值来表征基因间的相关性。在计算出这样的相关性统计量值之后,如何确定哪些基因是共表达的呢? WGCNA的做法是聚类分析,聚类分析属于
7+共病思路。WGCNA+多机器学习+实验简单验证,易操作
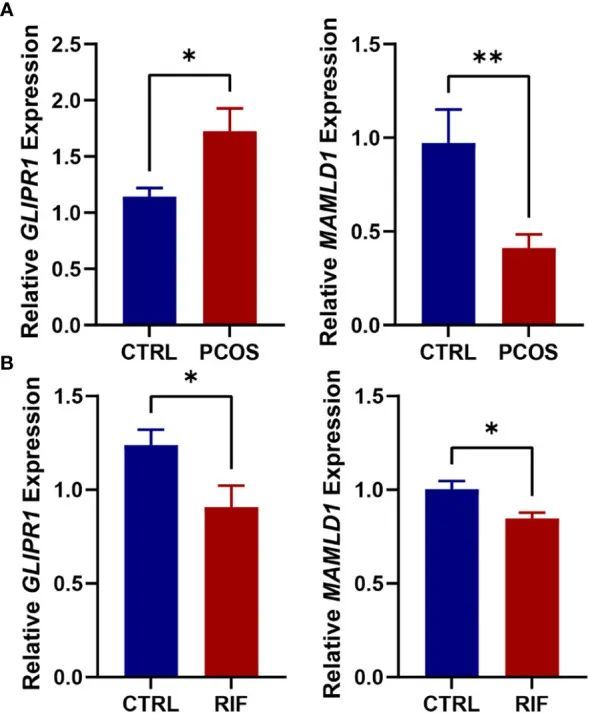
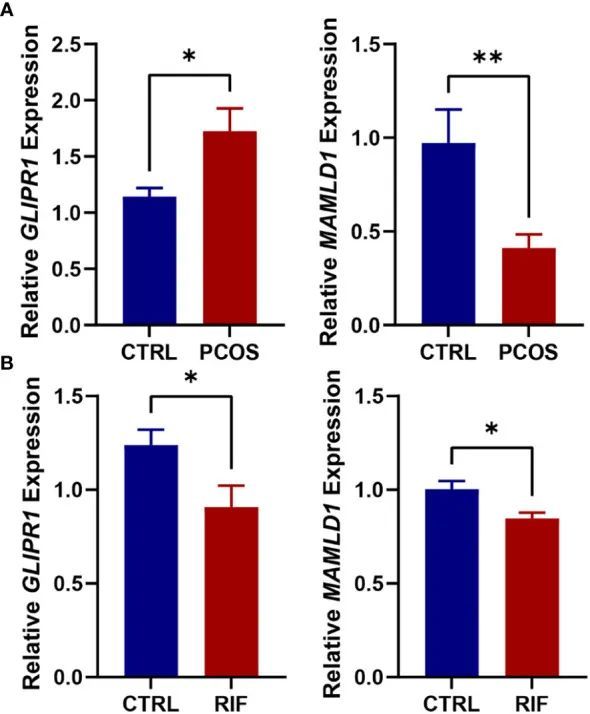
今天给同学们分享一篇共病+WGCNA+多机器学习+实验的生信文章“Shared diagnostic genes and potential mechanism between PCOS and recurrent implantation failure revealed by integrated transcriptomic analysis and machine learning”,
8+双疾病+WGCNA+多机器学习筛选疾病的共同靶点并验证表达
今天给同学们分享一篇双疾病+WGCNA+多机器学习的生信文章“Shared diagnostic genes and potential mechanism between PCOS and recurrent implantation failure revealed by integrated transcriptomic analysis and machine learning”,这篇
8+双疾病+WGCNA+多机器学习筛选疾病的共同靶点并验证表达
今天给同学们分享一篇双疾病+WGCNA+多机器学习的生信文章“Shared diagnostic genes and potential mechanism between PCOS and recurrent implantation failure revealed by integrated transcriptomic analysis and machine learning”,这篇
7+共病思路。WGCNA+多机器学习+实验简单验证,易操作
今天给同学们分享一篇共病+WGCNA+多机器学习+实验的生信文章“Shared diagnostic genes and potential mechanism between PCOS and recurrent implantation failure revealed by integrated transcriptomic analysis and machine learning”,
WGCNA分析教程五 | [更新版]
一边学习,一边总结,一边分享! 往期WGCNA分析教程 WGCNA分析 | 全流程分析代码 | 代码一 WGCNA分析 | 全流程分析代码 | 代码二 WGCNA分析 | 全流程分析代码 | 代码四 关于WGCNA分析教程日常更新 学习无处不在,我们的教程会在原有的基础上不断进行更新和添加新的内容,以及不断完善的分析代码。尽最大的能力,让大家拿到代码后直接上手分析。而不需要调整那些
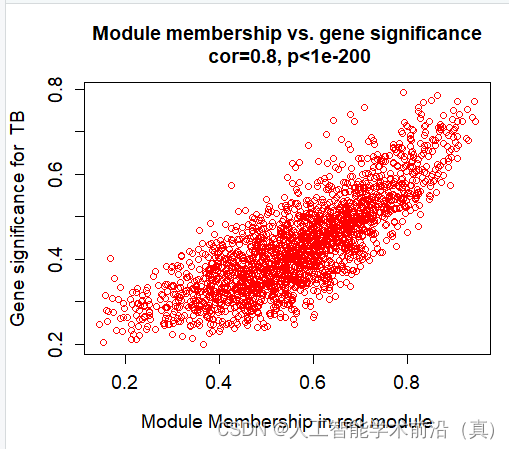
GEO生信数据挖掘(九)肺结核数据-差异分析-WGCNA分析(900行代码整理注释更新版本)
第六节,我们使用结核病基因数据,做了一个数据预处理的实操案例。例子中结核类型,包括结核,潜隐进展,对照和潜隐,四个类别。第七节延续上个数据,进行了差异分析。 第八节对差异基因进行富集分析。本节进行WGCNA分析。 WGCNA分析 分段代码(附运行效果图)请查看上节 运行后效果 rm(list = ls()) ######清除环境数据#========================
GEO生信数据挖掘(九)WGCNA分析
第六节,我们使用结核病基因数据,做了一个数据预处理的实操案例。例子中结核类型,包括结核,潜隐进展,对照和潜隐,四个类别。第七节延续上个数据,进行了差异分析。 第八节对差异基因进行富集分析。本节进行WGCNA分析。 目录 加载数据,进行聚类 初次聚类观察 自己定义红线位置,进行切割划分 载入性状数据 增加形状信息后,再次聚类 网络构建 选取soft-thresholding p
GEO生信数据挖掘(九)WGCNA分析
第六节,我们使用结核病基因数据,做了一个数据预处理的实操案例。例子中结核类型,包括结核,潜隐进展,对照和潜隐,四个类别。第七节延续上个数据,进行了差异分析。 第八节对差异基因进行富集分析。本节进行WGCNA分析。 目录 加载数据,进行聚类 初次聚类观察 自己定义红线位置,进行切割划分 载入性状数据 增加形状信息后,再次聚类 网络构建 选取soft-thresholding p
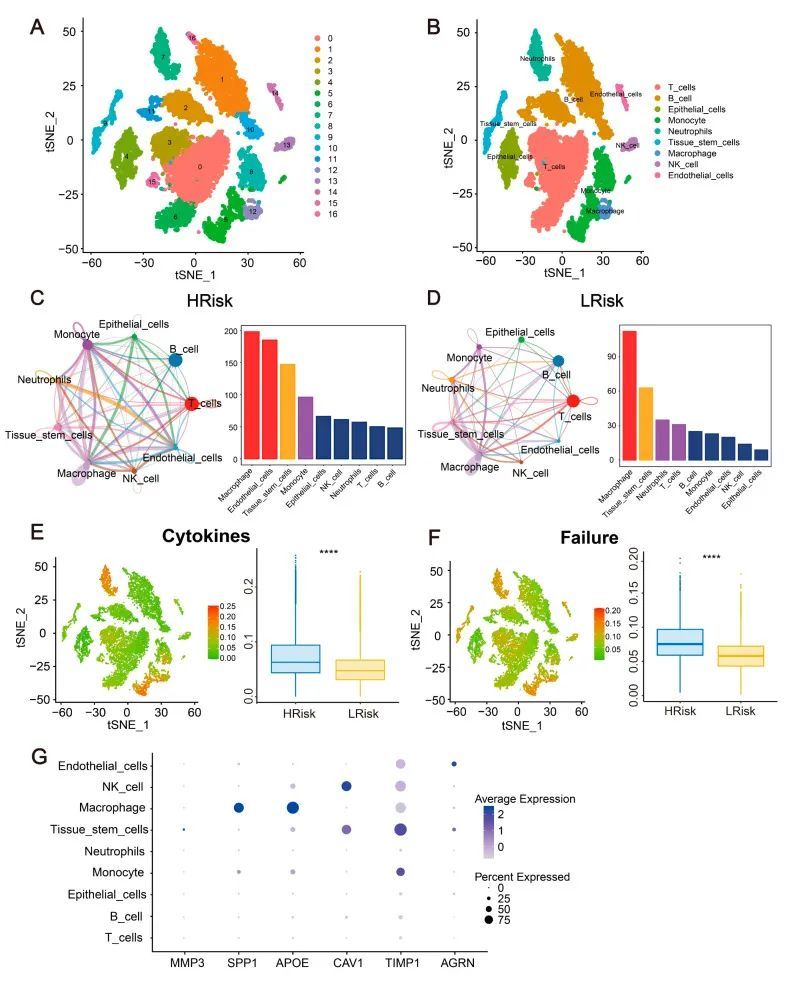
脂质代谢+预后模型+WGCNA+单细胞多种要素分析
今天给同学们分享一篇脂质代谢+预后模型+WGCNA+单细胞的生信文章“A Novel Lipid Metabolism and Endoplasmic Reticulum Stress-Related Risk Model for Predicting Immune Infiltration and Prognosis in Colorectal Cancer”,这篇文章于2023年9月8日发表在
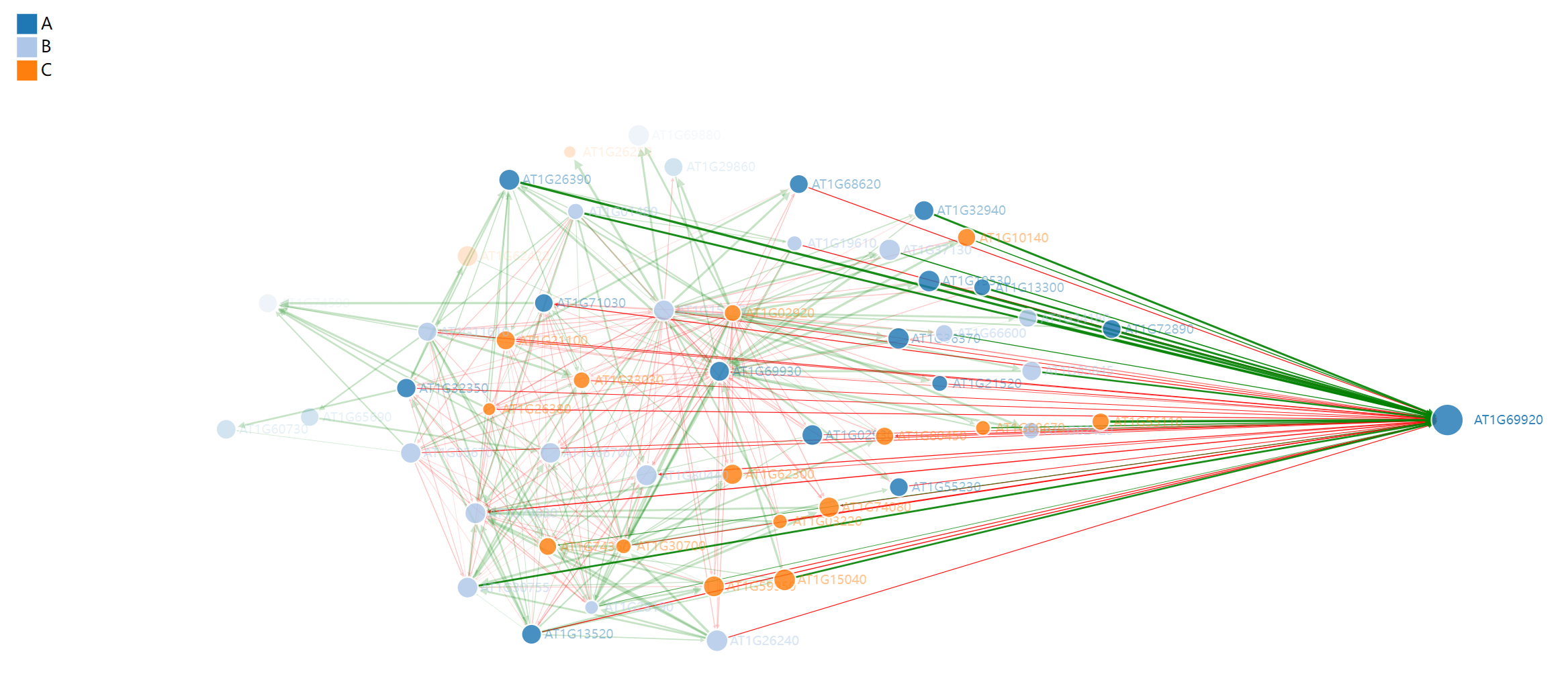
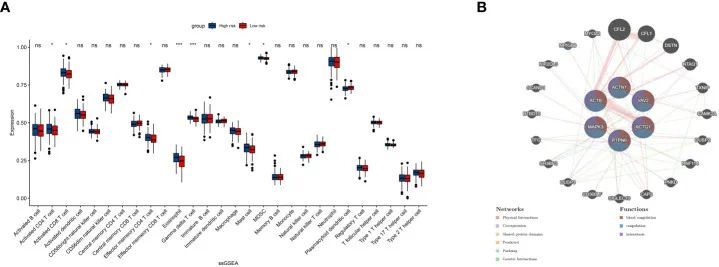
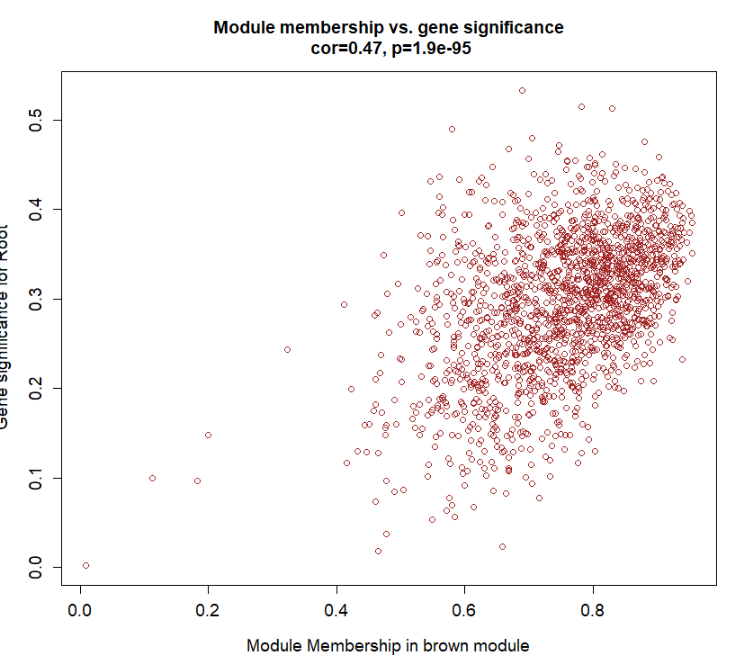
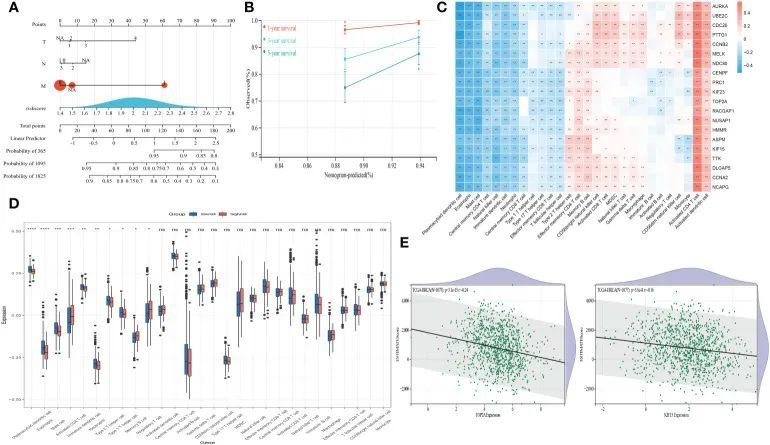
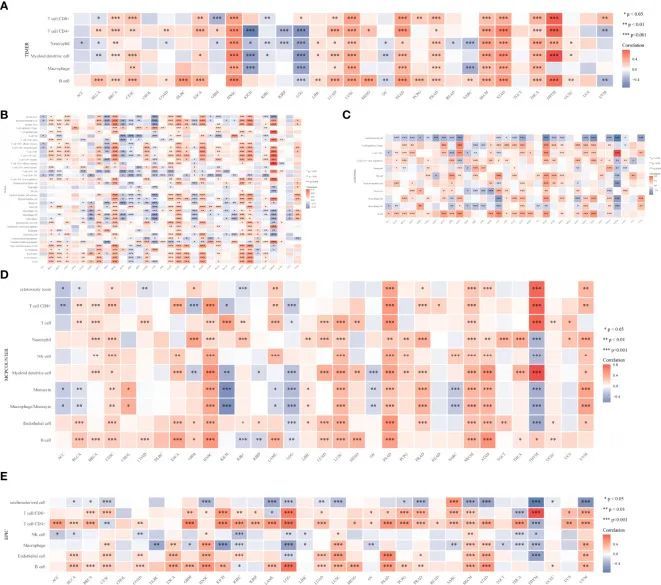
![WGCNA分析教程五 | [更新版]](https://img-blog.csdnimg.cn/img_convert/6b3ed167c11bfa4e4ccedc5e49452045.png)