本文主要是介绍脂质代谢+预后模型+WGCNA+单细胞多种要素分析,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
今天给同学们分享一篇脂质代谢+预后模型+WGCNA+单细胞的生信文章“A Novel Lipid Metabolism and Endoplasmic Reticulum Stress-Related Risk Model for Predicting Immune Infiltration and Prognosis in Colorectal Cancer”,这篇文章于2023年9月8日发表在Int Mol Sci期刊上,影响因子为5.6。
大肠癌(CRC)已成为第三大常见癌症和第二大癌症相关死亡原因[1]。不幸的是,约 20% 的 CRC 病例在确诊时已有转移[ 2]。由于手术方法、抗肿瘤药物和其他卓越治疗策略的进步,CRC 患者的临床治疗效果明显改善[3]。与传统化疗和靶向治疗相比,免疫疗法具有多种优势,包括持久的免疫反应、较少的副作用以及延长 DNA 错配修复缺陷或微卫星不稳定的转移性 CRC 患者的无进展生存期[4, 5]。然而,其中一些患者很快就会进入免疫抵抗阶段[6]。
1.探索 CRC 中差异表达的 LERGs
使用 "limma "R软件包鉴定差异基因,选择标准为p值< 0.05 and |FoldChange| > 1.5(|log 2 FoldChange| > 0.585)[ 25]。结果发现了 4940 个在 CRC 发展过程中表现出差异表达的关键基因,包括 2325 个上调基因和 2615 个下调基因(图 1A)。从 ER 应激和脂质代谢基因集中提取相关性得分大于 5 的基因,并与差异基因进行交叉,得到 271 个交叉基因(图 1B)。随后,Cox 比例-危害模型确定了 28 个对预后有意义的基因(p 值小于 0.05)(图 1C)。图1 基于差异表达的 LEGRs 构建预后特征
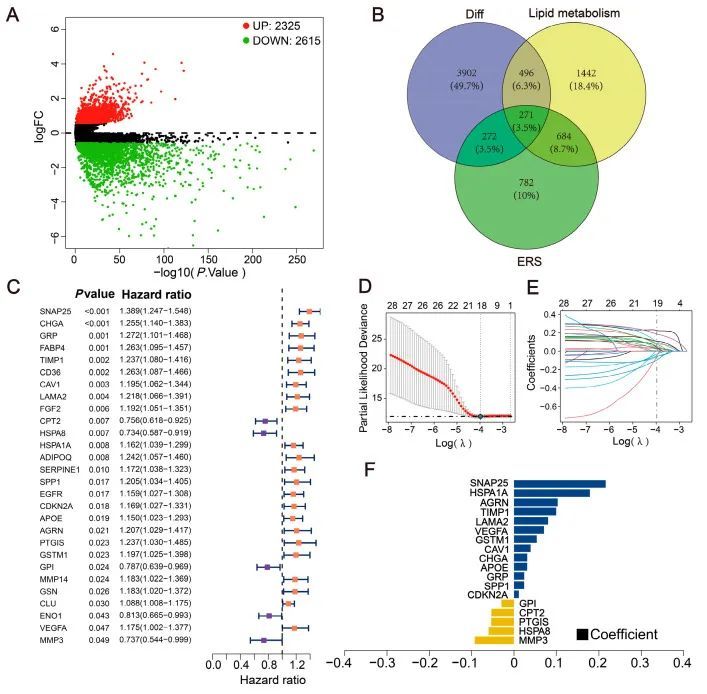
图1 基于差异表达的 LEGRs 构建预后特征
2.预后模型的构建
为了进一步确定预后基因集中的关键基因,作者收集了 CRC 患者的临床信息,并利用 lasso 回归筛选特征基因(图 1D-F)。TCGA 中的患者按 4:1 的比例随机分配到训练数据集和测试数据集。该风险模型用于预测肿瘤内的 ER 应激和脂质代谢水平。根据每个样本的中位风险评分,将患者分为高风险组和低风险组,并使用 Kaplan-Meier 曲线分析生存结果。在TCGA训练数据集(p < 0.001)和测试数据集(p = 0.016)中,高风险组的总生存期(OS)明显低于低风险组。此外,TCGA训练数据集和测试数据集的ROC曲线结果表明该模型具有良好的预测性能(图2A,B)。
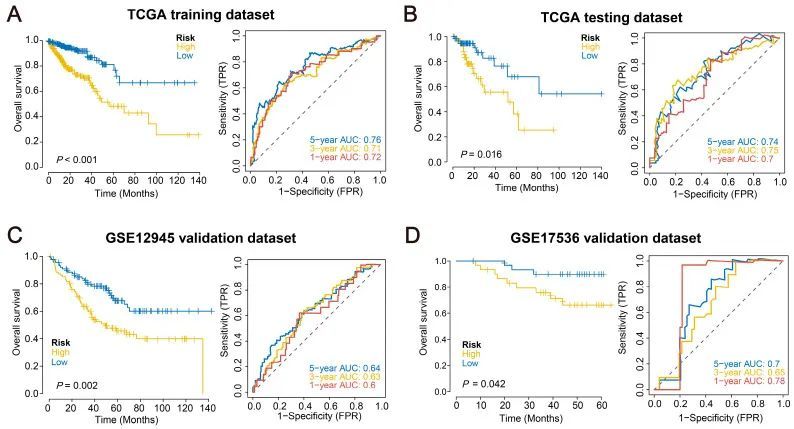
图2 评估风险模型预测患者生存率的效率
3.多个外部数据集对预后模型的验证
从 GEO 数据库中下载了 CRC 患者的生存信息数据(GSE12945、GSE17536)。根据中位风险评分,将验证数据集中的患者分为高风险组和低风险组进行生存分析。结果显示,在外部验证数据集GSE12945(p = 0.002)和GSE17536(p = 0.042)中,高风险组的生存率明显低于低风险组。为了验证模型的准确性,作者使用外部数据集对该模型进行了 ROC 曲线分析。结果表明,该模型对 CRC 患者的预后具有很强的预测性能和稳定性(图 2C,D)。
4.发病风险和独立预后分析
通过单变量分析(HR = 3.628 (2.687-4.898),p < 0.001)和多变量分析(HR = 3.006 (2.129-4.244),p < 0.001),发现风险评分是 CRC 患者的独立预后因素(图 3A、B)。结果以提名图的形式呈现(图 3C)。此外,还对 CRC 的 3 年期和 5 年期进行了预测分析,结果显示提名图具有很强的验证性能和预测准确性(图 3D-F)。
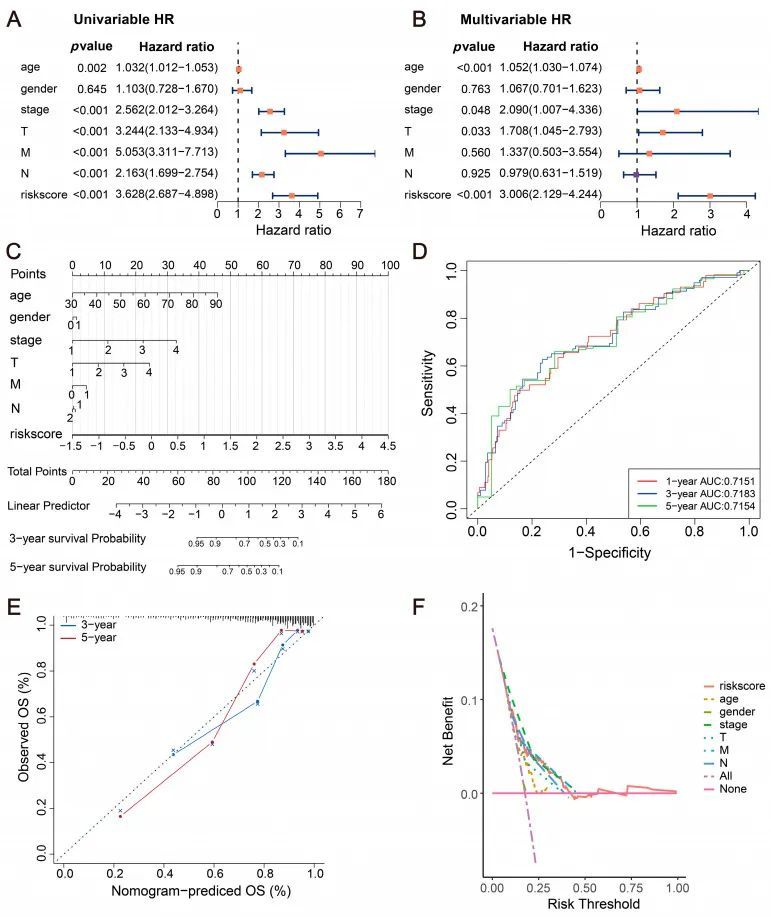
图3 构建评估患者生存期的预测提名图
5.疾病风险评分与多个临床指标的相关性分析
根据临床指标,将样本相关风险评分值分配到不同组别。经 Wilcoxon 检验发现,在临床指标(如分期、T、M 和 N)中,风险分值的分布显示出显著的组间差异(P < 0.05)(图 4)。这些结果表明,所构建的风险评分模型在显示 CRC 样本的分类方面具有良好的临床适用性。
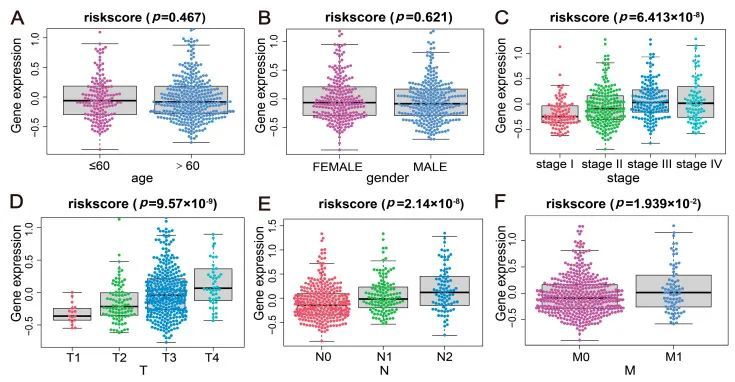
图4 风险评分与多个临床指标的相关性
6.CRC 的突变情况和肿瘤突变负荷 (TMB)
研究还进一步探讨了高风险组和低风险组患者的基因突变情况。研究结果表明,与低风险组相比,高风险组患者APC等基因的突变率明显较低(76% vs. 82%),而TP53和APC基因的突变率似乎高于低风险组(图5A,B)。随后进行了生存分析,将TMB和微卫星不稳定性(MSI)与风险评分相关联,结果见相应的图(图5C,D)。
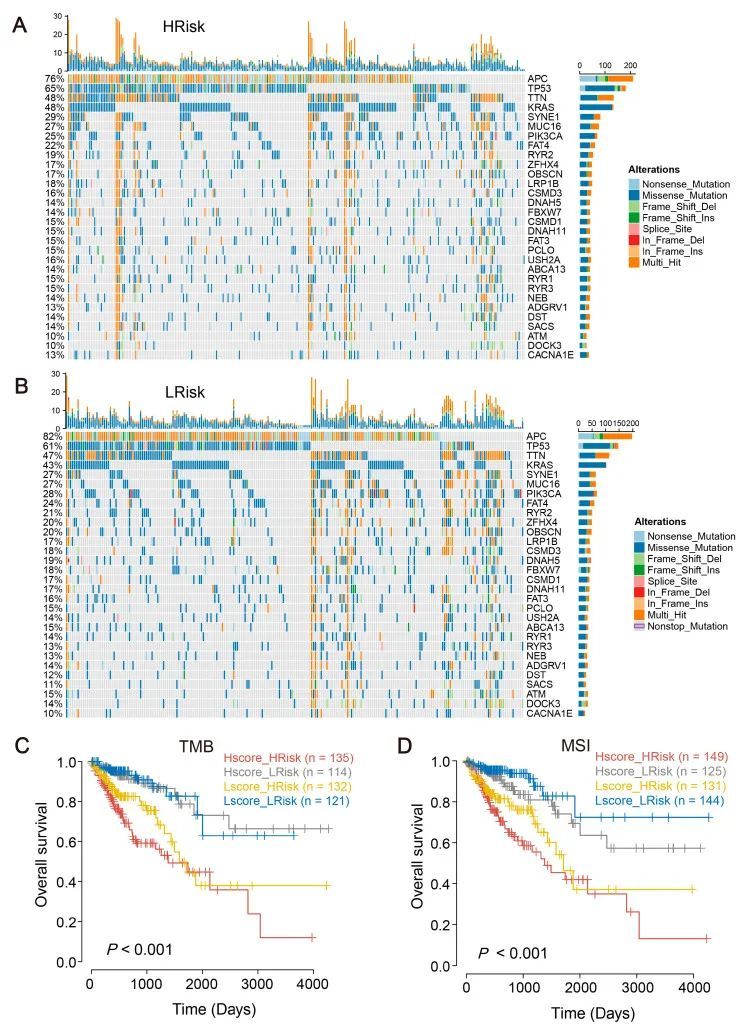
图5 图表格式相同
7.风险模型的药物敏感性和分子途径
手术治疗联合化疗已被证明对早期 CRC 有效。作者的研究结果表明,风险评分与对16种特定化疗药物的敏感性之间存在明显的相关性。低风险组患者对丝裂霉素 C、吉西他滨、AKT 抑制剂 VIII 和 Tipifarnib 的敏感性较高。另一方面,高风险组患者对多西他赛、吉非替尼、帕佐帕尼和雷帕霉素的敏感性更高(图 6A)。随后的分析侧重于特定的信号通路,以探索风险评分影响肿瘤进展的潜在分子机制。GSVA分析显示,两个患者队列之间的主要分歧通路主要存在于肌生成、表皮间质转换和髓鞘连接信号通路中(图6B)。GSEA分析显示,PI3K-Akt和Rap1信号通路在高风险组明显富集,而核苷酸代谢通路在低风险组富集(图6C)。
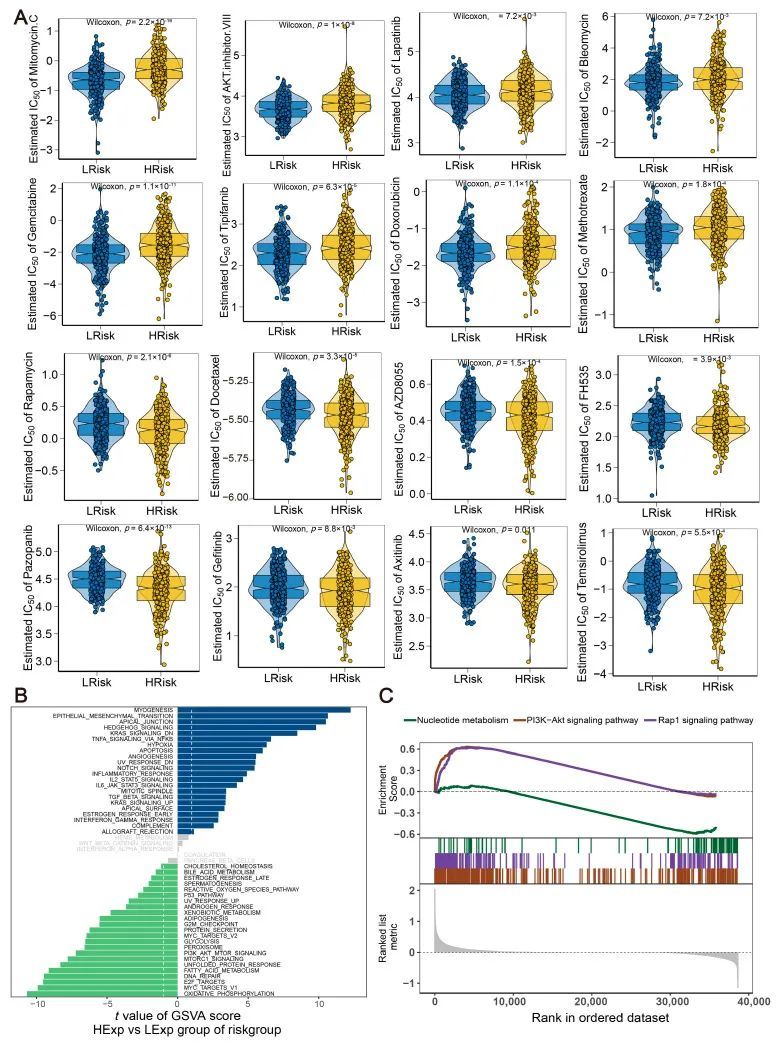
图6 风险模型的药物敏感性和分子途径
8.风险模型与免疫微环境之间的关系
TME主要由癌症相关成纤维细胞、免疫细胞、细胞外基质以及各种信号、分子和癌细胞组成,对肿瘤的诊断、预后和治疗敏感性有重要影响[27]。本研究进一步探讨了风险模型与 TME 中免疫浸润之间的关系。与不同风险组的免疫浸润相比,本研究发现高风险组的 T 细胞 CD4 + 记忆静息和 T 细胞 CD4 + 记忆激活明显减少。相反,天真 B 细胞、调节性 T 细胞(Tregs)和 M0 巨噬细胞则明显增加(图 7A)。风险评分与免疫细胞含量之间的相关性分析强调了与 M0 巨噬细胞和幼稚 B 细胞的显著正相关性,以及与静息和活化记忆 CD4 + T 细胞的负相关性(图 7B)。T 细胞呈负相关(图 7B)。此外,脂质代谢/ER 应激基因组与免疫浸润之间的相关性也通过 ssGSEA 得到了说明。肿瘤样本中的 M2 巨噬细胞和幼稚 B 细胞与脂质代谢/ER 应激水平呈正相关。相反,NK 细胞和 CD8 + T 细胞则显示出负相关。T细胞呈负相关(图7D)。这表明,在脂质代谢/ER应激水平升高的情况下,TME主要表现出免疫抑制表型,这与作者的风险模型的免疫浸润分析一致。作者对免疫调节基因进行了进一步分析,结果显示了高风险组和低风险组之间免疫相关抑制因子和细胞因子的表达差异(图 7E,F)。最后,对肿瘤免疫疗法的敏感性进行了预测,结果显示高风险组对免疫疗法的反应较差(图 7C)。
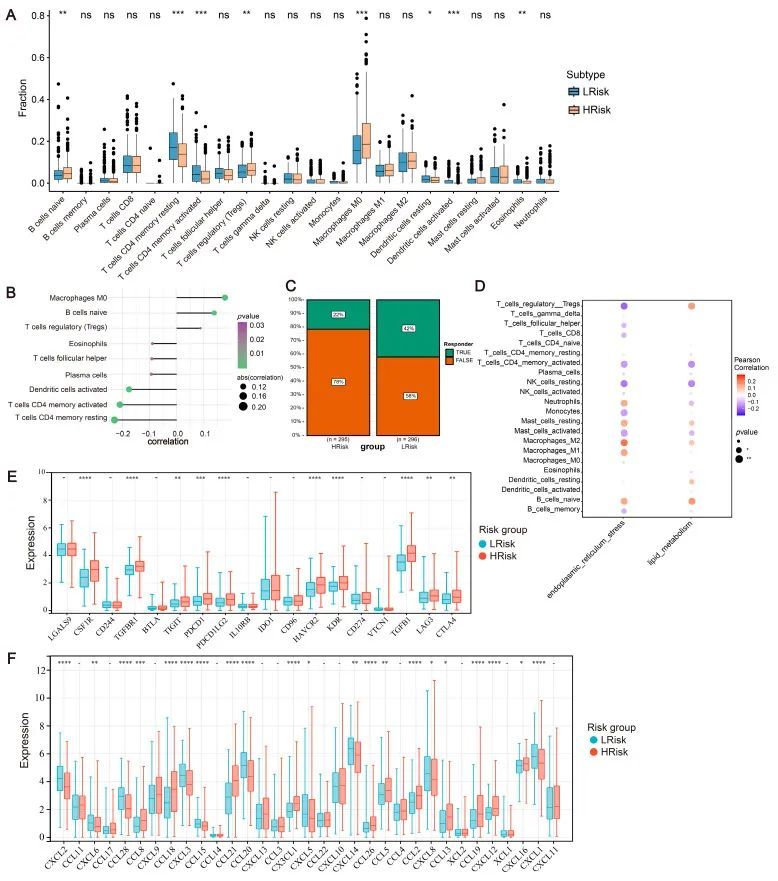
图7 低风险组和高风险组之间不同的免疫景观
9.构建 WGCNA 共表达网络并分析关键基因的上游机制
为了进一步确定与结直肠癌相关的关键基因,作者根据结直肠癌转录组采用了 WGCNA 来阐明相关的共表达调控网络[28]。软阈值 β 设为 6(图 8A,B),通过 TOM 共鉴定出 8 个基因模块(图 8C,D)。这些模块包括黑色(99)、蓝色(1052)、棕色(610)、绿色(454)、灰色(569)、红色(176)、绿松石色(1572)和黄色(468)模块。棕色模块的相关性最高(cor = 0.49,p = (5 × 10 −37 )。将棕色模块中的基因与 18 个模型基因交叉后,确定了 6 个基因作为进一步研究的关键候选基因(图 8E),即 MMP3、SPP1、APOE、CAV1、TIMP1 和 AGRN。随后的 PPI 网络分析显示了这些关键基因之间的相互作用(图 8F)。还对关键基因的上游转录因子(TF)进行了分析。结果表明,MOTIF注释 cisbp_M5419 的 NES 为 4.9,三个关键基因富集在这一主题中。图 8G 中显示了与关键基因相关的前六个主题及其相应的 TF。
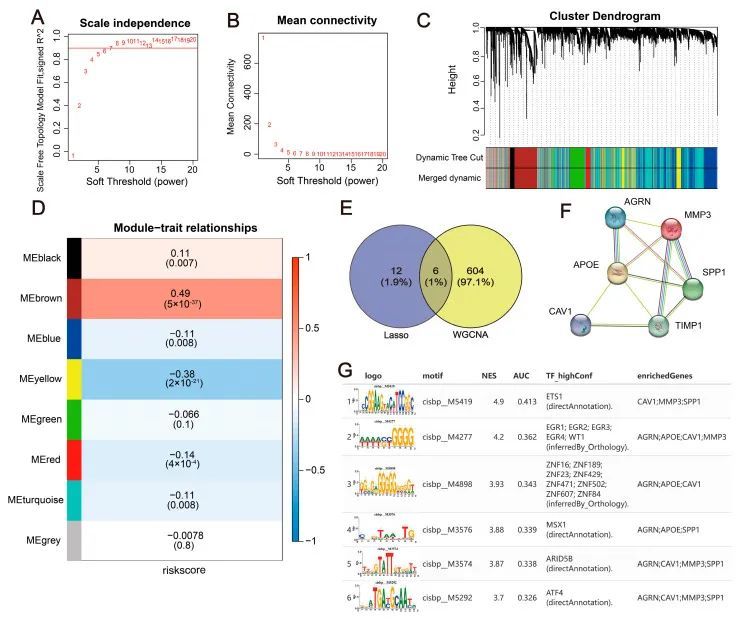
图8 WGCNA 共表达网络的构建及关键基因的上游机制分析
10.在 CRC 临床样本中验证关键基因
最后,作者利用定量实时聚合酶链反应qRT-PCR和HPA数据库中的病理切片图像对12对临床样本的基因表达进行了探讨(图9A-E)。在这六个中心基因中,与正常组织相比,AGRN、SPP1、MMP3 和 TIMP1 在 CRC 组织中的表达明显升高。此外,CAV1 在 RNA 和蛋白水平上的表达都明显下降。虽然 APOE 在 qRT-PCR 中未显示出明显变化,但蛋白水平却显示出下降趋势,这与 TCGA 分析结果一致。
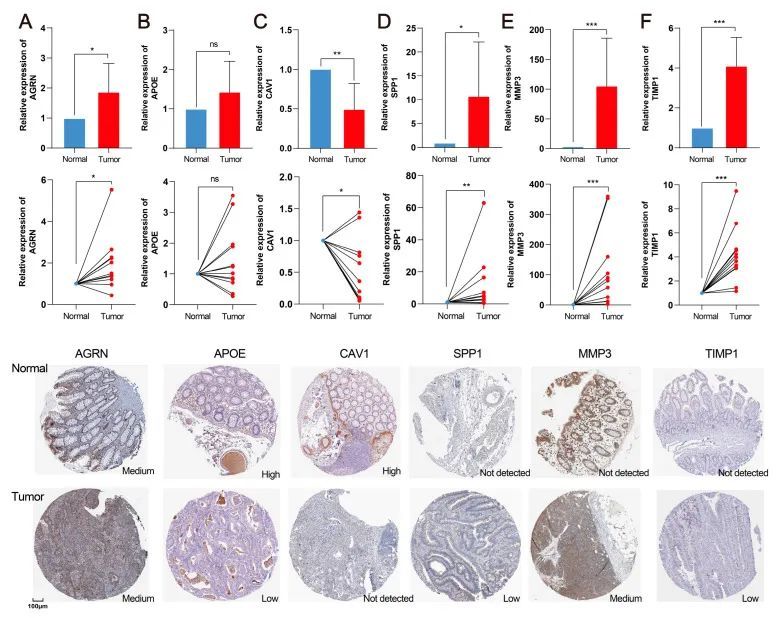
图9 验证 CRC 临床样本中的关键基因
11.CRC 免疫格局和基因表达的单细胞分析
使用 t-SNE 算法对细胞进行聚类,最终确定了 17 个亚型(图 10A)。利用 R 软件包 "SingleR "对每个亚型进行注释,17 个聚类属于 9 个细胞类别:T细胞、B细胞、上皮细胞、单核细胞、中性粒细胞、组织干细胞、巨噬细胞、NK细胞和内皮细胞(图10B)。六种中心基因在这九种细胞类型中的表达情况直观可见(图 10G)。研究发现,在高风险组和低风险组中,这些细胞亚型之间存在着错综复杂的相互作用模式。研究发现,在两个风险组中,巨噬细胞与其他细胞的潜在相互作用最多(图 10C、D)。随后,利用 GeneCards 数据库获得了失败评分和细胞因子评分,并对高风险组和低风险组进行了比较。分析结果如图所示(图 10E、F)。
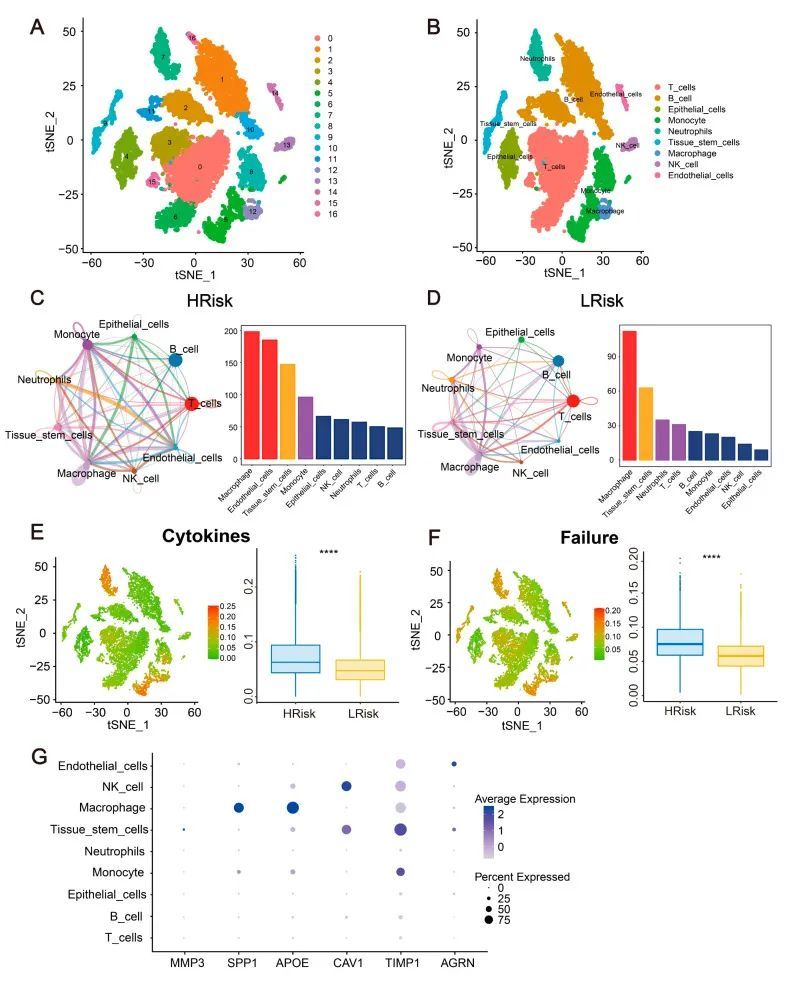
图10 免疫图谱的单细胞分析
总结
总之,作者发现了 18 个与脂质代谢和 ER 应激相关的差异表达基因,并利用这些基因构建了一个风险模型。作者的风险模型与 CRC 患者的总生存期、抗肿瘤治疗效果和免疫浸润相关。这揭示了ER应激和脂质代谢水平的升高与CRC的免疫抑制状况和不良预后相关,为精准治疗提供了战略性启示。通过探索脂质代谢和ER应激的新方向,这项研究可以帮助制定个性化治疗策略,并为进一步研究CRC的免疫微环境奠定重要基础。
这篇关于脂质代谢+预后模型+WGCNA+单细胞多种要素分析的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!








