本文主要是介绍7+非肿瘤+WGCNA+机器学习+诊断模型,构思巧妙且操作简单,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
今天给同学们分享一篇生信文章“Platelets-related signature based diagnostic model in rheumatoid arthritis using WGCNA and machine learning”,这篇文章发表在Front Immunol期刊上,影响因子为7.3。

结果解读:
DEGs和血小板相关基因的鉴定
作者通过R的“GEOquery”和“limma”包,在数据预处理后获得了GSE93272的基因表达矩阵和临床数据。DEG的选择标准为log2|FC|≥1和adj.P≤0.05。(FC,倍数变化;adj.P:调整后的P值)。作者获得了3776个上调的DEG和4714个下调的DEG(图1A)。每个样品中DEG的表达如图1B所示。
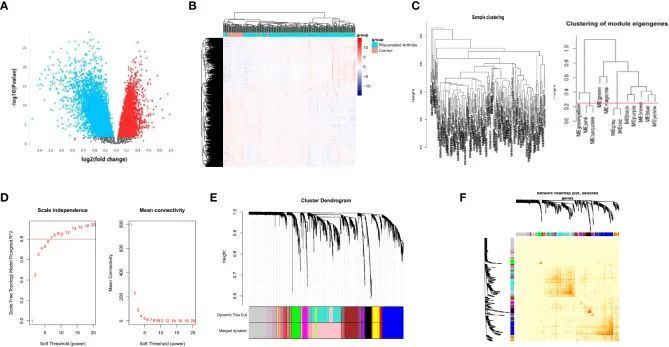
通过WGCNA识别PRS
作者使用样本聚类树来阐明异常值(图1C)。然后,作者通过WGCNA中的“pickSoftThreshold”函数选择软阈值β(图1D),并识别模块(图1C)。软阈值设置为7。作者进一步开发了一个层次聚类树,每个分支代表具有相似表达和生物功能的基因(图1E)。此外,作者通过计算连通度分析了已阐明模块之间的相互作用(图1F)。
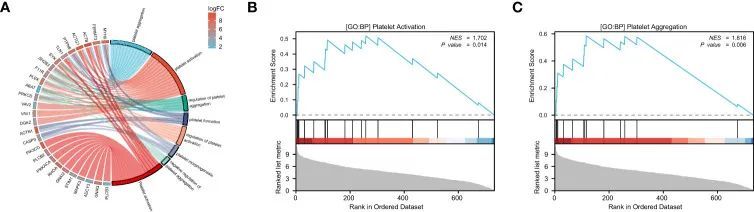
模块的富集度分析
作者使用R中的“clusterProfiler”包进行了GO和KEGG分析,以确定与血小板关系最密切的模块,即模块2。随后,作者对模块2进行了GO和KEGG分析,以确定血小板相关途径(图2A)。图2A所示的参与该途径的基因被鉴定为潜在的PRS候选基因。为了更准确地确定血小板相关途径的活性,作者采用了GSEA。具体而言,作者评估了模块2的GOBP血小板活化(图2B)和GOBP血小板聚集(图2C)的活性。
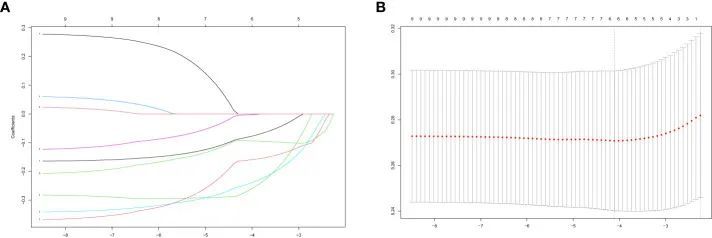
PRS模型的构建
作者从作为训练组的GSE93272中获得候选PRS的表达数据。然后,作者利用LASSO算法导出系数剖面图(图3A)和部分似然偏差(图3B)。从这些分析中,作者确定了六个非零系数特征,即MAPK3、ACTB、ACTG1、VAV2、PTPN6和ACTN1,用于构建风险评分模型。
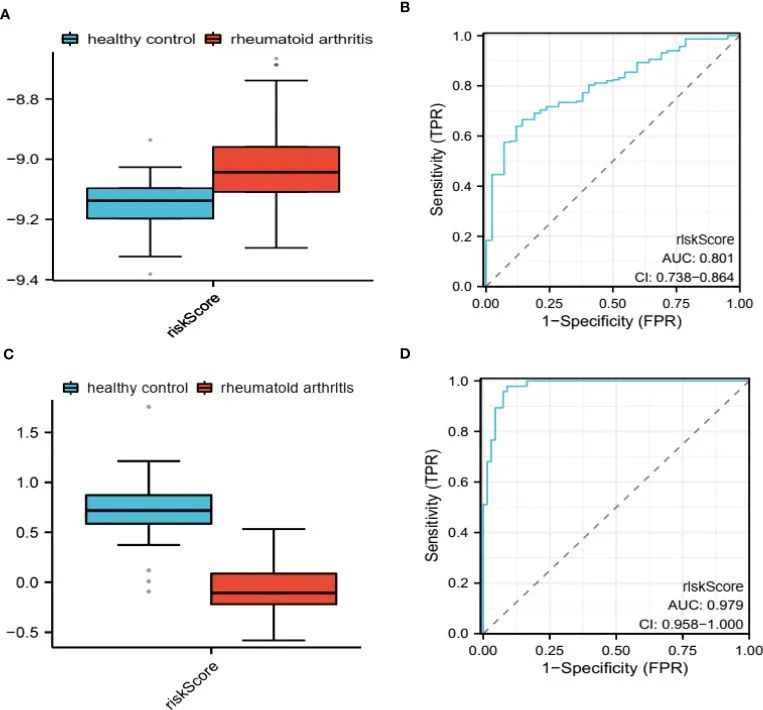
训练和验证队列中诊断潜力的评估
作者通过计算这些队列中每个样本的风险得分,评估了作者的PRS在训练组和验证组中的预测能力(图4A、C)。随后,作者采用ROC分析来确定作者的模型的诊断潜力。训练组和验证组的AUC值分别为0.801(图4B)和0.979(图4D),表明两个队列的诊断准确性都很高。作者还通过R的“modEVA”应用了精度-召回曲线,以进一步评估其效率。为了区分高风险组和低风险组,作者在训练组中使用了9.08的临界值。
免疫相关状态和基因分析
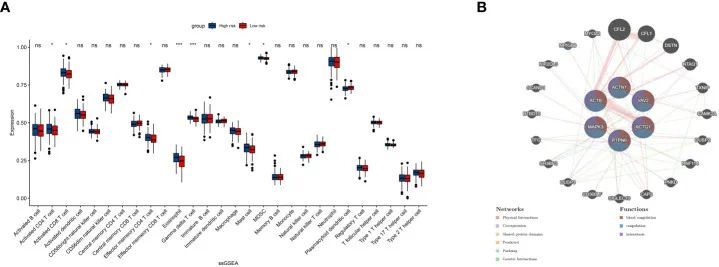
为了进一步检查与PRS相关的免疫状态,作者使用单样本基因集富集分析(ssGSEA)来分析免疫景观(图5A)。作者的研究结果表明,活化的CD4/8 T细胞、效应记忆CD4 T细胞、嗜酸性粒细胞、γ-ΔT细胞、肥大细胞、髓源性抑制细胞(MDSCs)和浆细胞样树突状细胞在高危组和低危组之间存在显著差异。此外,作者使用GeneMANIA来确定与PRS最密切相关的前20个基因(图5B)。此外,作者检测了PRS的共表达网络,发现其与凝血、凝血和止血有很强的相关性。
总结
总之,虽然作者的研究突出了PRS作为RA诊断工具的潜力,但还需要进一步研究来解决上述限制,并全面评估作者的方法在RA诊断和管理中的临床实用性。
这篇关于7+非肿瘤+WGCNA+机器学习+诊断模型,构思巧妙且操作简单的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!




