本文主要是介绍2D人体姿态识别-对Human3.6M数据集预处理(1):用python读取并处理cdf文件,cdflib包中各函数介绍,Human3.6M数据集2d关节点格式解读,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
文章目录
- 一、Human3.6M数据集结构介绍
- 二、处理cdf.文件
- 1.法一:使用nasa官方提供的CDF library
- 2.法二:使用CDF library的纯python替代品:cdflib
- 1)安装cdflib
- 2)cdflib中各函数介绍
- ①读取一个cdf.文件 cdflib.CDF('xxx.cdf')
- ②查看此cdf.文件信息: cdf_info()
- ③提取此cdf文件中的变量:varget ()
- 3)*cdflib实例:处理H3.6M中存储2d keypoints的cdf文件
- 4)解读Human3.6M数据集中视频和2d关节点数据
前言
一、Human3.6M数据集结构介绍
Human3.6M数据集有360万个3D人体姿势和相应的图像,共有11个实验者(Subject)(6男5女,论文一般选取1,5,6,7,8作为train,9,11作为test),和17个动作场景(Scenario),诸如讨论、吃饭、运动、问候等动作。该数据由4个数字摄像机,1个时间传感器,10个运动摄像机捕获。
我需要训练的网络输入应该为两个角度的人物照片(从视频中提取关键帧),2d关节的关键点作为监督(从cdf文件中提取)。从官网的以下分类中下载:

大批量预处理Human3.6M数据集的方法可以参考:github: h36m-fetch
这次的小批量预处理先以S1(人物)的sitting1.55011271(人物右前方,观察者的左侧,下面用左来命名)和sitting.60457274(人物左前方,观察者的右侧,下面用右来命名)为例。
二、处理cdf.文件
1.法一:使用nasa官方提供的CDF library
参考:使用python读取cdf数据
- 第一步:下载CDF Library:nasa cdf 官网
- 第二步:编译
make OS=linux ENV=gnu CURSES=yes FORTRAN=no UCOPTIONS=-O2 SHARED=yes all - 第三步:安装
sudo make INSTALLDIR=/usr/local/cdf install - 第四步:安装
spacepy pip install spacepy - 第五步:使用代码如下实例
import os
from spacepy import pycdf
os.environ["CDF_LIB"] = "/usr/local/cdf/lib"
anno_Greeting=pycdf.CDF('/home/maddaff/Documents/TestSpace/Greeting.54138969.cdf')
data=anno_Greeting.copy()
anno_Greeting.close()
print data.keys()
NASA官网提供的可下载文件太多了,我不知道下载哪个才是正确的CDF Lib,故而放弃了这种方法。
2.法二:使用CDF library的纯python替代品:cdflib
cdflib模块详情(github地址)
1)安装cdflib
在cmd中执行pip install cdflib
或者直接在Pycharm中安装此package

2)cdflib中各函数介绍
①读取一个cdf.文件 cdflib.CDF(‘xxx.cdf’)
import cdflib
cdf_file = cdflib.CDF('/path/to/cdf_file.cdf')
②查看此cdf.文件信息: cdf_info()
返回一个显示基本CDF信息的字典。这些信息包括:
cdfCDF的名称
version CDF的版本
encoding CDF的字节顺序
Majority 行/列多数
zVariables 一个zVariables名称的列表。
rVariables 一个rVariables名称的列表。
Attributes 一个包含属性名及其作用域的字典对象列表,例如 - {attribute_name : scope}。
Checksum 验算符
Num_rdim 维度数,仅适用于 rVariables。
rDim_sizes 维度大小,仅适用于 rVariables。
Compressed CDF压缩到此文件级
LeapSecondUpdated 最后更新的闰年表(如适用)
③提取此cdf文件中的变量:varget ()
varget (variable = None, [epoch=None], [[starttime=None, endtime=None] | [startrec=0, endrec = None]], [,expand=True])
- 默认情况下,将返回完整的变量数据。要仅获取记录可变变量的一部分数据,可以指定时间或记录(从0开始)范围。
epoch可用于指定该变量所依赖的时间变量,并将在时间范围内进行搜索。对于符合ISTP的CDF,时间变量将来自此变量的属性“ DEPEND_0”。该功能将自动搜索它,因此无需指定“epoch”。- 如果未指定开始时间或结束时间(
starttime和end time),则假定特定时期数据类型可能的最小值或最大值。 - 如果未指定开始或结束记录(
startrec和endrec),则范围从0开始,结束于最后写入的数据。
开始(和结束)时间应在列表中显示为:
- [year month day hour minute second millisec] for CDF_EPOCH
- [year month day hour minute second millisec microsec nanosec picosec] for
CDF_EPOCH16 - [year month day hour minute second millisec microsec nanosec] for CDF_TIME_TT2000
如果没有提供足够的时间分量,则只有最后一项可以具有子时间分量的浮动部分。
注意:CDF的CDF_EPOCH16数据类型为每个数据值使用2个8字节双精度数。在Python中,每个值都表示为complex或numpy.complex128。
返回变量数据。可以输入变量名或变量号。默认情况下,它会根据数据类型返回带有变量data及其规范的numpy.ndarray or或list()class对象。
如果为expand=True,则返回具有以下定义的键的字典用于输出
Rec_Ndim 每个变量记录的维数
Rec_Shape 变量的形状
Num_Records 记录总数
Records_Returned 检索到的记录数
Data_Type CDF数据类型
Data 检索变量数据
Real_Records 列表中稀疏记录变量的真实数据记录号
例如:
x = cdf_file.varget("NameOfVariable", startrec = 0, endrec = 150)
此命令将返回变量Variable10中的所有数据,从记录时刻0到150
3)*cdflib实例:处理H3.6M中存储2d keypoints的cdf文件
import cdflib#load a cdf file
cdf= cdflib.CDF('Sitting 1.55011271.cdf')#View the Information about the cdf file
info = cdf.cdf_info()#Get the variables in the cdf file
x = cdf.varget("Pose")
4)解读Human3.6M数据集中视频和2d关节点数据
上述代码生成的变量如下图所示:

我们重点关注x(1,3304,64),也就是从cdf文件中提取出来的变量,表示的是视频每一帧中各关节点的2d坐标标注。
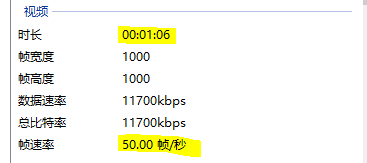
- 行-帧数:cdf文件的每一行的定义是一帧中的标注,因此一个标注文件对应的视频有多少帧,这个文件就有多少行。这个sitting1.55011271.mp4共有3304帧。
- 列-32个关键点的2D坐标:这3304行的每一行里面有64个数据,每2个数据为一组,共32组,每组表示的便是这个关键点的2D坐标。
论文《Human3.6M: Large Scale Datasets and Predictive Methods for 3D Human Sensing in Natural Environments》提到,数据集中共有32个关节点,但并未给出具体的标注顺序和对应关节。
在h3.6m中,会有一些看似冗余标注点,如图中的11和0是重合的,其实是因为h36m的节点来说,如果它们的父节点相同,那么它们共享同一个旋转向量,因此所有父节点相同的节点相对父节点旋转角度都是一样的,这样会造成一些问题,比如节点0有3子节点,但是胯部的方向和脊椎的旋转方向是不一样的,因此就需要一个多设置一个11节点作为12的父节点来改变12节点的旋转方向。同理,20与19重合用来改变21的方向,28与27重合用来改变29的方向,16和24都与13重合,16用来改变17的方向,24用来改变25的方向,剩下的23和31则为无意义节点。
————————————————
版权声明:本文为CSDN博主「alickr」的原创文章,遵循CC 4.0 BY-SA版权协议,转载请附上原文出处链接及本声明。
原文链接:https://blog.csdn.net/alickr/article/details/107837403
由此可知,H36M数据集中标注32个关键点(见右图),一部分是为了满足表达旋转向量的需要的。我们只研究关节点的2d位置时,不需要考虑全部的32个关键点,只需要提取出有效表示对应关节的关键点即可(见左表)。

右图是参考博文CSDN:human3.6m数据集格式解析中给出的全部关键点(keypoints)的标注顺序,左表是博文COCO和Human3.6m两个数据集的人体骨骼标注顺序 中给出的关键点和主要关节对应关系(注意这篇博文里coco的标注是错的!正确的关系见Coco key point json file parsing):
这篇关于2D人体姿态识别-对Human3.6M数据集预处理(1):用python读取并处理cdf文件,cdflib包中各函数介绍,Human3.6M数据集2d关节点格式解读的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!



