本文主要是介绍biostar handbook(七)| BLAST,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
Basic local alignment search tool (BLAST)
包括:blastn, blastp, blastx, tblastn, tblastx等. 使用conda安装即可。
conda install -c bioconda blast
# blast安装perl模块的方法
conda isntall perl-digest-md5
BLAST的主要理念
- Search may take place in nucleotide and/or protein space or translated spaces where nucleotides are translated into proteins.
- Searches may implement search “strategies”: optimizations to a certain task. Different search strategies will return different alignments.
- Searches use alignments that rely on scoring matrices
- Searches may be customized with many additional parameters. BLAST has many subtle functions that most users never need.
本地BLAST的基本步骤
- 用makeblastdb为BLAST提供数据库
- 选择blast工具,blastn,blastp
- 运行工具,有必要的还可以对输出结果进行修饰
第一步:建立检索所需数据库
BLAST数据库分为两类,核酸数据库和氨基酸数据库,可以用makeblastbd创建。可以用help参数简单看下说明。
$ makeblastdb -help
USAGEmakeblastdb [-h] [-help] [-in input_file] [-input_type type]-dbtype molecule_type [-title database_title] [-parse_seqids][-hash_index] [-mask_data mask_data_files] [-mask_id mask_algo_ids][-mask_desc mask_algo_descriptions] [-gi_mask][-gi_mask_name gi_based_mask_names] [-out database_name][-max_file_sz number_of_bytes] [-logfile File_Name] [-taxid TaxID][-taxid_map TaxIDMapFile] [-version]
-dbtype <String, `nucl', `prot'>
具体以拟南芥基因组作为案例,介绍使用方法:
注: 拟南芥的基因组可以在TAIR上下在,也可在ensemblgenomes下载。后者还可以下载其他植物的基因组
# 下载基因组
wget ftp://ftp.ensemblgenomes.org/pub/plants/release-36/fasta/arabidopsis_thaliana/dna/Arabidopsis_thaliana.TAIR10.dna.toplevel.fa.gz
gzip -d Arabidopsis_thaliana.TAIR10.dna.toplevel.fa.gz
# 构建核酸BLAST数据库
makeblastdb -in Arabidopsis_thaliana.TAIR10.dna.toplevel.fa -dbtype nucl -out TAIR10 -parse_seqids# 下载拟南芥protein数据
wget ftp://ftp.ensemblgenomes.org/pub/plants/release-36/fasta/arabidopsis_thaliana/pep/Arabidopsis_thaliana.TAIR10.pep.all.fa.gz
# 构建蛋白BLAST数据库
gzip -dArabidopsis_thaliana.TAIR10.pep.all.fa.gz
makeblastdb -in Arabidopsis_thaliana.TAIR10.pep.all.fa -dbtype prot -out TAIR10 -parse_seqids
如果你从NCBI或者其他渠道下载了格式化过的数据库,那么可以用blastdbcmd去检索blast数据库,参数很多,常用就如下几个
- db string : string表示数据库所在路径
- dbtype string,: string在(guess, nucl, prot)中选择一个
- 检索相关参数
- -entry all 或 555, AC147927 或 gnl|dbname|tag'
- -entry_batch 提供一个包含多个检索关键字的文件
- -info 数据库基本信息
- 输出格式 -outfmt %f %s %a %g ...默认是%f
- out 输出文件
- show_blastdb_search_path: blast检索数据库路径
使用案例
# 查看信息
blastdbcmd -db TAIR10 -dbtype nucl -info
# 所有数据
blastdbcmd -db TAIR10 -dbtype nucl -entry all | head
# 具体关键字,如GI号
blastdbcmd -db TAIR10 -dbtype nucl -entry 3 | head
还有其他有意思的参数,可以看帮助文件了解
可选:BLAST安装和更新nr和nt库
安装nt/nr库需要先进行环境变量配置,在家目录下新建一个.ncbirc配置文件,然后添加如下内容
; 开始配置BLAST
[BLAST]
; 声明BLAST数据库安装位置
BLASTDB=/home/xzg/Database/blast
; Specifies the data sources to use for automatic resolution
; for sequence identifiers
DATA_LOADERS=blastdb
; 蛋白序列数据库存本地位置
BLASTDB_PROT_DATA_LOADER=/home/xzg/Database/blast/nr
; 核酸数据库本地存放位置
BLASTDB_NUCL_DATA_LOADER=/home/xzg/Database/blast/nt
[WINDOW_MASKER]
WINDOW_MASKER_PATH=/home/xzg/Database/blast/windowmasker
配置好之后,使用BLAST+自带的update_blastdb.pl脚本下载nr和nt等库文件(不建议下载序列文件,一是因为后者文件更大,二是因为可以从库文件中提取序列blastdbcmd -db nr -dbtype prot -entry all -outfmt "%f" -out nr.fa ,最主要是建库需要花费很长时间),直接运行下列命令即可自动下载。
nohup time update_blastdb.pl nt nr > log &
如果你不像通过update_blastdb.pl下载nr和nt等库文件,也可以是从ncbi上直接下载一系列nt/nr.xx.tar.gz文件,然后解压缩即可,后续还可以用update_blastdb.pl进行数据更新。
报错: 使用update_blastdb.pl更新和下载数据库时候出现模块未安装的问题。解决方法,首先用conda安装对应的模块,然后修改update_blastdb.pl的第一行,即shebang部分,以conda的perl替换,或者按照如下方法执行。
perl `which update_blastdb.pl`
下载过程中请确保网络状态良好,否则会出现Downloading nt.00.tar.gz...Unable to close datastream报错。
第二步:选择blast工具
根据不同的需求,比如说你用的序列是氨基酸还是核苷酸,你要查找的数据是核甘酸还是氨基酸,选择合适的blast工具。不同需求的对应关系可以见下图(来自biostars handbook)
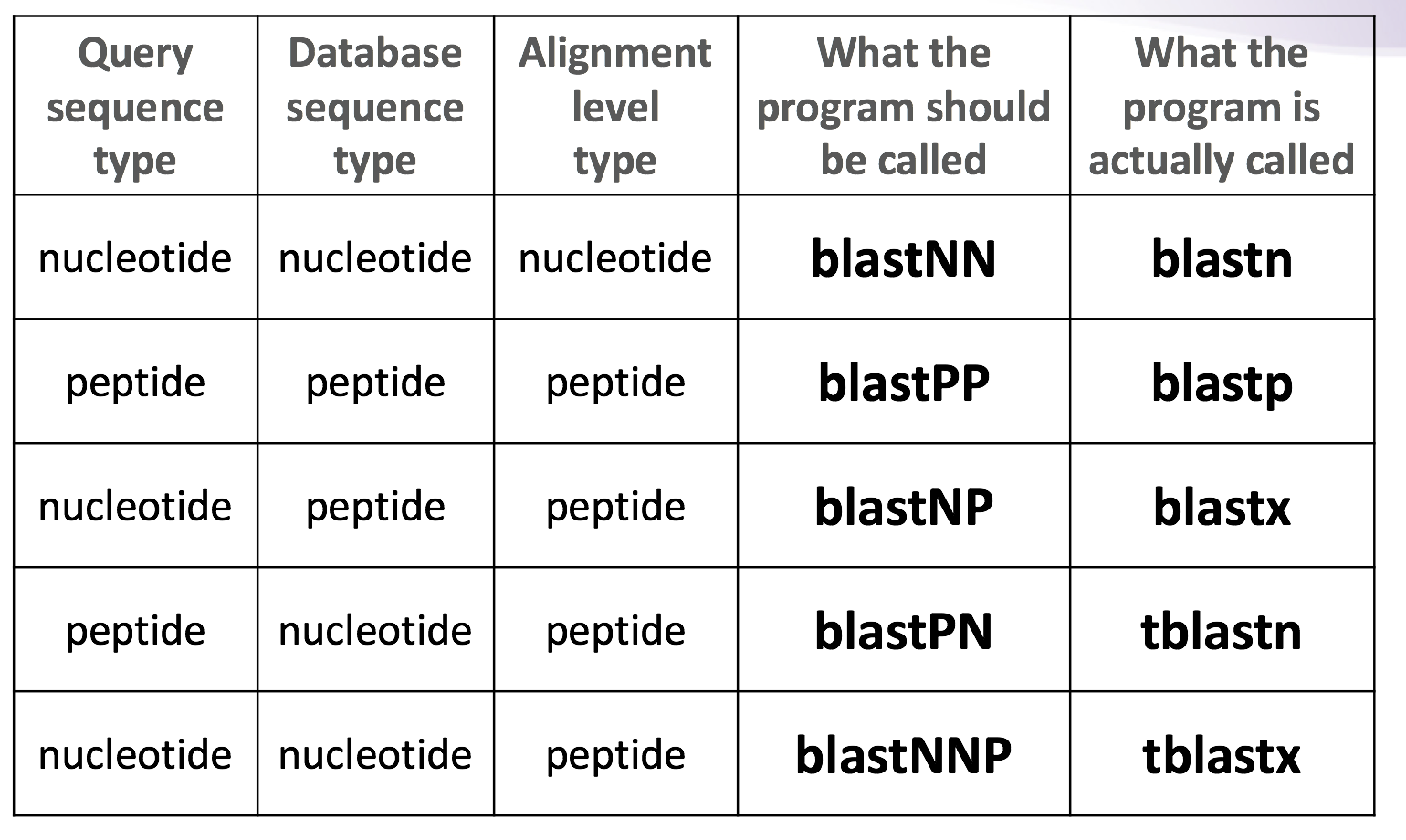
不同工具的应用范围虽然不同,但是基本参数都是一致的,学会blastn,基本上其他诸如blastp,blastx也都会了。
blastn的使用参数很多 blastn [-h] ,但是比较常用是如下几个
- -db : 数据库在本地的位置,或者是NCBI上数据库的类型,如 -db nr
- -query: 检索文件
- -query_loc : 指定检索的位置
- -strand: 搜索正义链还是反义链,还是都要
- out : 输出文件
- -remote: 可以用NCBI的远程数据库, 一般与 -db nr
- -evalue 科学计数法,比如说1e3,定义期望值阈值。E值表明在随机的情况下,其它序列与目标序列相似度要大于这条显示的序列的可能性。 与S值有关,S值表示两序列的同源性,分值越高表明它们之间相似的程度越大
E值总结: 1.E值适合于有一定长度,而且复杂度不能太低的序列。2. 当E值小于10-5时,表明两序列有较高的同源性,而不是因为计算错误。3. 当E值小于10-6时,表时两序列的同源性非常高,几乎没有必要再做确认。
与联配计分相关参数: -gapopen,打开gap的代价;-gapextend, gap延伸的代价;-penalty:核算错配的惩罚; -reward, 核酸正确匹配的奖励;
结果过滤:-perc_identity, 根据相似度
注 BLAST还提供一个task参数,感觉很有用的样子,好像会针对任务进行优化速度。
第三步:运行blast,调整输出格式。
我随机找了一段序列进行检索
echo '>test' > query.fa
echo TGAAAGCAAGAAGAGCGTTTGGTGGTTTCTTAACAAATCATTGCAACTCCACAAGGCGCCTGTAATAGACAGCTTGTGCATGGAACTTGGTCCACAGTGCCCTACCACTGATGATGTTGATATCGGAAAGTGGGTTGCAAAAGCTGTTGATTGTTTGGTGATGACGCTAACAATCAAGCTCCTCTGGT >> query.fa
用的是blastn 检索核酸数据库。最简单的用法就是提供数据库所在位置和需要检索的序列文件
blastn -db BLAST/TAIR10 -query query.fa
# 还可以指定检索序列的位置
blastn -db BLAST/TAIR10 -query query.fa -query_loc 20-100
# 或者使用远程数据库
blastn -db nr -remote -query query.fa
blastn -db nt -remote -query query.fa
以上是默认输出,blast的-outfmt选项提供个性化的选择。一共有18个选择,默认是0。
0 = Pairwise,
1 = Query-anchored showing identities,
2 = Query-anchored no identities,
3 = Flat query-anchored showing identities,
4 = Flat query-anchored no identities,
5 = BLAST XML,
6 = Tabular,
7 = Tabular with comment lines,
8 = Seqalign (Text ASN.1),
9 = Seqalign (Binary ASN.1),
10 = Comma-separated values,
11 = BLAST archive (ASN.1),
12 = Seqalign (JSON),
13 = Multiple-file BLAST JSON,
14 = Multiple-file BLAST XML2,
15 = Single-file BLAST JSON,
16 = Single-file BLAST XML2,
17 = Sequence Alignment/Map (SAM),
18 = Organism Report
其中6,7,10,17可以自定输出格式。默认是
qaccver saccver pident length mismatch gapopen qstart qend sstart send evalue bitscore
| 简写 | 含义 |
|---|---|
| qaccver | 查询的AC版本(与此类似的还有qseqid,qgi,qacc,与序列命名有关) |
| saccver | 目标的AC版本(于此类似的还有sseqid,sallseqid,sgi,sacc,sallacc,也是序列命名相关) |
| pident | 完全匹配百分比 (响应的nident则是匹配数) |
| length | 联配长度(另外slen表示查询序列总长度,qlen表示目标序列总长度) |
| mismatch | 错配数目 |
| gapopen | gap的数目 |
| qstart | 查询序列起始 |
| qstart | 查询序列结束 |
| sstart | 目标序列起始 |
| send | 目标序列结束 |
| evalue | 期望值 |
| bitscore | Bit得分 |
| score | 原始得分 |
AC: accession
以格式7为实例进行输出,并且对在线数据库进行检索
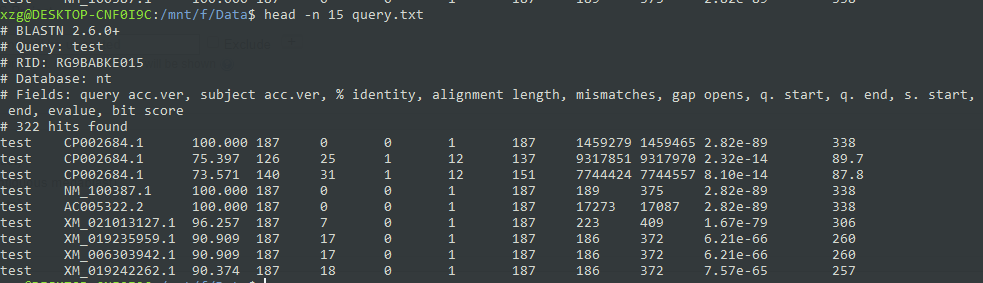
blastn -task blastn -remote -db nr -query query.fa -outfmt 7 -out query.txt
head -n 15 query.txt
术语列表
引自BLAST Glossary
参考资料
- Linux系统中NCBI BLAST+本地化教程
- BLAST Command Line Applications User Manual
这篇关于biostar handbook(七)| BLAST的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!