本文主要是介绍biostar handbook|如何模拟NGS测序结果,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
如何用软件模拟NGS数据
为了评价一个工具的性能,通常我们都需要先模拟一批数据。这样相当于有了参考答案,才能检查工具的实际表现情况。因此对于我们而言,面对一个新的功能,可以先用模拟的数据测试下不同工具的优缺点。有如下几个工具值得推荐一下:
- 'wgsim/dwgsim': 从全基因组中获取测序reads
- 'msbar': EMBOSS其中一个工具,能够从单个序列中模拟随机突变
- 'biosed': EMBOSS的一个工具,可以按照我们给定突变位点模拟
- 'ReadSim': 专门用于模拟PacBio/Nanopore这类仪器产生的long read
- 'Art': 目前最复杂的模拟工具,能够模拟测序仪测序引入的错误位点
- 'Metasim': 用于模拟宏基因组得到的reads
- 'Polyester': 用于模拟RNA-seq
值得注意的是,这些工具模拟效果是有限,比如建库操作中超声破碎会出现的误差就很难模拟。但是最好的用途就是看看不同生物学事件在数据的情况,比如说发生了“大规模倒置”的基因组得到的数据比对到参考基因组上会是什么情况。
使用dwgsim进行模拟
wgism和dwgsim能够根据参考基因组模拟出测序reads,主要是二倍体基因组的SNPs和插入缺失(INDEL)多态位点。wgism容易安装,但是参考答案是以简单的文本格式保存,不容易可视化。dwgsim受wgism启发,虽然安装稍微麻烦了点,但是参考答案是以VCF格式保存,很方便可视化。
# 请先安装好ncurse
# 安装dwgsim
mkdir -p ~/scr
mkdir -p ~/.local/bin
cd ~/src
git clone --recursive https://github.com/nh13/DWGSIM.git
cd DWGSIM
make
ln -s ~/src/DWGSIM/dwgsim ~/.local/bin/dwgsim
ln -s ~/src/DWGSIM/dwgsim_eval ~/.local/bin/dwgsim_eval
简单地模拟一批数据
# efetch 需要用到conda安装启动
# conda create -n entrez entrez-direct
# conda activate entrez
# 获取参考基因组
efetch -db=nuccore -format=fasta -id=AF086833 > genome.fa
# 模拟数据
~/.local/bin/dwgsim genome.fa data
会得到如下数据
|-- data.bfast.fastq.gz # 用于bfast
|-- data.bwa.read1.fastq.gz # 用于BWA的R1
|-- data.bwa.read2.fastq.gz # 用于BWA的R2
|-- data.mutations.txt
|-- data.mutations.vcf # VCF形式擦
随后将这批数据用BWA比对,以bcftools检测变异和参考答案比较一下。
# conda install bwa samtools bcftools
bwa index genome.fa
bwa mem genome.fa data.bwa.read1.fastq.gz data.bwa.read2.fastq.gz | samtools sort -o data.bwa.bam
samtools mpileup -uf genome.fa data.bwa.bam | bcftools call -mv -o data.bwa.vcf
samtools index data.bwa.bam
利用使用IGV可视化,检查分析结果和真集的一致性
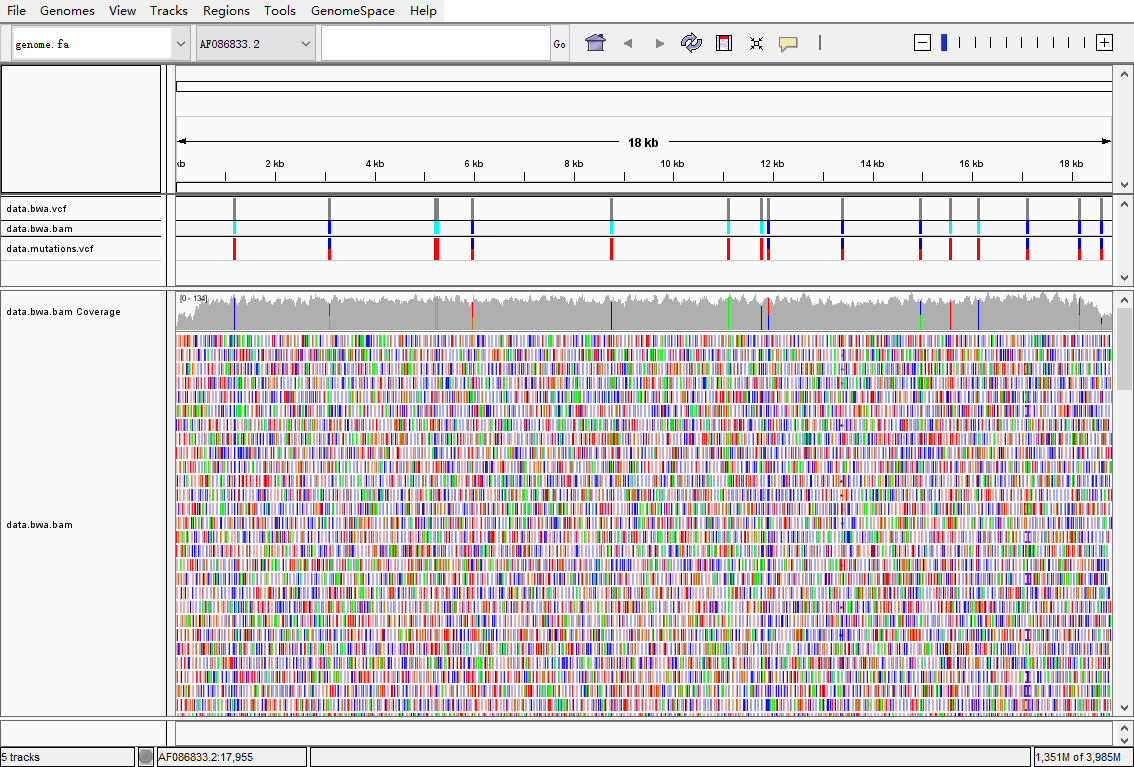
说明samtools+bcftools找变异这个组合其实还是靠谱的,至少在动植物领域研究里应该够用。
这篇关于biostar handbook|如何模拟NGS测序结果的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!









