本文主要是介绍蛋白质FASTA与药物分子SMILES数据集文本数据处理与可视化分析(一)2021SC@SDUSC,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
本文基于davis数据集进行操作2021SC@SDUSC
首先最直接的方式是学习一下别人的项目中是如何处理数据的
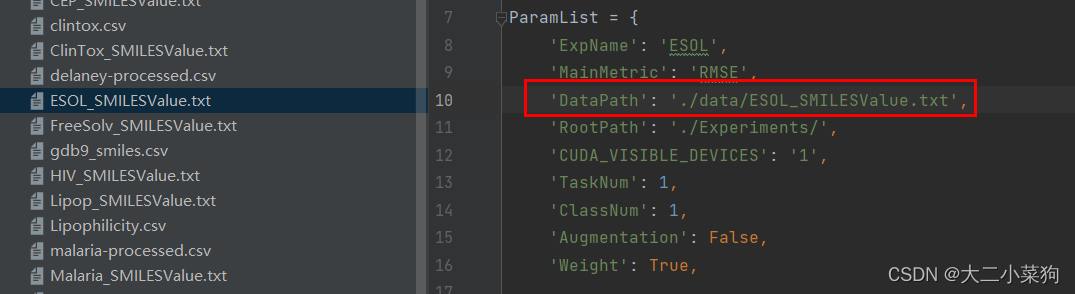
在项目代码中,指定了一个ParamList的字典用键值对的方式存储配置信息
下图为所选的数据集ESOL_SMILESValue.txt的结构,每一行数据逗号左侧为分子SMILES序列,右侧为该分子对应的label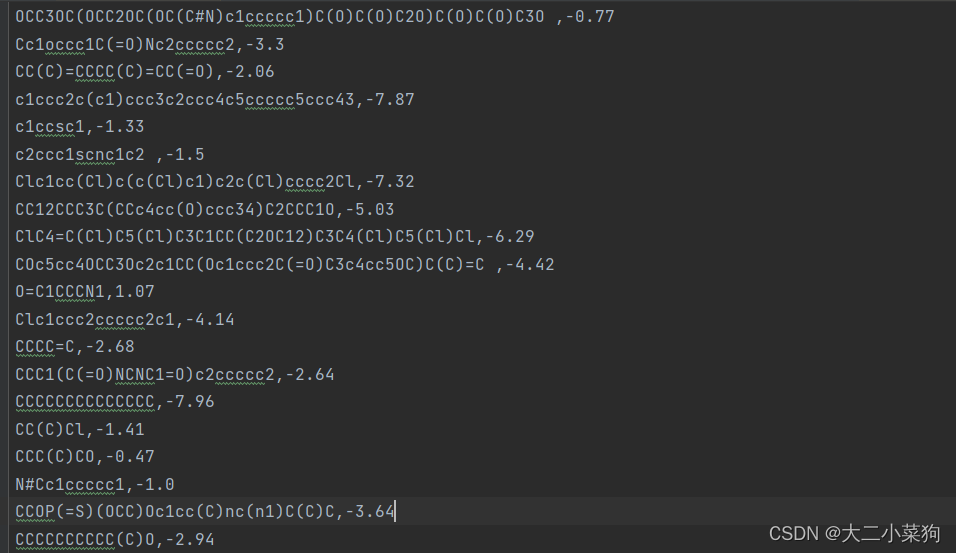
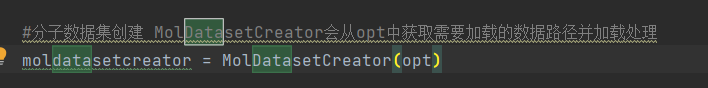
下一步就是把刚刚的ParamList(此次已封装到opt里),将opt作为参数送入MolDatasetCreator构造方法中创建一个对应的对象,后续会使用此对象进行进一步处理,接下来看看该构造方法有关opt部分的具体实现
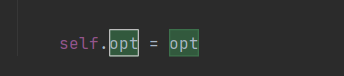
仅仅是将opt赋值给了self.opt
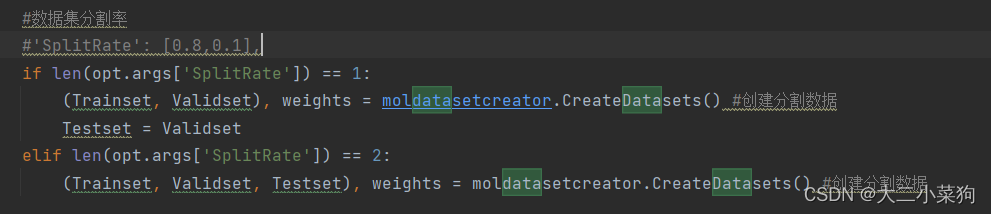
返回到上一层,下面随即使用maldatasetcreator调用CreateDatasets函数进行处理,接下来看实现
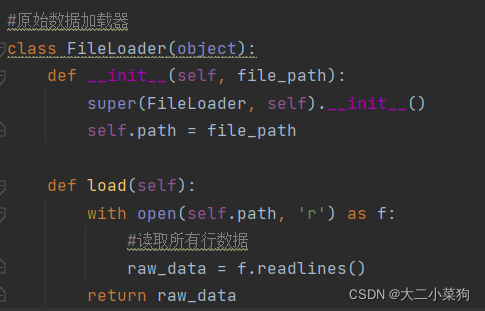
此函数开头会获取前面paramList里的DataPath键的值,也就是ESOL_SMILESValue.txt的路径,然后创建文件加载器fileloader,并对文件使用load函数,下图可以看到load函数会通过readlines读出文本文件中的所有行
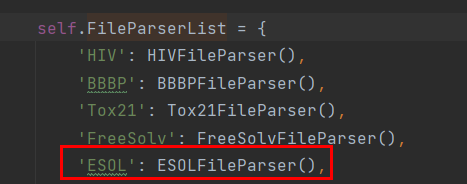
回到上层继续下面的执行,这一步是将opt中的ExpName对应的值作为FileParserList的键,又取得了一个值赋给到parser变量

可以看到,最终对应到的是ESOLFileParser(),从字面上猜测,应该是由于不同实验使用的数据集结构不同,所以要采用不同的parser进行处理
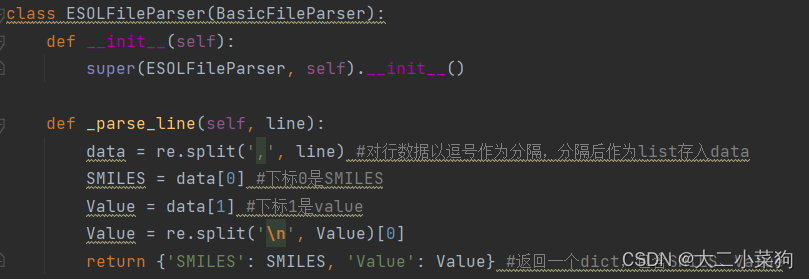
此次截取了两个parser类的实现,其中包括我们的ESOLFileParser,它们都有一个叫做_parse_line的函数,对于不同的数据集有着不同实现方式
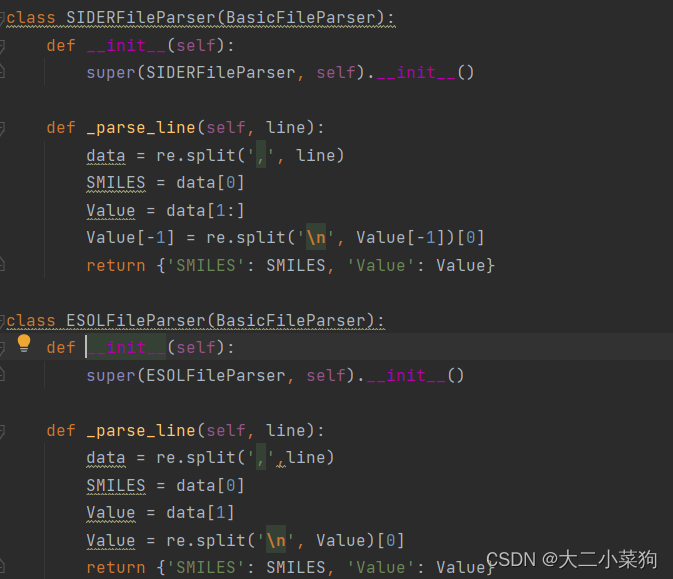
可以看到,对一行数据的处理,先使用re对文本进行逗号分隔,分隔结果返回一个list,将list赋值给data,再分别取下标0、1的元素赋值给SMILES和Value,最后打包为一个dict,至此完成对该数据集中一行文本数据的分隔处理
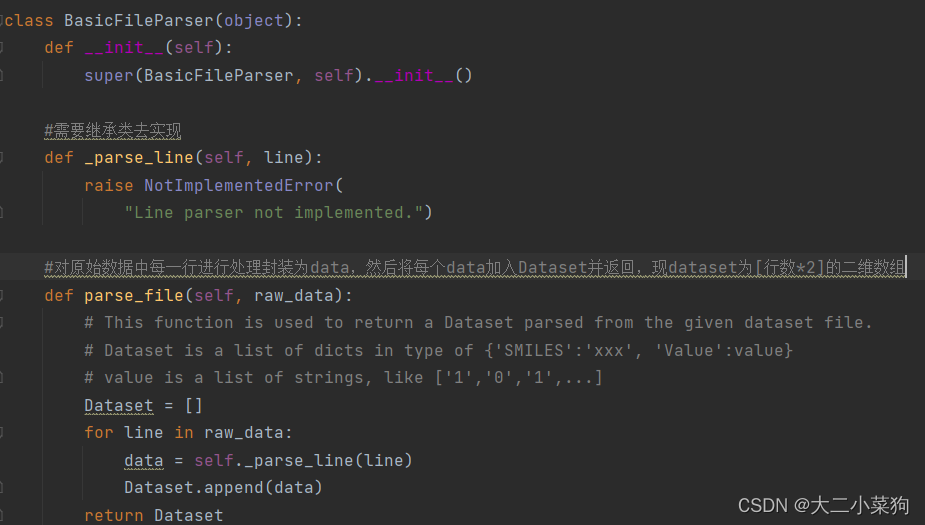
所有的parser都继承了这个basicFileParser,可以看到它有一个子类中没有重写的函数parse_file,作用见下图注释信息
至此,该ESOL中的数据集处理已经明确,下一篇我们会对DTA任务中基准数据集davis中的蛋白质序列进行进一步处理。
这篇关于蛋白质FASTA与药物分子SMILES数据集文本数据处理与可视化分析(一)2021SC@SDUSC的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!





