smiles专题
如何将化学分子SMILES字符串转化为Pytorch图数据结构——ESOL分子水溶性数据集解析
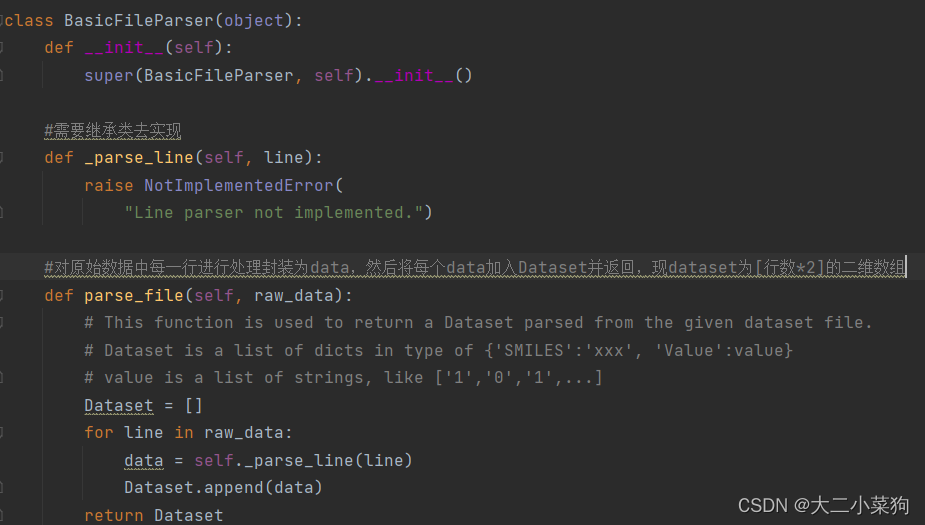
硬核原创,转载请注明出处: https://leytton.blog.csdn.net/article/details/130406553 一、前言 在用Pytorch图神经网络对化学分子进行数据分析的时候,经常使用现有的数据集。看到自动处理完毕的数据结构,里面的特征值让我们一脸懵逼,不知道代表的是什么含义。本文将带大家分析这些数据结构的来龙去脉。 二、数据原始特征 在使用图神经网络(GN
蛋白质FASTA与药物分子SMILES数据集文本数据处理与可视化分析(一)2021SC@SDUSC
本文基于davis数据集进行操作2021SC@SDUSC 首先最直接的方式是学习一下别人的项目中是如何处理数据的 在项目代码中,指定了一个ParamList的字典用键值对的方式存储配置信息 下图为所选的数据集ESOL_SMILESValue.txt的结构,每一行数据逗号左侧为分子SMILES序列,右侧为该分子对应的label 下一步就是把刚刚的ParamList(此次已封装到opt里),