tcga专题
【TCGA】命名方式
以下是TCGA-LUAD数据集中Diagnostic类型的一张WSI名称: · TCGA: Project,项目名称 · 44: Tissue Source Site,样本机构来源 · 7662: Participant,项目参与者 因此,TCGA-44-7662就是这张WSI的病人编号 · 01: Sample,样本组织来源类型 肿瘤类型为01-09,正常类型为10-19,对照组为20
【TCGA】将TCGA数据移动到一个文件夹下
下载过TCGA数据库中数据的都知道,下载后数据都是在多个文件夹下的,但是这样会使得后续的数据处理和分析工作非常繁琐。因此,本人用Python写了一个程序,可以将原数据移动到一个文件夹下。本程序以WSI为例,其他形式的数据同理,改个后缀即可。 import pandas as pdimport numpy as npimport osimport shutilimport argparse
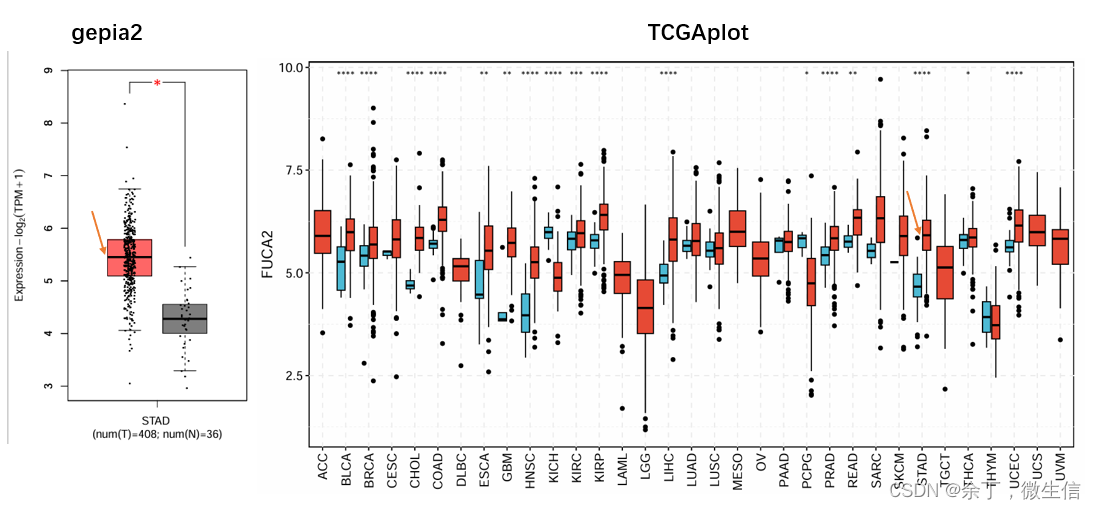
TCGAplot在线版:输入基因,一键绘制TCGA泛癌肿瘤vs正常box图
1.TCGA简介 TCGA(The Cancer Genome Atlas)收集了大量癌症样本的数据,涉及33种不同癌症、超过 20000个样本,包括外显子组测序、RNA 测序、microRNA 测序、拷贝数变异、蛋白质组和甲基化组,临床信息等数据,研究者可以利用各种生物信息学工具和统计方法来挖掘数据中的有用信息,推动癌症研究的进展。 2.TCGAplot简介 华科同济医院的王雄老师课题组利
TCGA数据下载及矩阵整理
首先我们进入TCGA数据库TCGA官网 首先看一下文件类型,悬着数据处理方式及工作流程 看一下例子里面各种类型,有组织是什么,癌症项目。 点击进入购物车 下载所有文件点击cart 所有压缩文件合并到一个文件内 ###将所有压缩包移到一个名为files的文件里面use strict;use warnings;use File::Copy;my $newDir="files";unl
TCGA超过1G的病理wsi数据下载-gdc-client
使用网页端下载TCGA超过1G的病理wsi数据,数据下载到1G后就不能完整下载。遂采用gdc-client下载。 Win 环境下新建这个文件夹放在系统盘进行储存,否则会报错:ERROR: Unable to write state file: [WinError 17] 系统无法将文件移到不同的磁盘驱动器。好像是 TCGA数据集下载及gdc-client相关问题汇总_gdc client er
ChAMP分析TCGA结直肠癌甲基化数据实战
前面用几篇推文详细介绍了ChAMP包用于甲基化分析的流程,并使用肠癌领域的GSE149282进行了演示。 16.ChAMP分析甲基化数据:标准流程 17.ChAMP分析甲基化数据:从β值矩阵开始的流程 下面我们用TCGA-COAD和TCGA-READ的甲基化数据再做一次演示,从IDAT文件开始。 有了这个结果之后,你就可以去做各种联合分析~ 太长不看版 下面的步骤都是在服务器上做的,因
TCGA相关分析之数据筛选 | python从TCGA-GBM的RNA-seq表达数据count中筛选出各genes对应的案例cases的表达量count矩阵
接上一篇文章,现在开始筛选数据组成count矩阵。 上一篇:TCGA下载GBM患者的RNA-seq数据 上一篇结束,下载到初始数据(图一图二是下载之后的文件夹以及每一个文件夹中的count数据文件) 需要从每一个count数据文件中筛选出gene_name、gene_type为lncRNA、FPKM表达量,效果图如下: 由于不会R语言,就用python来实现 步骤: 从每一
零代码复现-TCGA联合GEO免疫基因结合代谢基因生信套路(一)
经过一段时间的积累,搭建的分析平台已经日渐进入稳定的状态,很多粉丝也在和我们反馈,让我们出一下零代码生信套路课。 小编找了很久,发现某某机构出的TCGA联合GEO 免疫基因+代谢基因的生信套路,该套路应用常见相对来说比较广,又属于双热点套路。但是代码运行真的是太麻烦了,还有各种各样的报错,实在是不好弄,为此,我们推出了复现课程,该课程内容较长, 分成几篇进行讲解,如带来不便,请大家谅解! 一、
零代码复现-TCGA联合GEO免疫基因结合代谢基因生信套路(二)
零代码复现-TCGA联合GEO免疫基因结合代谢基因生信套路(二)-关键基因集的获取和生存数据准备 前面的分析中,下载TCGA和GEO的数据,并进行简单的处理,接下来就是相关基因集的获取和整理,为后期聚类和降维做准备工作 三、获取免疫基因和代谢相关的基因集 这里可以选择从GSEA官网下载免疫和代谢相关基因,或者换成其他的基因也是可以的 1、取对数,TCGA的数据一般跨度比较大,所以在使用li
TCGA病理图像深度学习的肿瘤淋巴细胞浸润(文献)
Spatial Organization and Molecular Correlation of Tumor-Infiltrating Lymphocytes Using Deep Learning on Pathology Images - ScienceDirect 该文献展示了:基于13种TCGA肿瘤类型的H&E图像的肿瘤浸润淋巴细胞(TIL)的映射。 流程 方法使用两种CNN: