bionano专题
「BioNano系列」那些Bionano未覆盖的区域是什么?
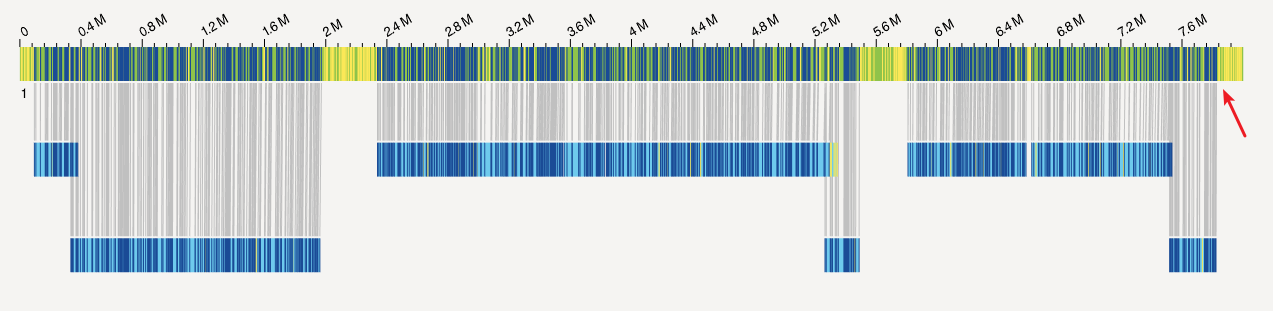
在「Bionano系列」光学图谱混合组装应该怎么做?这篇文章中,我展示了下面这张图。 未覆盖区域 和之前的图不同的是,我加了几个箭头,这些箭头所指向的区域的特征就是,这些区域并未被Bionano所覆盖。如果不去思考这些区域到底是什么,直接进行混合组装,那么这其实对最后结果的不负责任。因为这完全可能是组装软件没有正确的处理错误的overlap,将不应该连接的序列连接在一起(尽
「BioNano系列」光学图谱混合组装应该怎么做?
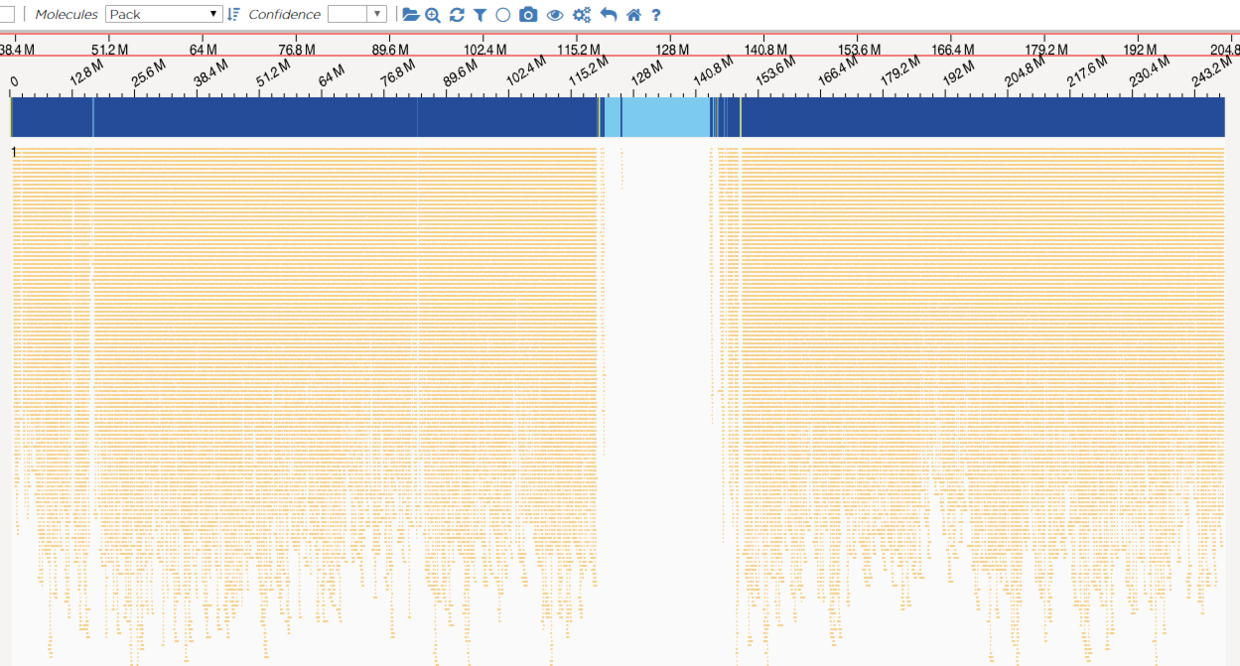
评估从头组装结果 Bionano从头组装出光学图谱CMAP可以和参考序列的CMAP进行比对,通过Access上可视化检查参考基因组的组装质量,比较两者间的不同。 这里所用的CMAP图谱来自于一篇发表在NC的拟南芥的基因组文章(原本计划用他们的bnx文件介绍从头组装,但是通讯作者根本不搭理我), 光学图谱的下载方式为: wget https://submit.ncbi.nlm.nih.gov
「BioNano系列」如何从头组装出一个Bionano图谱
官方并没有一个很详细的文档描述Bionano的从头组装流程的具体过程,所以我只能根据自己实际项目进行介绍: 流程 AutoNoise + SplitBNX: 这一步会将bnx和参考的cmap文件进行比对,估算出噪声系数,然后把bnx进行拆分便与后续比对Pairwse: 这一步进行molecules之间的两两比较,寻找overlap, 结果存放在"align"文件夹下Asse
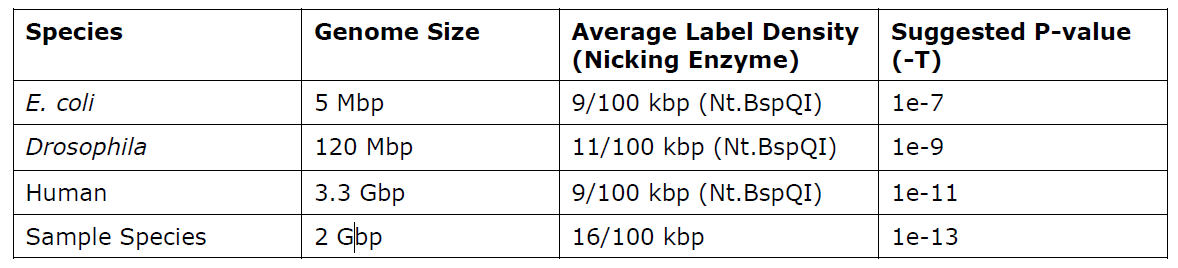
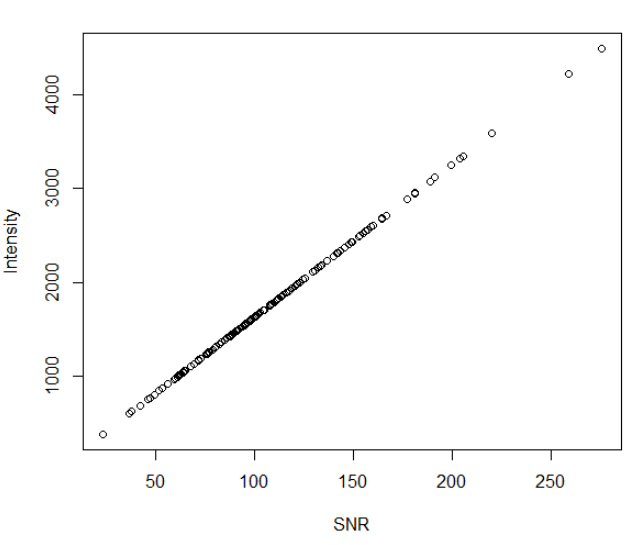
「Bionano系列」下机原始数据过滤和评估
从这部分开始,就开始涉及一些软件的操作和数据分析,因此在进入正文之前,我们需要准备好环境。 环境准备 第一步:从 https://bionanogenomics.com/library/datasets/下载人类测试数据集,以及对应的NA12878人类基因组。 wget http://bnxinstall.com/publicdatasets/DLS/20180413_NA12878_S
「Bionano系列」下机数据的BNX文件到底说了什么
最近我拿到了一批Bionano数据,用关键字 “Bionano+组装” 进行检索时,并没有发现任何的教程,所以这应是中文网络世界里第一个Bionano数据分析系列 Bionano技术简单来说,就是给分子加上荧光标记,然后拍照,所以最原始的下机数据就是TIFF格式,但是用户拿到的一般都是经AutoDetect/IrysView 转换过的BNX格式。这篇文章主要就是讲讲BNX格式的具体含义。
「BioNano系列」如何进行cmap之间的比对
BioNano以cmap格式存放光学图谱,为了评估基因组的组装质量或者了解光学图谱中冗余情况(高杂合基因组组装结果偏大),我们就需要进行cmap之间的比较。 CMAP间比对 Solve套件提供了runCharacterize.py脚本封装了RefAligner,用于进行CMAP之间的比对。 python2.7 runCharacterize.py \-t RefAligner的二进制文件路径