本文主要是介绍「Bionano系列」下机原始数据过滤和评估,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
从这部分开始,就开始涉及一些软件的操作和数据分析,因此在进入正文之前,我们需要准备好环境。
环境准备
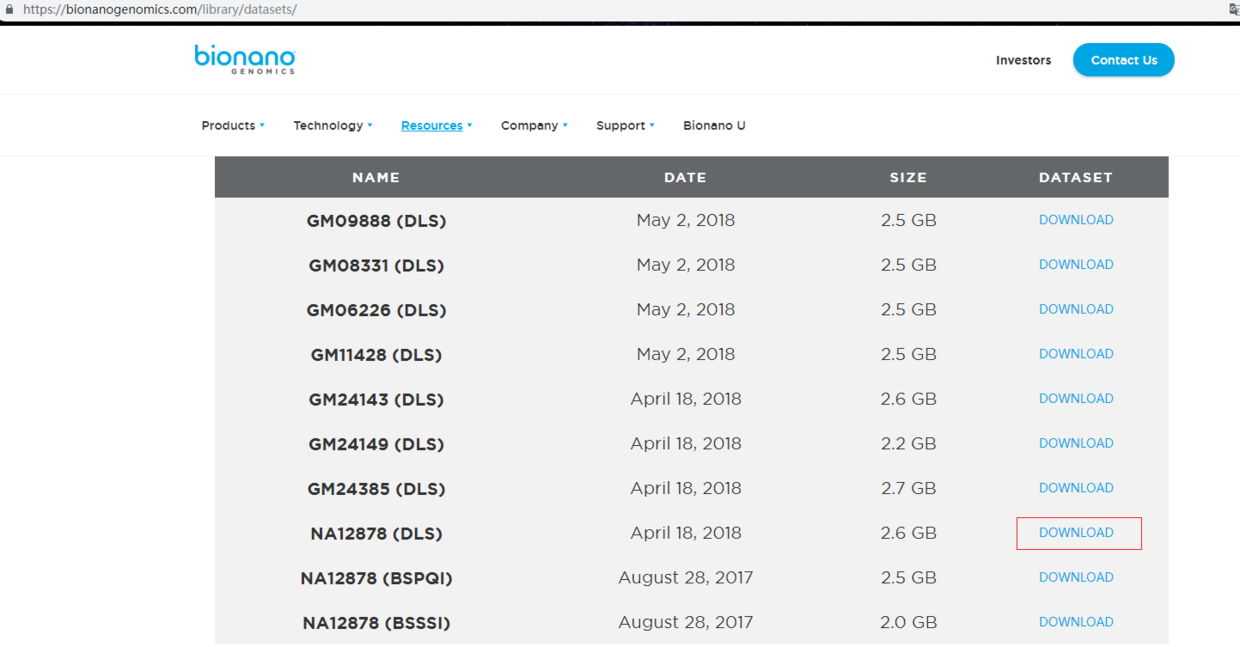
第一步:从 https://bionanogenomics.com/library/datasets/下载人类测试数据集,以及对应的NA12878人类基因组。
wget http://bnxinstall.com/publicdatasets/DLS/20180413_NA12878_S3_compressed.tar.gz
tar xf 20180413_NA12878_S3_compressed.tar.gz
wget ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCA/002/077/035/GCA_002077035.3_NA12878_prelim_3.0/GCA_002077035.3_NA12878_prelim_3.0_genomic.fna.gz
gunzip GCA_002077035.3_NA12878_prelim_3.0_genomic.fna.gz
mv GCA_002077035.3_NA12878_prelim_3.0_genomic.fna NA12878.fa
第二步: 在https://bionanogenomics.com/support/software-downloads/下载Solve软件,

服务器要满足如下需求:
- Python=2.7.x
- perl=5.14.x或5.16.x
- R > 3.1.2,并且安装data.table, igraph, intervals, MASS, parallel, XML, argparser
- glibc >= 2.14 和 gcc库
- 至少有一个256G节点的内存,最好有一些32G内存的小节点
tar -zxvf Solve3.3_10252018.tar.gz
解压缩后里有如下几个文件夹
- cohortQC: 主要是MQR运行脚本
- HybridScaffold: 单酶系统和双酶系统混合组装工具脚本
- Pipeline:从头组装的脚本
- RefAligner:用于序列联配和组装
- RefGenome:hg19和hg38的cmp文件
- SVMerge: 用于合并单酶系统得到SV结果
- UTIL: 运行从头组装的一些实用shell脚本,可以根据需要进行修改
- VariantAnnotation: 对找到的SV进行注释
- VCFConverter: 将SMAP和SVMerge的结果输出成VCF格式
数据过滤
目前的主流Bionano设备已经是Sapjyr,BNX文件产生于Saphyr,经由Bionano Access 下载到本地。
从公司拿到的是"RawMolecules.bnx"文件, 不过我们练习用的数据是"output/all.bnx.gz",
mkdir test
mv output/all.bnx.gz test
zcat all.bnx.gz| head -n 20000 | grep "# Run Data" | wc -l
# 281
我们发现发现数据集来自于281个通道。
Label SNR 过滤: 过滤信噪比较低的分子,信噪比低意味着质量差。 有如下几个情况,不需要做Label SNR 过滤,或在你BNX文件的"#rh"部分有"SNRFilterType"定义,就不需要过滤
- 人类样本不需要过滤。
- Bionanao Access 1.2 以后新的图像检测算法得到的BNX文件不需要SNR过滤.
- 对于AutoDetect或Irysview处理过的数据,默认会进行label SNR过滤,处理之后就是Molecules.bnx
由于我们是人类数据集,因此下面的代码就不需要运行了,并且绝大部分情况下也用到下面的命令。
perl /opt/biosoft/Solve3.3_10252018/cohortQC/10252018/filter_SNR_dynamic.pl -i RawMolecules.bnx -o Molecules_filter.bnx -P diag_hist.pdf > snr.log &
分子长度过滤: 过滤短与某个阈值的分子,公司一般会只保留100kb或120Kb以上的分子(取决于数据量,数据越多,阈值越高)
gunzip all.bnx.gz
RefAlinger -i all.bnx -minlen 120 -merge -o output -bnx > run.log &
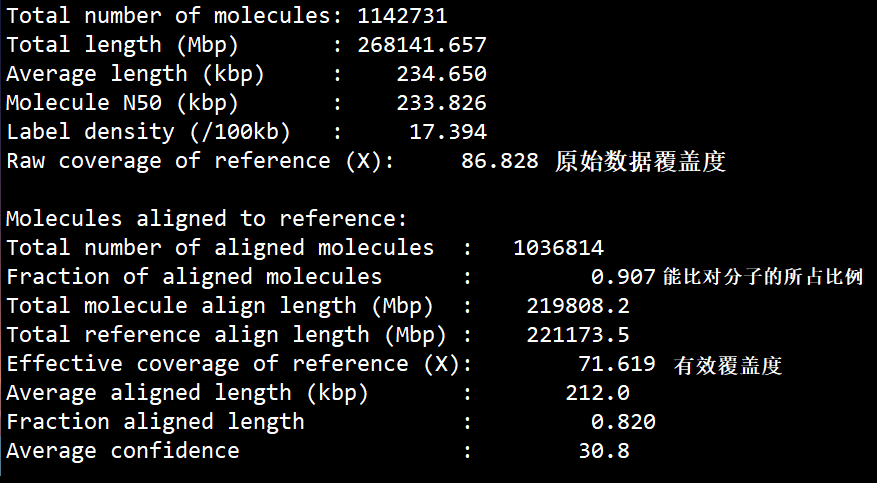
在输出的内容中,注意如下部分
Final maps=1147524, sites=46764680, length= 268845841.028kb (avg= 234.283 kb, label density= 17.395 /100kb)
表明过滤后,还有113万条分子,涉及到4676万标记,总测序量为258G,标记密度是17.395/100kb,平均分子长度大于234Kb… 测序深度等于总测序量除以基因组大小,这是人类基因组,按照3G计算,那么深度就是80X.
过滤后平均分子长度应大于200Kb。 标记密度不能过高,过高会因分辨率不够而无法区分,过低则无法用于比对。一般DLS在10~25 , NRLS在8~15.
组装评估: 在正式组装之前,我们还需要判断下当前数据是否满足组装要求。 为了获取所需的评估参数,得将过滤后的BNX文件和基因组模拟模切得到的CMAP进行比对
第一步: 对基因组序列模拟酶切,得到CMAP文件
perl /opt/biosoft/Solve3.3_10252018/Pipeline/10252018/fa2cmap_multi_color.pl -i ../NA12878.fa -e cttaag 1
mv ../NA12878_CTTAAG_0kb_0labels.cmap .
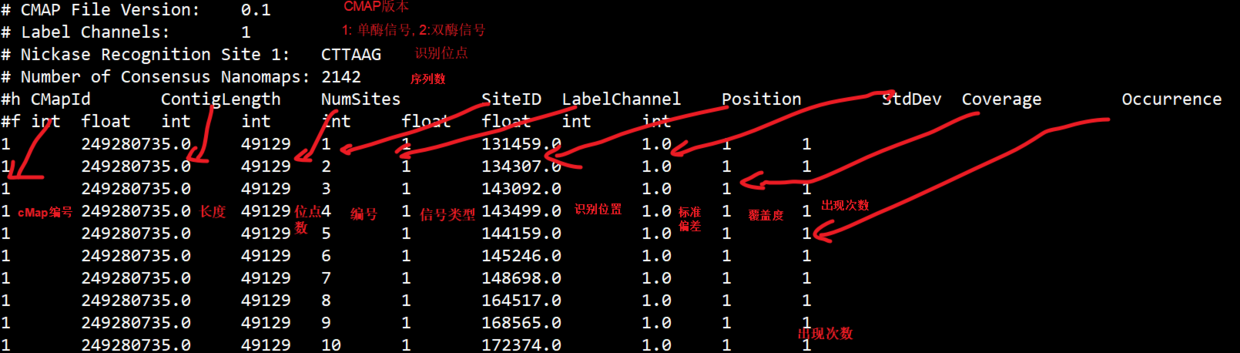
CMAP的格式比较简单,说明如下:
第二步: 用align_bnx_to_cmap.py进行比对。 bionano光学图谱比对的基本原理是基于标记的相对位置。
python /opt/biosoft/Solve3.3_10252018/Pipeline/10252018/align_bnx_to_cmap.py \--prefix human \--mol molecules120k.bnx \--ref NA12878_CTTAAG_0kb_0labels.cmap \--ra /opt/biosoft/Solve3.3_10252018/RefAligner/7915.7989rel \--nthreads 80 \--output prealign \--snrFilter 2 \--color 1
参数说明可自行阅读帮助说明。我们重点关注输出结果中如下几方面内容:
- “contigs/alignmolvref/alignmol_log_simple.txt”里的“Fraction of aligned molecules”和"Effective coverage of reference (X)". 我们要判断数据是否符合最低的比对率。比对率和基因组实际情况有关(组装质量,错误率,重复坍缩情况)。对于人类基因可以达到90%以上,对于不怎么完整度的基因组,即便Bionano的质量很高,比对率可能也只有30%~40%(仅统计150 kb 的分子)
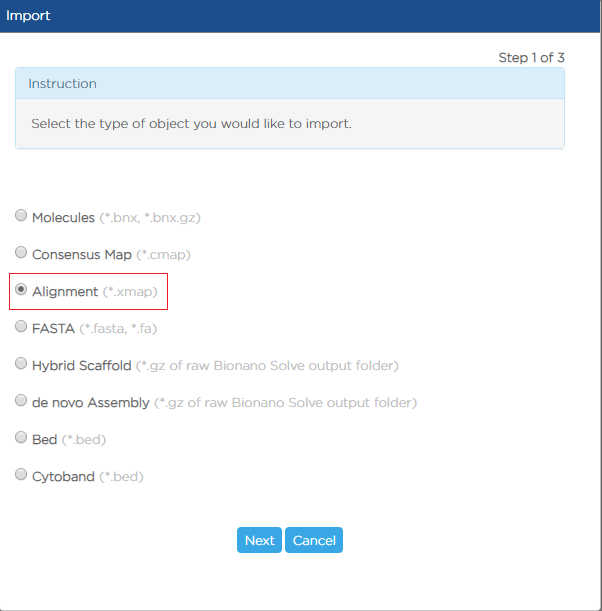
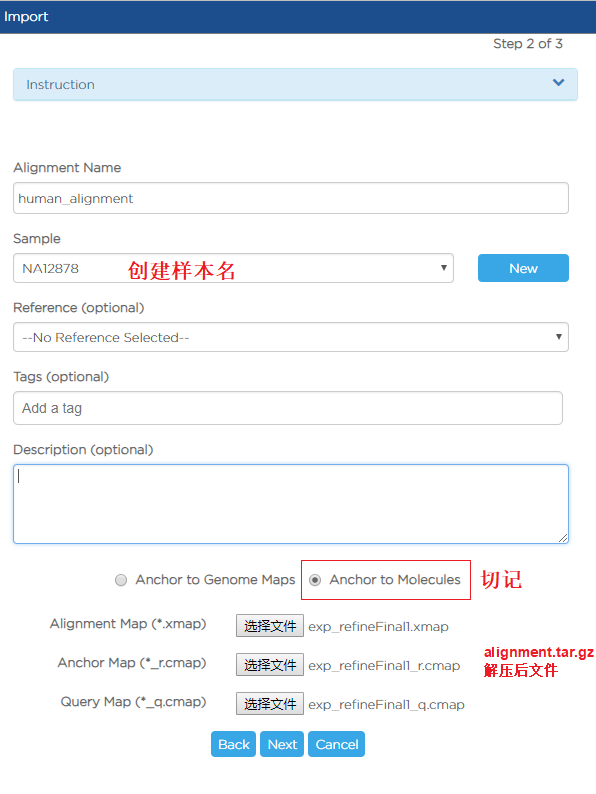
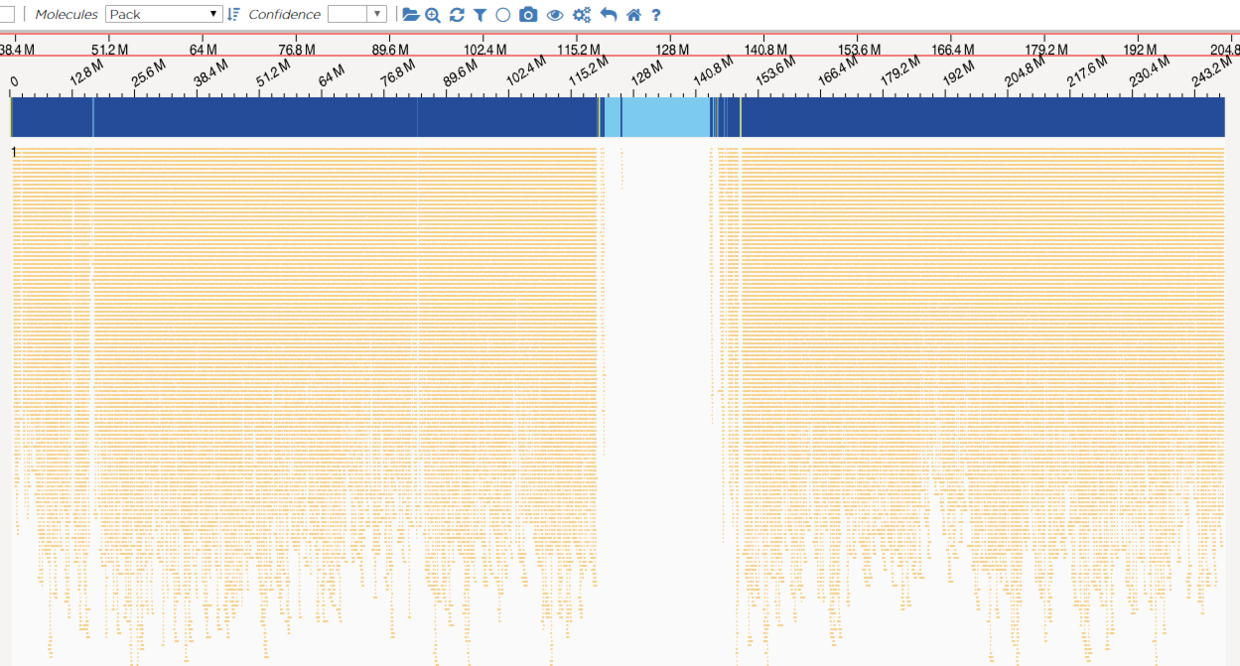
- “alignments.tar.gz”, 里面包含的三个文件可以输入到Bionano提供的另一个工具Access中进行可视化,注意导入要选择"Anchor to Molecules"。
由于人类基因组足够的大,因此需要等待一段时间才能处理完成,之后就可以对比对结果有一个更加直观的了解。
那么合格后的数据应该如何组装呢?请等待后续教程!
这篇关于「Bionano系列」下机原始数据过滤和评估的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!