本文主要是介绍「BioNano系列」如何从头组装出一个Bionano图谱,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
官方并没有一个很详细的文档描述Bionano的从头组装流程的具体过程,所以我只能根据自己实际项目进行介绍:
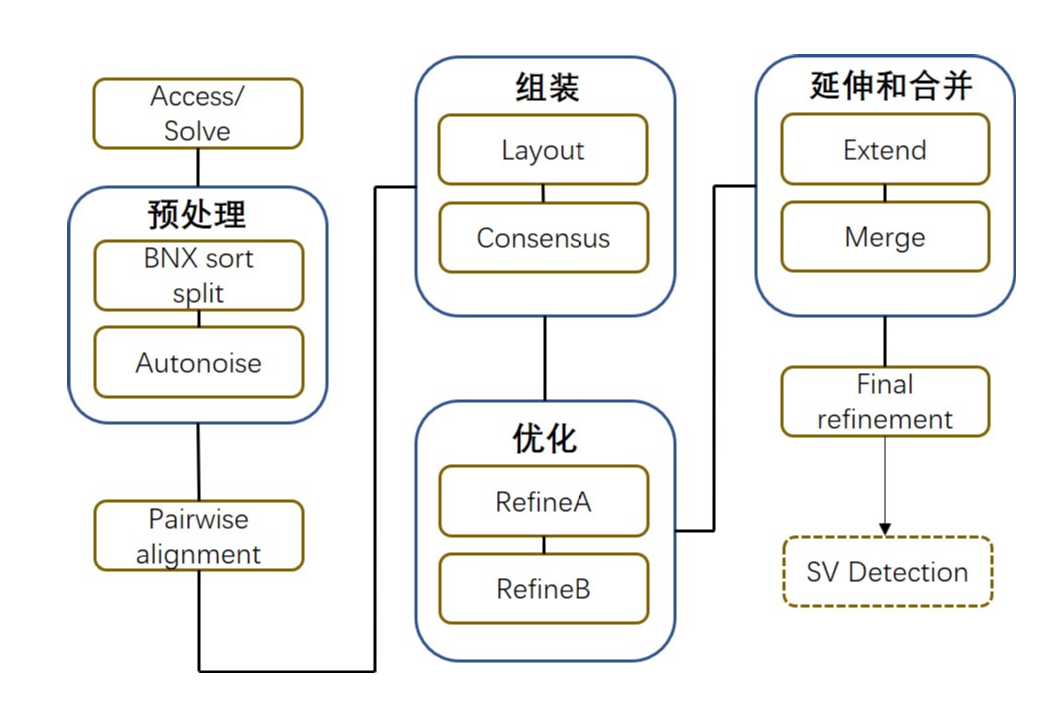
- AutoNoise + SplitBNX: 这一步会将bnx和参考的cmap文件进行比对,估算出噪声系数,然后把bnx进行拆分便与后续比对
- Pairwse: 这一步进行molecules之间的两两比较,寻找overlap, 结果存放在"align"文件夹下
- Assembly: 根据两两比对结果,通过OLC算法进行组装,结果在"contigs/exp_unrefined"下,合并后的文件为"EXP_UNREFINED.cmap", 同时还会将"EXP_UNREFINED.cmap"和参考基因组的cmap进行比对,结果放在"contigs/exp_unrefined/exp_unrefined/alignref", 此外还将拆分后Bnx文件和参考基因组的cmap文件进行比对,结果放在"contigs/alignmolvref"下
- refineA: 第3步得到的图谱先会进行第一次优化 输出结果在"contigs/exp_refineA", 这一步不会使用所有的原始数据,而是pairwise阶段用的质量比较好的分子,所以速度会快一些
- refineB: 在第4步的基础上进行第二次的优化, 输出结果在"contigs/exp_refineB0"和"contigs/exp_refineB1". 这一步会使用所有的输入原始数据,速度稍微慢一些。第一轮和第二轮的结果会将地覆盖度的区域进行打断,然后更新标记和标记的位置。
- Extension and merge: 将上一步的contig回贴到参考基因组的map,进行延伸和合并,这一步可以迭代3-5次。中间结果在"contigs/exp_extensionX_X"和"contigs/exp_mrgX"
- Final refinement: 最后一步的优化
- SV Decetion: 在有参考基因组的前提下,最后还会寻找一些大规模的结构变异。
上述这些流程由Solve工具中Pipeline文件夹下的脚本pipelineCL.py进行控制,以之前的过滤后的人类数据为例
python /opt/biosoft/Solve3.3_10252018/Pipeline/10252018/pipelineCL.py \-T 96 -N 4 -f 1 \-i 5 \ # 延伸和合并的迭代次数,默认是1,介于0~20之间-b molecules120k.bnx \ # 输入的BNX文件-r NA12878_CTTAAG_0kb_0labels.cmap \ #参考的reference,可选-l Assembly \ # 输出文件夹-y \ # 自动确定噪声参数,需要-r-t /opt/biosoft/Solve3.3_10252018/RefAligner/7915.7989rel \ # RefAligner和Assembler的所在文件夹-a /opt/biosoft/Solve3.3_10252018/RefAligner/7915.7989rel/optArguments_nonhaplotype_saphyr_human.xml # 配置参数文件
上面的-a参数最为重要,因为指定的xml文件控制了流程中每一步的具体参数,所以要慎重选择。
重点1: XML文件命名解释说明:
- irys/saphyr: 数据来源仪器
- DLE1: DEL1标记系统
- human: 物种是人类
- BG: big genome. 大于5G,主要是优化内存使用
- noES: no extend and split: 即便等位基因里有超过30kbp的结构变异,也不要将他们分开
- haplotype/nonhaplotype: 单倍型优化指的是将那些含有超过500bp或者更大的SV差异的等位基因进行分开,不推荐用于非人类基因组组装是用haplotype,这会导致组装结果过度碎片化,组装的基因组会变大.
- nocut: 不对CMPR(complex multipath regions)进行拆分,所谓的CMPR指的是长度超过130kbp的高度重复序列,因为相似度过高,组装的时候不知道如何处理。
对于非人类的物种,推荐参数为:
- 除非是小鼠的SV缺失,大部分情况都用nonhaplotype,用于后续的Hybrid scaffold Pipeline(HS)
- 要noES
- 是否cut看情况而定。大部分人喜欢不cut。
对于人类, 推荐参数为:
- 仅在SV检测时用haplotype, 对于HS用nonhaplotype
- 加上ES
- 大部分情况下加上CMPR, 除非你知道在CMPR区间上有SV,才使用nocut
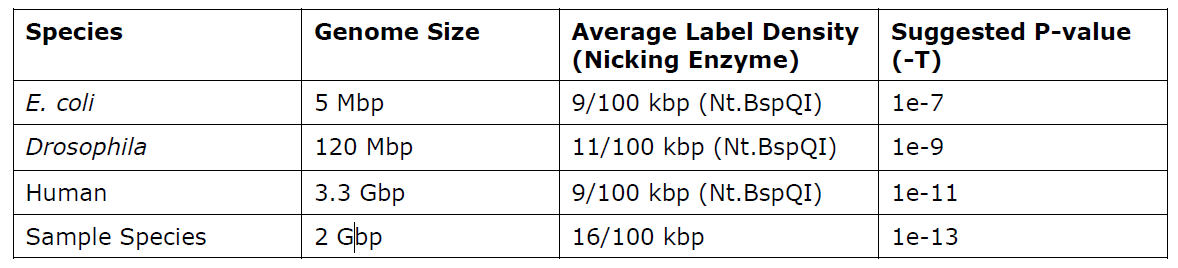
重点2: 如何设置组装时pairwise alignment中的-T参数。 基本原则是: 标记每增加一个,p值降低100倍; 基因组每增加一个数量级,p值降低100倍。
- 对于大于1Gb, 标记密度小于 15/100bkp的情况,设置为1e-11,
- 对于基因组大于1Gb, 标记密度大于15/100 kbp,每增加一个标记,就降低100倍,例如17/100kbp, 推荐1e-15
此外可以用-B跳过部分流程,-e则是输出文件的前缀,-x表示在自动去噪后退出。 如果有集群可用参数-C。如果基因组比较差, 可用-R 参数进行初步组装,然后基于第一个版本进一步的组装。还有一些参数用于关闭一些默认启动的功能
- -A: 不将bnx文件和最后的contig比较
- -m: 不将bnx和参考基因组比较
- -E: 不检查输出的完整性
运行过程中, 可以用"grep 'Executing' bionanoAssembly/exp_pipelineReport.txt" 查看执行进度
最后输出结果是"contigs/exp_refineFinal1/EXP_REFINEFINAL1.cmap",而结果好坏则要看"exp_informaticsReportSimple.txt", 两个核心标准
- Bionano图谱应该占原来的物理图谱的90%以上
- Bionano图谱的N50 会由于物种不同有很大差异,动物一般在1M以上,植物不确定。DLE系统差异更明显
这篇关于「BioNano系列」如何从头组装出一个Bionano图谱的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!









