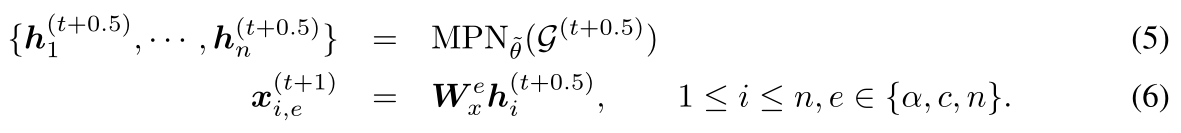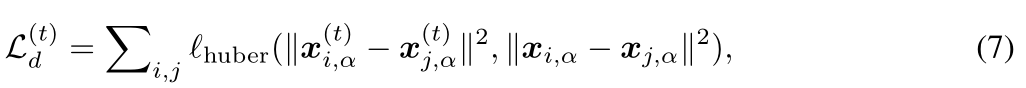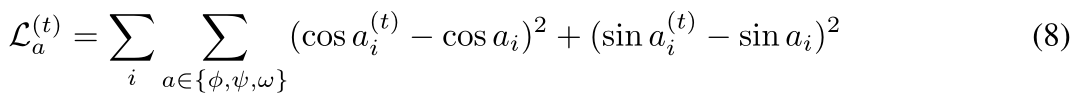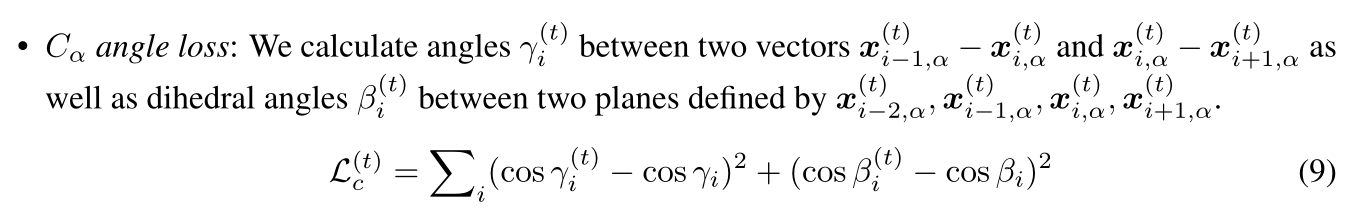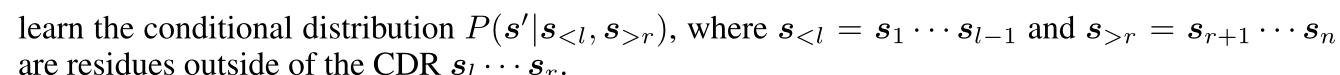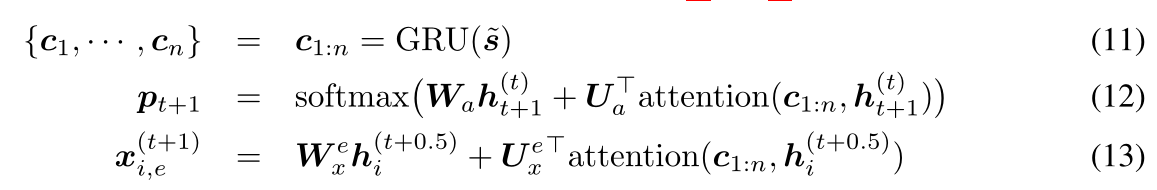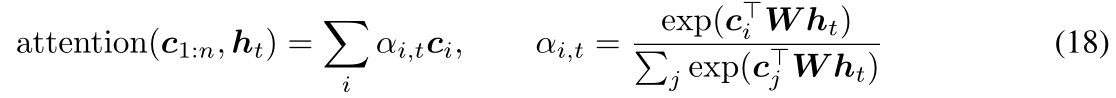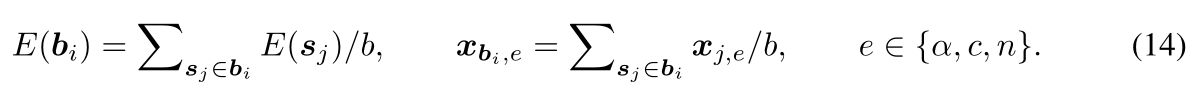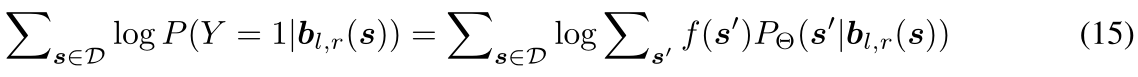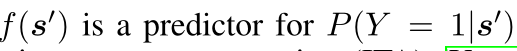本文主要是介绍生成抗体的生成GNN:Iterative Refinement Graph Neural Network for Antibody Sequence-Structure Co-design,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
网址: Iterative Refinement Graph Neural Network for Antibody Sequence-Structure Co-design | OpenReview
ICLR 2022的高分论文[8,8,8],目前没有给代码
内容:抗体结合的特异性是由这些Y形蛋白末端的互补决定区(CDR)决定的。这篇论文提出了一个生成模型来同时生成CDR序列和相应的结构,并具有迭代修改已生成子图的能力
有关抗体设计的一些问题
- 挑战:CDR序列的组合搜索空间到了20的60次方,而同时满足亲和力、稳定性与可合成性的解空间较小
- CDR生成的三个关键问题
- 如何对序列及其底层3D结构之间的关系进行建模-->同时设计
- 不考虑结构生成序列会导致次优性能
- 预定义结构再生成序列不适合抗体,因为结构很少是先验已知的
- 如何在给定序列(上下文)剩余部分的情况下对CDR的条件分布进行建模,注意力方法忽略了结构,而结构也是至关重要的
- 如何针对各种属性进行模型优化。传统基于物理的模型关注结合能最小化,本文模型关注的更多
- 如何对序列及其底层3D结构之间的关系进行建模-->同时设计
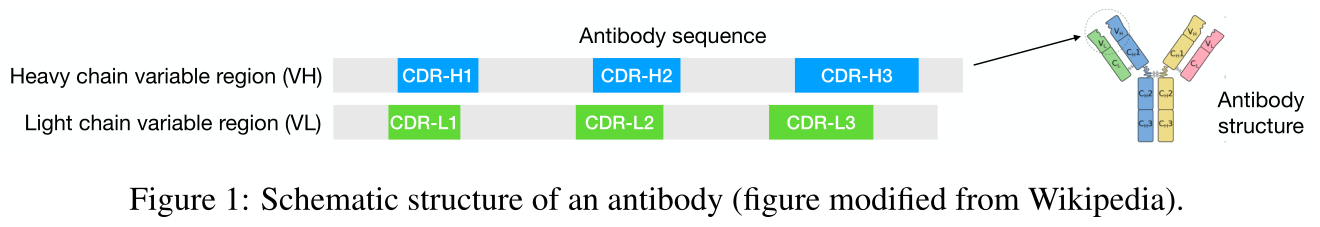
抗体的组成
- 抗体由一个重链(heavy chain)和一个轻链(light chain)组成,每个重链由一个可变区variable domain (VH/VL)和一些恒定区constant domain 组成。可变区进一步划分为框架区和三个补充性决定区(CDR)。重链上的三个CDR被标记为CDR-H1、CDR-H2、CDR-H3,每个CDR占据一个连续的序列。作为抗体中变异最大的部分,CDRs是结合和中和的主要决定因素-->抗体的设计任务可以认为是CDR的生成任务
模型结构
- 本文只关注VH的CDR的生成,VL的同理
- VH可表示为氨基酸的序列𝑠=𝑠1𝑠2…𝑠𝑛,其中每个si可称为残基(residue)
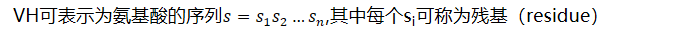
- 残基的值为20中氨基酸中的一个,或者<MASK>
- 使用主干坐标将其折叠为3D结构
- 阿尔法碳:𝑥𝑖,𝛼
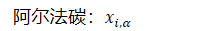
- 碳原子:𝑥𝑖,𝑐
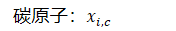
- 氮原子:𝑥𝑖,𝑛
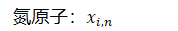
- 阿尔法碳:𝑥𝑖,𝛼
- VH可表示为氨基酸的序列𝑠=𝑠1𝑠2…𝑠𝑛,其中每个si可称为残基(residue)
VH的表示
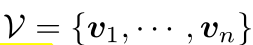
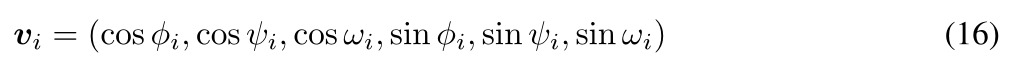
- 这三个角与残基的主干坐标有关。对于每个残基,计算一个表示其局部坐标框架的方向矩阵𝑂𝑖
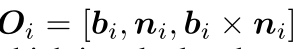
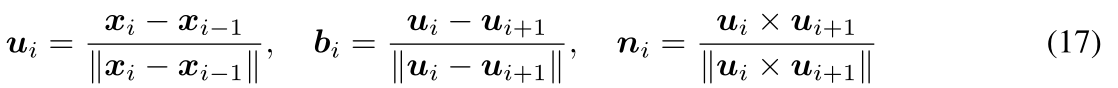
- 对应论文:John Ingraham, Vikas K Garg, Regina Barzilay, and Tommi Jaakkola. Generative models for graph-based protein design.Neural Information Processing Systems, 2019.
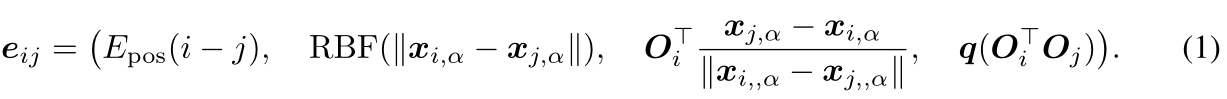
- 第一项:编码抗体序列中两个残基之间的相对距离
- 第二项:提升为径向基的距离编码
- 第三项:对应于残基𝑖的局部框架中的𝑥𝑗的相对方向的方向编码

- 第四项:空间旋转矩阵𝑂𝑖𝑇𝑂𝑗的四元数表示q(·)的定向编码

- 对于每一个残基,只考虑K=8个最近邻居
迭代改进的GNN:RefineGNN
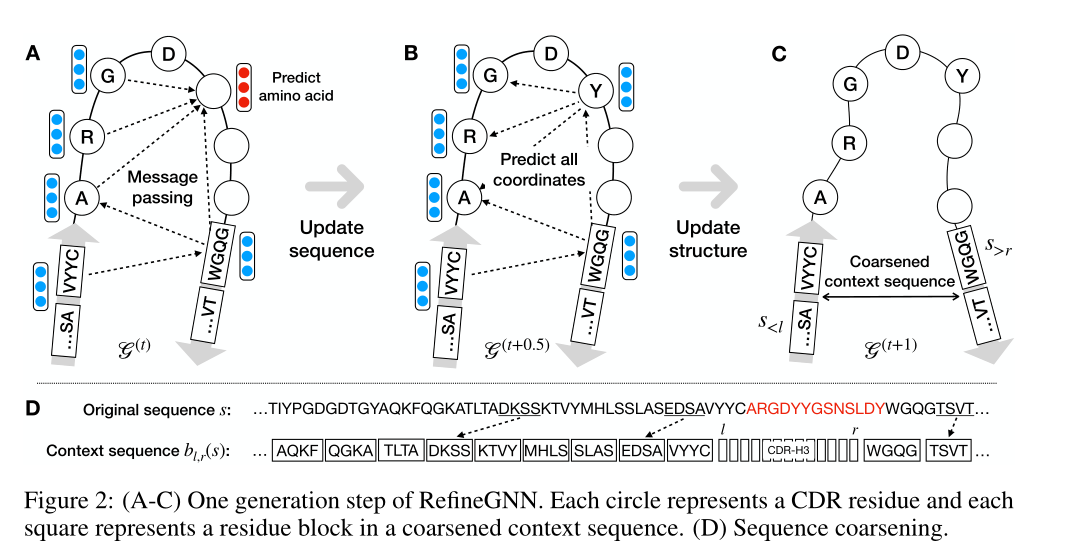
初始化
- 节点:全部初始化为<MASK>
- 边:每条边初始化距离为3|𝑖−𝑗|,因为连续残基之间的平均距离约为3。方向和方向特征设置为零

生成过程
- 在步骤t中,修改当前的图𝐺𝑡,然后预测下一个残基𝑡+1

- 修改步骤
- 使用消息传递框架MPN,h为每一个残基的表征
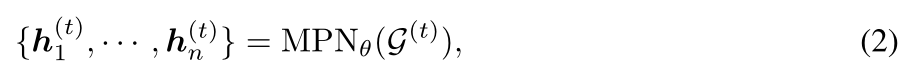
- 其中,MPN有L层,每一层都由一个两层的FFN(激活函数为ReLU)表示
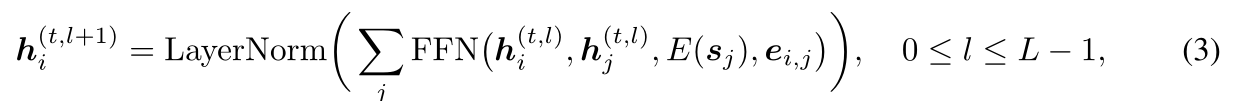
- 𝐸(𝑠𝑗)为学习到的氨基酸的表征

- 𝐸(𝑠𝑗)为学习到的氨基酸的表征

- 使用消息传递框架MPN,h为每一个残基的表征
- 预测步骤(上图中的A)

- 注意,预测步骤之后,图𝐺𝑡的边没有发生改变,但是节点特征变了(有一个<MASK>变为了预测出来的标签),因而这时的图为𝐺𝑡+0.5(上图中的B),需要进行结构上的修改(上图中的C):

- 可以发现,结构预测(公式5,6)和序列预测(公式2,4)是采用的不同的参数,进了解耦,使得两个网络可以专注于两个不同的任务
两种训练
- 离散氨基酸类型预测:第t步中,𝑠1~𝑠𝑡采用真实氨基酸类型, 之后的残基t+1~n为padding token

- 连续结构预测:在每次迭代中,该模型改进上一步预测的整体结构,基于预测坐标
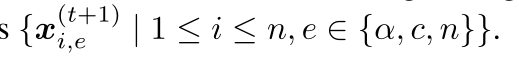
- 构建一个新的K-nearest邻居生成新图𝐺𝑡+1
损失函数
- 损失函数包含序列预测损失和结构损失两个方面
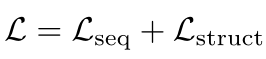
- 序列预测损失为交叉熵函数

- 结构损失包含三部分
- 距离损失:在预测与实际的阿尔法碳之间使用Huber loss
- 二面角损失(Dihedral angle loss):预测的二面角与真实的二面角之间的均方误差
- Cα angle loss
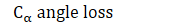
- 距离损失:在预测与实际的阿尔法碳之间使用Huber loss
给定框架区域的条件生成
- 上述的方法没有为抗体生成添加限制,而在实验中,通常需要固定抗体的框架区域,只设计CDR序列
- 学习条件分布
- 使用GRU为将CDR区域编码为上下文,然后预测中间被padding的CDR序列,使用注意力机制,将公式4,6修改为
- 单纯使用注意力不行,没有考虑到结构;而每次迭代都修改结构在计算上过于昂贵了;在一开始就固定所有要预测残基的坐标更不行,因为他们需要得到相应的调整--->提出了粗粒度的模型:对残基进行聚类分块(上图D)为b,对应块的属性:
属性导向的序列优化
- 最终目标是产生具有所需特性的新抗体,可以看成是一个优化问题
- 在ITA(iterative target augmentation)优化开始之前,首先在一组真实抗体结构上预先训练模型,以学习CDR序列和结构的先验分布
- 在每个ITA微调步骤中,首先随机抽取D的序列,D是一组抗体,其CDR需要重新设计

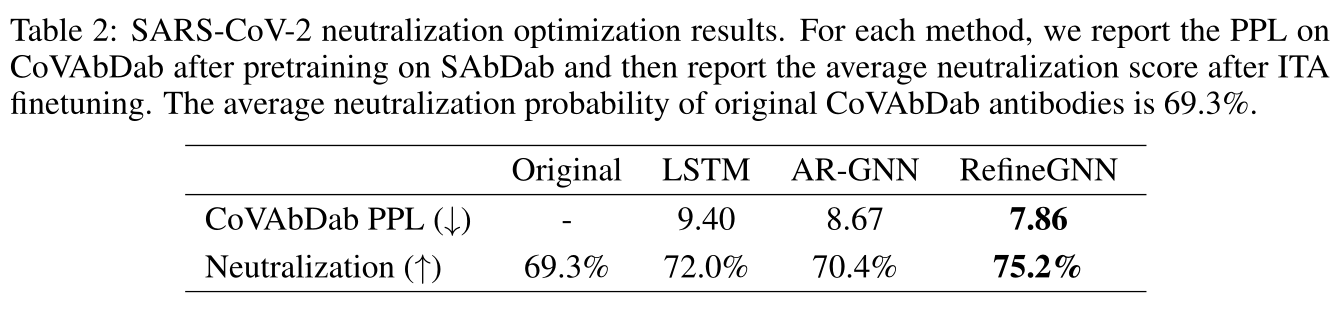
实验及结果
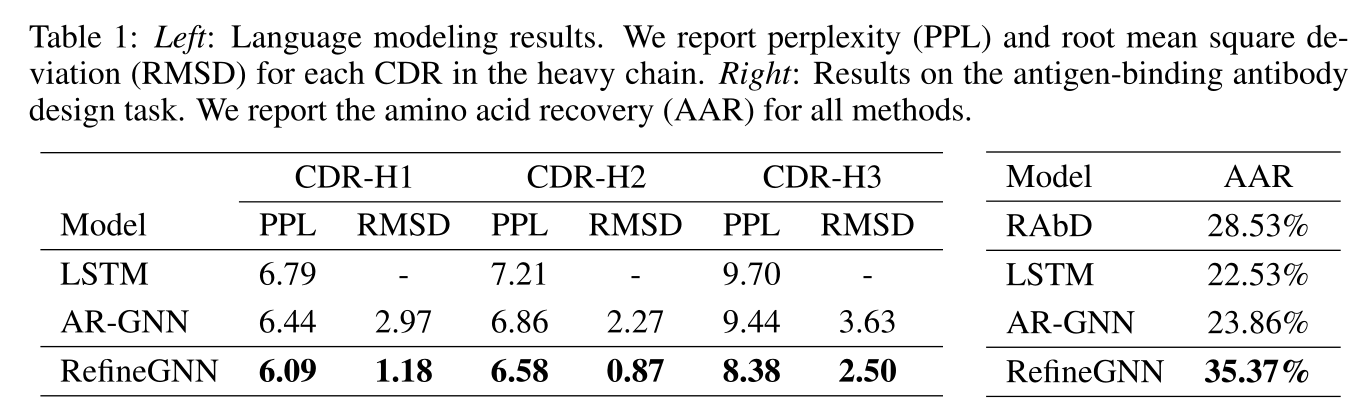
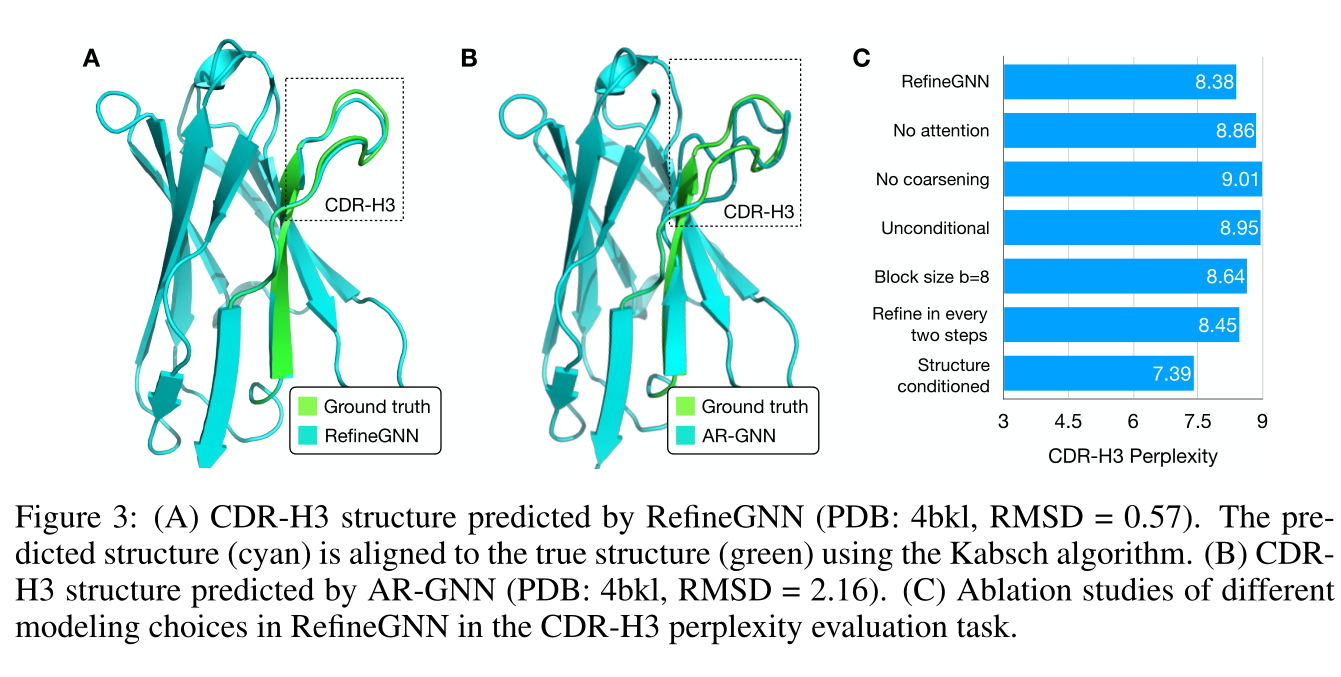
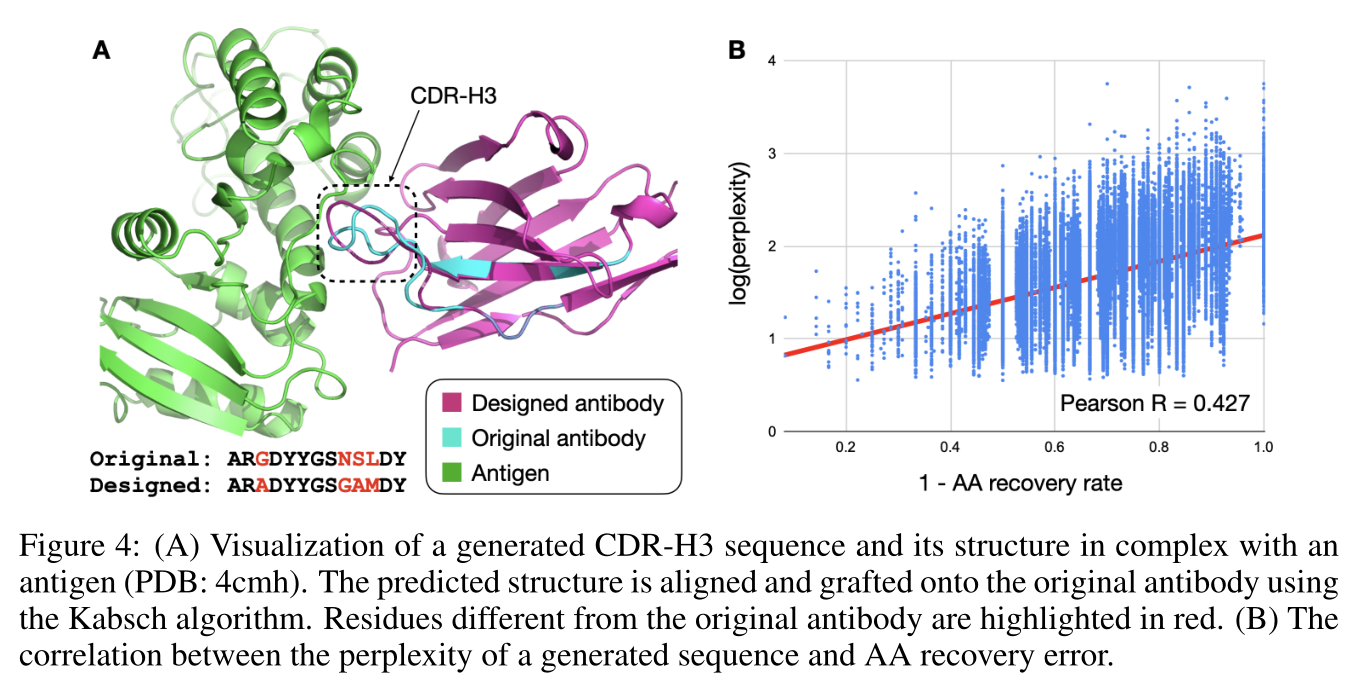
这篇关于生成抗体的生成GNN:Iterative Refinement Graph Neural Network for Antibody Sequence-Structure Co-design的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!