本文主要是介绍综述 2020-Bioinformatics and biology insights:多组学数据整合和研究方法,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
Subramanian, Indhupriya, et al. "Multi-omics data integration, interpretation, and its application." Bioinformatics and biology insights 14 (2020): 1177932219899051.
- 被引次数:746
- 期刊影响因子:5.97
- 多组学数据联合分析
一、组学数据类型
- 本文:基因组、蛋白质组、转录组、代谢组和表观基因组;脂质组、磷酸蛋白质组和乙二醇蛋白质组
- 之前看的综述里的组学类型:综述 2017-Genome Biology:疾病的多组学方法-CSDN博客

二、组学数据存储库
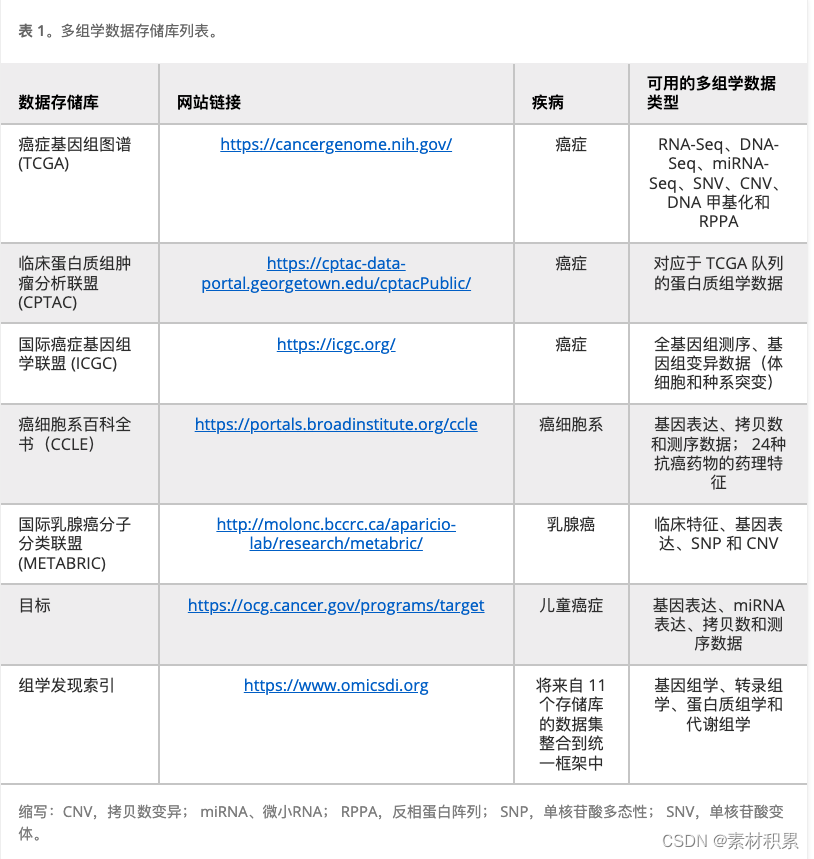
1. 癌症基因组图谱
癌症基因组图谱(TCGA;https://cancergenome.nih.gov/)是一个拥有超过33种不同类型癌症的20,000个个体肿瘤样本的多组学数据集之一。该倡议旨在生成、整合、分析和解释肿瘤样本的DNA、RNA、蛋白质和表观遗传学变化以及临床和组织学数据的概要。它包含来自各种癌症及其亚型的原发肿瘤样本的丰富分子和遗传概要。它生成高通量RNA-Seq、DNA-Seq、miRNA-Seq、单核苷酸变异(SNV)、拷贝数变异(CNV)、DNA甲基化和逆相蛋白质阵列(RPPA)数据。全癌图谱被研究社区广泛使用,有助于对癌症的进展、表现和治疗进行新的发现。TCGA的生物样本通过质谱技术进行分析,癌症队列的蛋白质组数据可在临床蛋白质瘤分析协会(CPTAC)(https://cptac-data-portal.georgetown.edu/cptacPublic/)上获得。
2. 国际癌症基因组协会
国际癌症基因组协会(ICGC;https://icgc.org/)协调了来自21个原发癌症部位的76个癌症项目的大规模基因组研究生成,包括来自20,383个捐赠者(截至2017年12月)。该项目主要包含来自各种族群的各种癌症类型的突变相关基因组改变数据(包括生殖系和体细胞突变)。该联盟为每种肿瘤类型定义目录,并确保生成的数据质量,并在研究社区之间管理数据共享。ICGC数据协调中心(DCC)运营ICGC数据门户,其中包含数据的公开和限制访问部分。16 ICGC门户已用于推导癌症生物学的里程碑观察。17,18全基因组的全癌症分析(PCAWG;https://dcc.icgc.org/pcawg)允许探索和分析来自ICGC的2800多个全基因组。
3. 癌细胞系百科全书
癌细胞系百科全书(CCLE;(https://portals.broadinstitute.org/ccle)由Broad研究所托管,汇编了来自947个人类细胞系和36种肿瘤类型的基因表达、拷贝数和测序数据。它还包含479个癌细胞系对24种抗癌药物的药理学资料。该项目使我们能够在不同癌细胞系中识别新的生物标志和药物反应的机制效应。19
4. 乳腺癌国际分子分类联盟
乳腺癌国际分子分类联盟(METABRIC;http://molonc.bccrc.ca/aparicio-lab/research/metabric/)是一个加拿大-英国的项目,包含从乳腺肿瘤中得到的临床特征、表达、单核苷酸多态性(SNP)和CNV数据。该项目旨在使用潜在的多组学分子标志将乳腺肿瘤进一步分类。这个数据库确定了乳腺癌的10个亚组和以前未描述的新药物靶点,因此将有助于设计乳腺癌的最佳治疗方案。20
5. TARGET
TARGET(https://ocg.cancer.gov/programs/target)类似于TCGA,由国家癌症研究所(https://www.cancer.gov/)推动,旨在确定推动儿童癌症的分子事件。21这些数据包含24种分子类型的癌症的临床信息、基因表达、miRNA表达、拷贝数和测序数据。该数据库旨在为评估小儿癌症中的基因组改变提供强有力的基础。21,22
6. OmicsDI
组学发现指数(OmicsDI;https://www.omicsdi.org/)包含来自11个存储库的数据集,具有共同的数据结构。这是一个开源平台,用于访问、发现和整合基因组学、转录组学、蛋白质组学和代谢组学数据集。它包含来自人类、模式生物和非模式生物的数据集。除了索引数据集外,OmicsDI还包括每个数据集的标准化和注释步骤,可进行集成。23
7. NCBI
8. GEO
除了这些专用于多组学的数据库外,国家生物技术信息中心(NCBI)基因表达数据库(GEO)存档了来自多个平台和阵列的多种测序数据,如基因组学和转录组学。
二、组学数据的工具和方法
1. 工具和方法

- 图 1。多组学数据集成工具概述。这些工具/方法根据其方法进行分组,并根据其应用程序进行颜色编码。
- FSMKL表示特征选择多核学习;
- JIVE,联合和个体变异解释;
- MCIA,多重协同惯量分析;
- MDI,多数据集集成;
- MFA,多因素分析;
- MOFA,多组学因子分析;
- NEMO,基于邻域的多组学聚类;
- PFA,模式融合分析;
- PMA,惩罚多元分析;
- sMBPLS,稀疏多块偏最小二乘法;
- SNF,相似网络融合;
- NMF,非负矩阵分解;
- BCC,贝叶斯共识聚类;
- PSDF,患者特定数据融合。
2. 对应生物问题
- 基于多组学概况的疾病亚型和分类;
- 预测各种应用的生物标志物,包括疾病的诊断和驱动基因;
- 获得对疾病生物学的见解。
3. 问题、方法、输入数据等
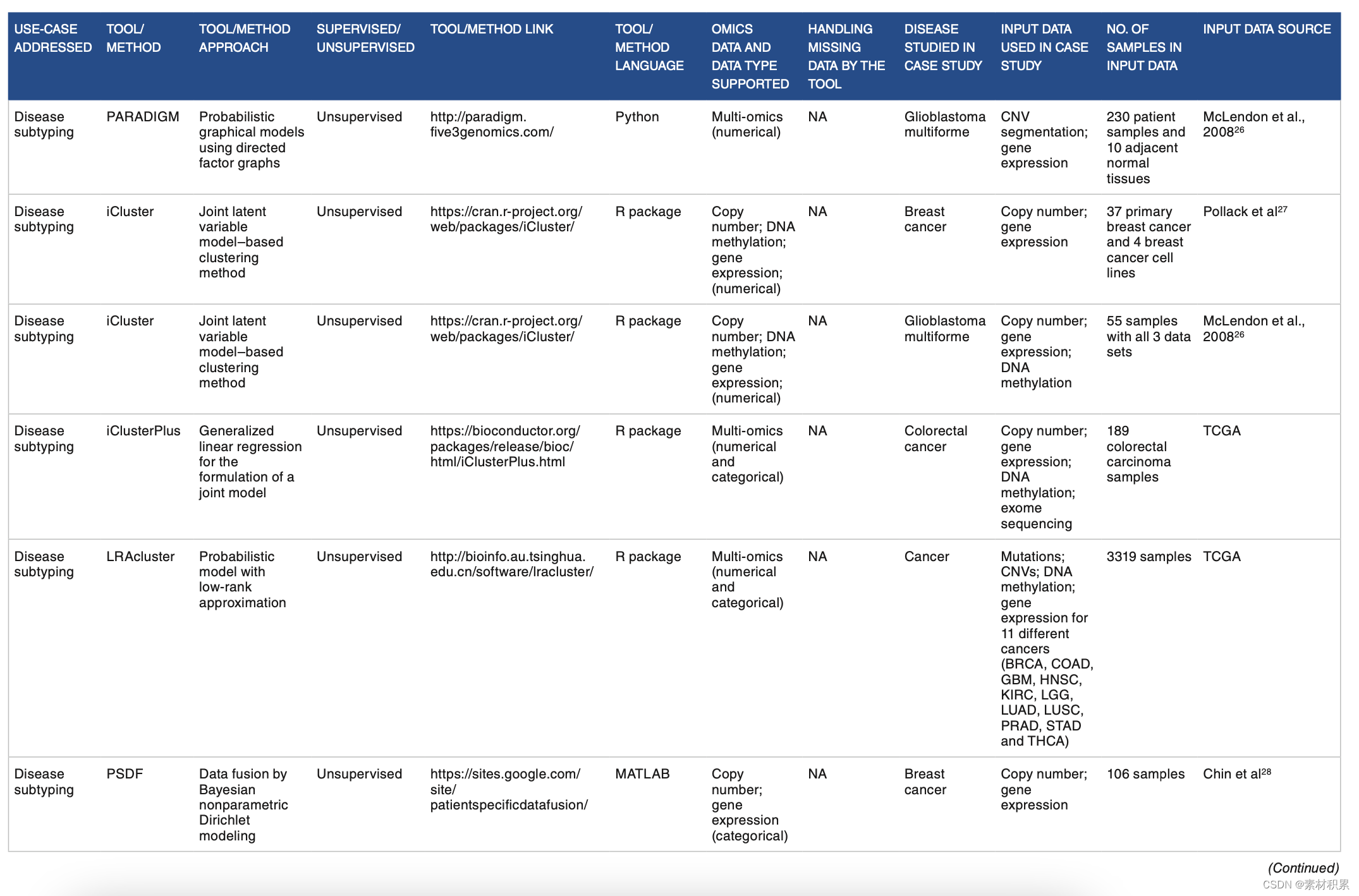
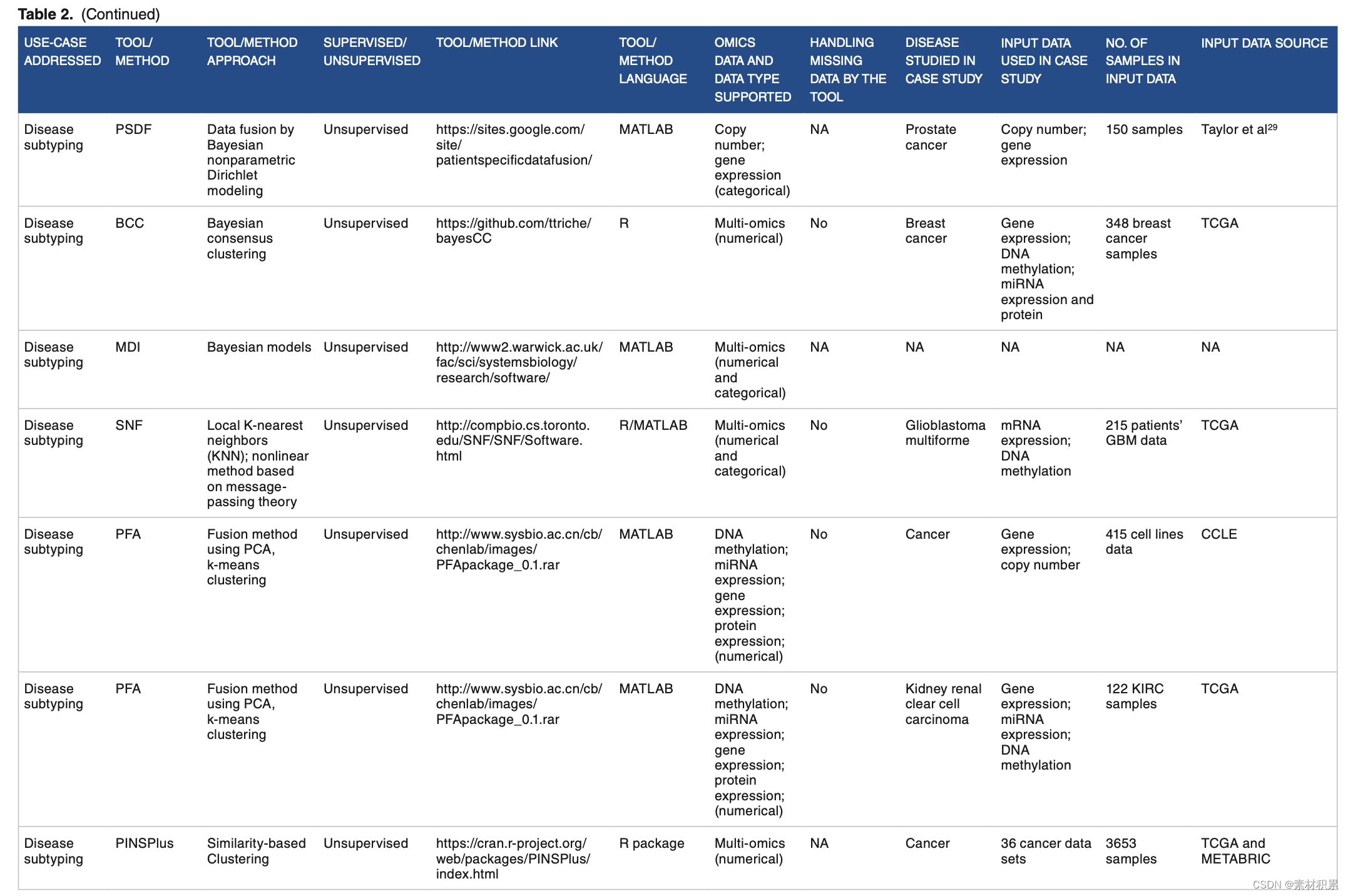
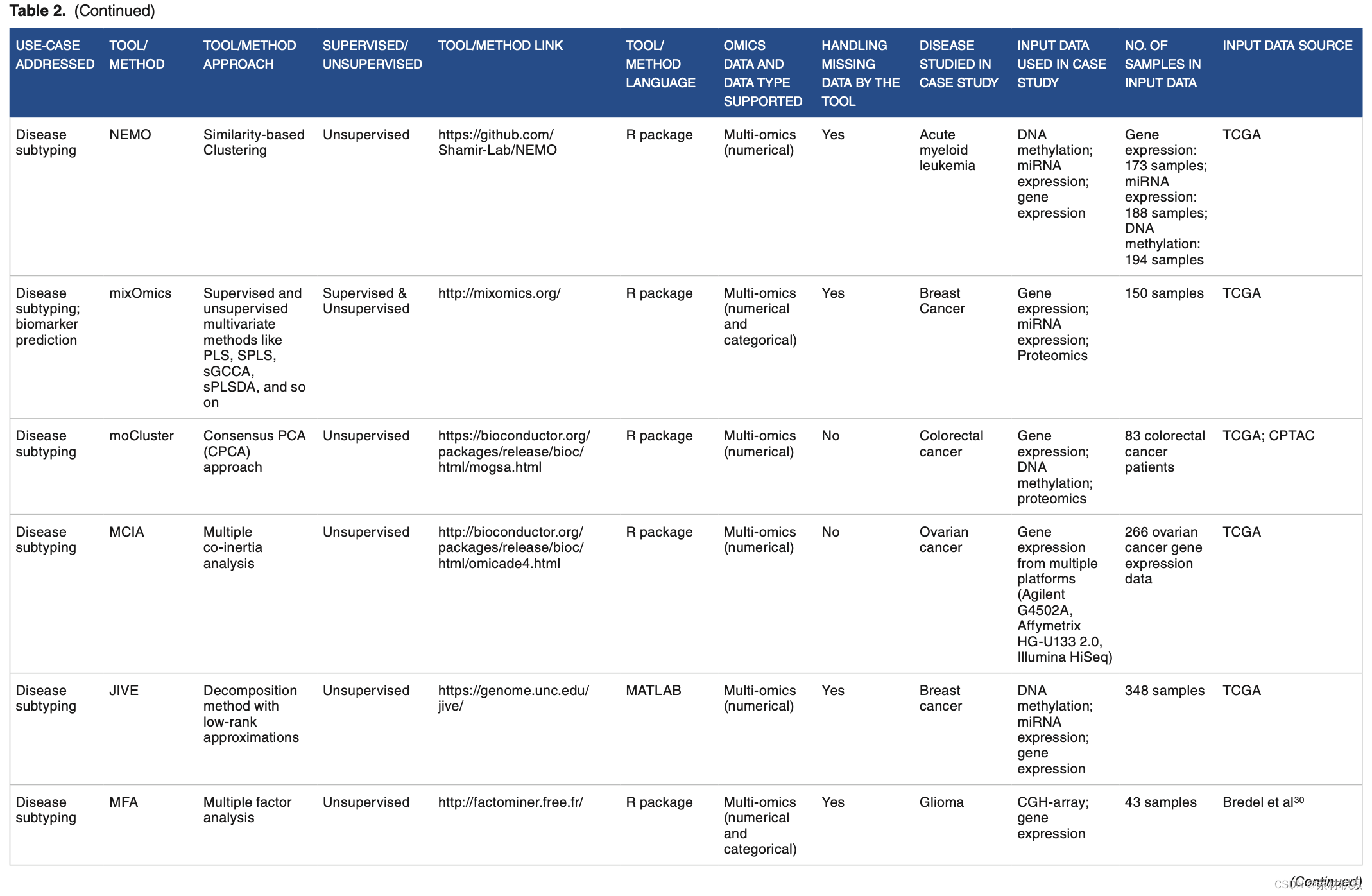
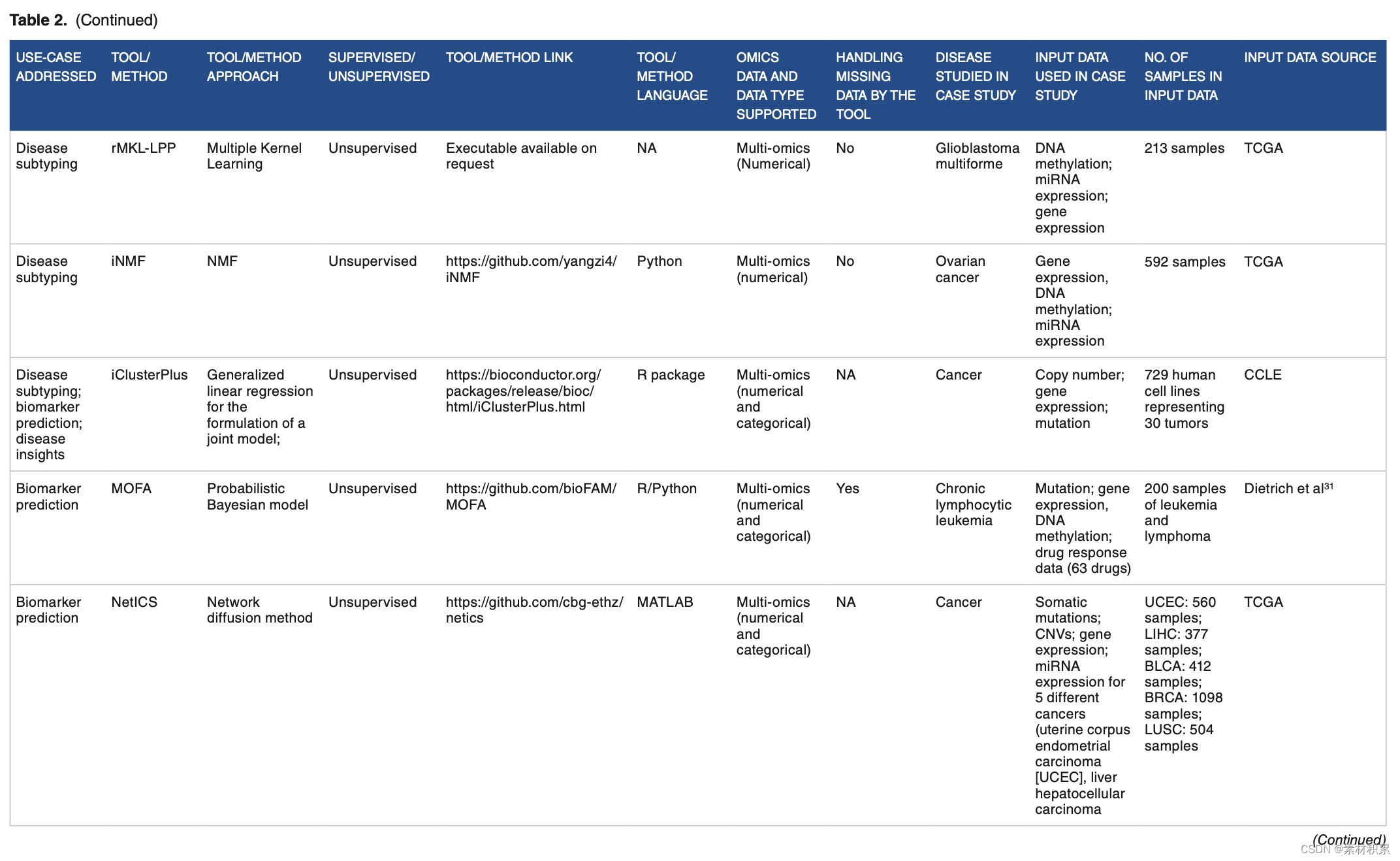

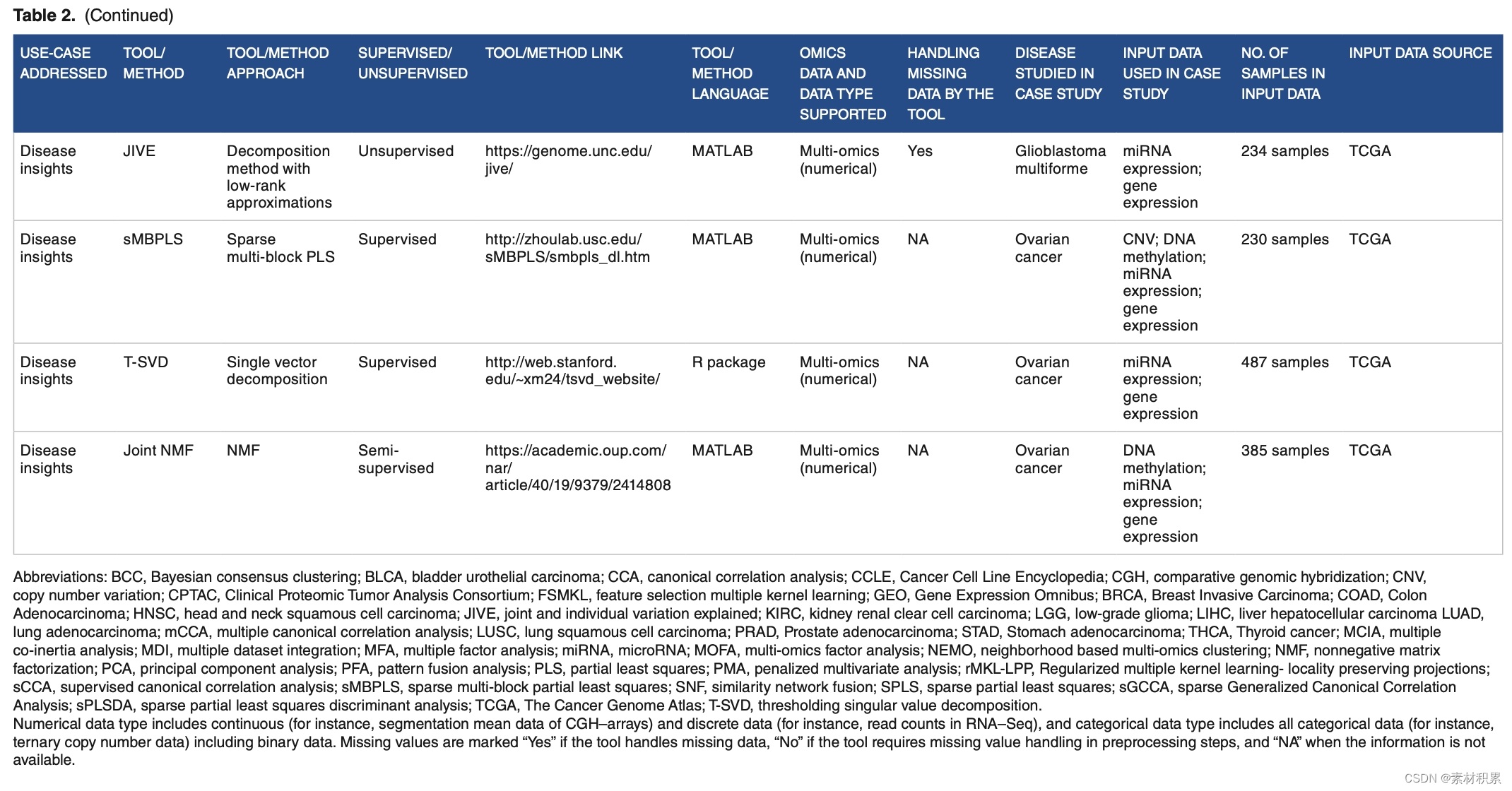
4. 分析和可视化门户
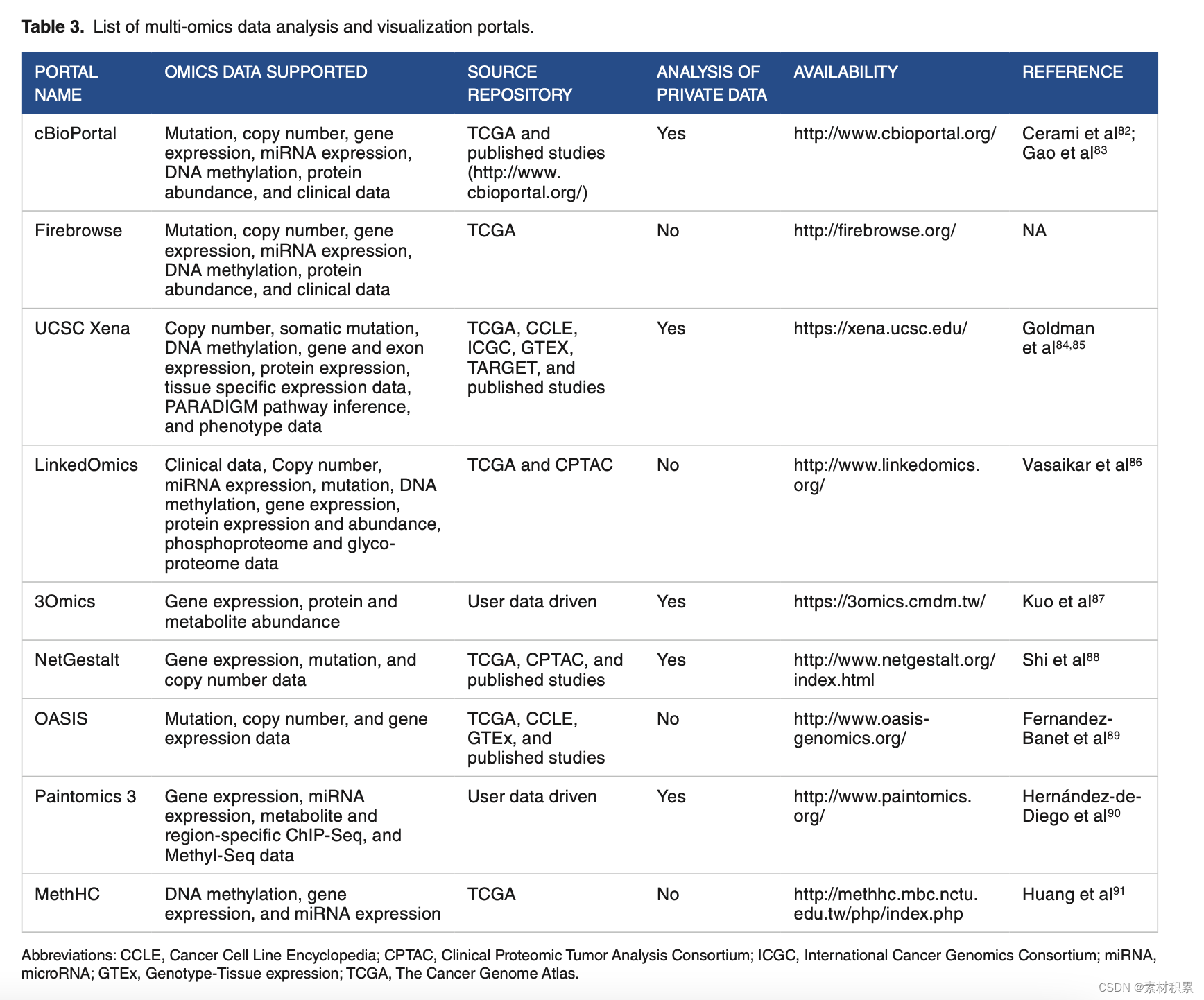
二、应用
1. 识别疾病亚型和样本分类
2. 通路识别
3. 生物标志物预测
4. 驱动基因检测
三、局限性和解决方法
- 一些组学异质性
- 数据规模大
- 缺乏计算密集型工具
- 多平台生成、数据存储格式差异大
- 预处理步骤复杂,缺乏通用标准:过滤、标准化、消除批次效应、质控等
解决方法:
- 预处理步骤可以进行特征选择
- 正确选择感兴趣的生物学问题,对集成工具进行基准测试
- 多组学数据解释可以增加临床信息
这篇关于综述 2020-Bioinformatics and biology insights:多组学数据整合和研究方法的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!




