本文主要是介绍PSP - 蛋白质-核酸复合物结构预测 RoseTTAFoldNA 算法框架 (Protein-RNA、Protein-DNA、RNA),希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
欢迎关注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://spike.blog.csdn.net/article/details/134208615
Paper: Accurate prediction of nucleic acid and protein-nucleic acid complexes using RoseTTAFoldNA
- GitHub: RoseTTAFold2NA
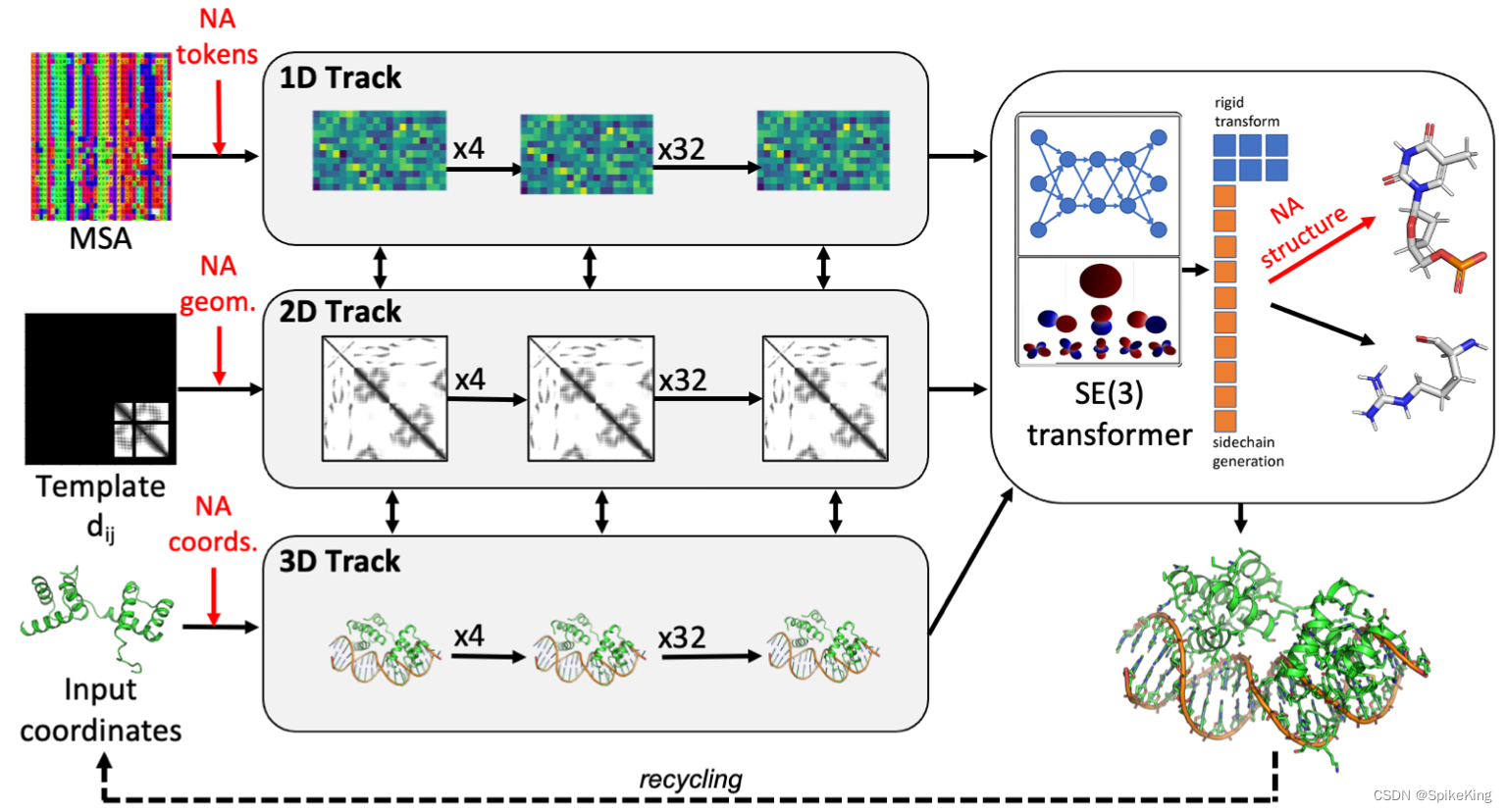
蛋白质-核酸复合物 (Protein - Nucleic Acid Complexes),在生物学中发挥着关键作用。 尽管蛋白质结构预测方面,最近取得了相当大的进展,但是,与已知复合物没有同源性的蛋白质-核酸复合物的结构预测,在很大程度上是一个尚未解决的问题。RoseTTAFold2NA 将 RoseTTAFold 端到端深度学习方法,扩展到核酸和蛋白质-核酸复合物的建模。 已训练的网络 RoseTTAFoldNA,可以快速生成 3D 结构模型,并且对 蛋白质-DNA 和 蛋白质-RNA 复合物以及 RNA 三级结构进行置信估计。 在这三种情况下,置信预测的准确性,都比当前最先进的方法高得多。 RoseTTAFoldNA 应广泛用于模拟天然存在的 蛋白质-核酸 复合物的结构,以及设计序列特异性 RNA 和 DNA 结合蛋白。
David Baker 组的工作,作者 Minkyung Baek
1. Docker 配置
Docker 环境:
# 下载 glm-nvidia-pytorch-1.11.0-cu116-py3.tar
docker image load -i glm-nvidia-pytorch-1.11.0-cu116-py3.tarnvidia-docker run -it --name rfna-v1 --shm-size 72G -v [nfs]/[nfs] glm:nvidia-pytorch-1.11.0-cu116-py3
glm:nvidia-pytorch-1.11.0-cu116-py3,比较稳定的 docker image ,建议作为 base image。
准备 GitHub 工程:
git clone https://github.com/uw-ipd/RoseTTAFold2NA.git
cd RoseTTAFold2NAcd /nfs_beijing_ai/chenlong/workspace/rosettafold2na-by-chenlong
GitHub 加速代理:
https://gitclone.com/
https://ghproxy.com/
https://toolwa.com/github/
配置 Docker 环境中的 conda 源,即:
vim ~/.condarc
conda 清华源,添加:
channels:- defaults
show_channel_urls: true
default_channels:- https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main- https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/r- https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/msys2- https://mirrors.sustech.edu.cn/anaconda/pkgs/free- https://mirrors.sustech.edu.cn/anaconda/pkgs/pro
custom_channels:conda-forge: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloudmsys2: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloudbioconda: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloudmenpo: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloudpytorch: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloudsimpleitk: https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloudnvidia: https://mirrors.sustech.edu.cn/anaconda-extra/cloud
channel_priority: disabled
allow_conda_downgrades: true
conda 南方科技源 (速度很慢,建议与清华源合并使用),添加:
channels:- defaults
show_channel_urls: true
default_channels:- https://mirrors.sustech.edu.cn/anaconda/pkgs/main- https://mirrors.sustech.edu.cn/anaconda/pkgs/free- https://mirrors.sustech.edu.cn/anaconda/pkgs/r- https://mirrors.sustech.edu.cn/anaconda/pkgs/pro- https://mirrors.sustech.edu.cn/anaconda/pkgs/msys2
custom_channels:conda-forge: https://mirrors.sustech.edu.cn/anaconda/cloudmsys2: https://mirrors.sustech.edu.cn/anaconda/cloudbioconda: https://mirrors.sustech.edu.cn/anaconda/cloudmenpo: https://mirrors.sustech.edu.cn/anaconda/cloudpytorch: https://mirrors.sustech.edu.cn/anaconda/cloudsimpleitk: https://mirrors.sustech.edu.cn/anaconda/cloudnvidia: https://mirrors.sustech.edu.cn/anaconda-extra/cloud
channel_priority: disabled
allow_conda_downgrades: true
显示全部 channels:
conda config --show
配置 pip 源:
rm /opt/conda/pip.conf
rm /root/.config/pip/pip.conf
vim ~/.pip/pip.conf[global]
no-cache-dir = true
index-url = http://mirrors.aliyun.com/pypi/simple/
extra-index-url = https://pypi.ngc.nvidia.com
trusted-host = mirrors.aliyun.com pypi.ngc.nvidia.com
2. 安装 Python 库
使用 yaml 安装,适合网络较好。或逐步安装 python 库,解决问题。
2.1 yaml 安装
安装 conda 环境,需要耐心等待:
conda env create -f RF2na-linux.yml
其中 RF2na-linux.yml,如下:
name: RF2NA
channels:- pytorch- nvidia- defaults- conda-forge
dependencies:- python=3.10- pip- pytorch- requests- pytorch-cuda=11.7- dglteam/label/cu117::dgl- pyg::pyg- bioconda::mafft- bioconda::hhsuite- bioconda::blast- bioconda::hmmer>=3.3- bioconda::infernal- bioconda::cd-hit- bioconda::csblast- pip:- psutil- tqdm
2.2 逐步安装 (速度较快)
单步创建环境,注意安装顺序:
ource ~/.bashrc
conda env list
conda create -y -n RF2NA python=3.10
conda activate RF2NAconda install pip requests scipy
pip install psutil tqdm# 优先安装 conda 的 python 库
conda install -y hmmer hhsuite==3.3.0 -c bioconda
conda install -y mafft blast infernal cd-hit csblast -c bioconda
# conda install dgl-cuda11.7 -c dglteam # 可选# pip install torch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2
# nvidia 下载较慢,建议使用 pip 安装 pytorch
conda install pytorch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2 pytorch-cuda=11.7 -c pytorch -c nvidia --show-channel-urls
conda install pyg -c pyg
2.2.1 hhsuite 与 环境冲突
遇到冲突错误,即遇到 hhsuite==3.3.0 与环境冲突,无法安装,解决方法是使用不同的 docker image 即可,如下:
UnsatisfiableError: The following specifications were found to be incompatible with each other
建议使用 glm:nvidia-pytorch-1.11.0-cu116-py3 作为 docker 的 Base Image。
2.2.2 安装 dglteam (可选)
(可选) 安装 dgl 较慢,建议下载,再配置安装 dglteam,如下:
# dgl-cuda11.7 dglteam/linux-64::dgl-cuda11.7-0.9.1post1-py310_0
# dgl-cuda11.7-0.9.1post1-py310_0.tar.bz2, 约 220 M
# 下载链接:https://conda.anaconda.org/dglteam/linux-64
conda install ./dgl-cuda11.7-0.9.1post1-py310_0.tar.bz2
2.2.3 安装 PyTorch
使用 conda 安装 pytorch 较慢时,建议使用 pip 进行安装,速度较快。测试 PyTorch 是否可用:
conda install scipy
pip install psutil==5.8.0 tqdm
pip install torch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2
即:
pythonimport torch
print(torch.__version__) # 2.0.1+cu117
print(torch.cuda.is_available()) # True
2.2.4 安装 PyG
安装 pyg 包,也可以使用 pip 进行安装,参考 pytorch-geometric - Installation,即
pip install torch_geometric# Optional dependencies:
pip install pyg_lib torch_scatter torch_sparse torch_cluster torch_spline_conv -f https://data.pyg.org/whl/torch-2.0.0+cu117.html
3. 测试
数据位置:/nfs_beijing_ai/bj-common-data/ods/uw-ipd/RoseTTAFold2NA/RNA/
建立蛋白质序列库的软链接,建议使用之前的下载:
ln -s [your folder]/uniref30 uniref30
ln -s [your folder]/bfd bfd
ln -s [your folder]/pdb100_2021Mar03 pdb100_2021Mar03
RNA 库安装:
# Rfam
cp [your folder]/RoseTTAFold2NA/RNA/Rfam/Rfam.cm.gz .
cp [your folder]/RoseTTAFold2NA/RNA/Rfam/Rfam.full_region.gz .
gunzip Rfam.cm.gz
gunzip Rfam.full_region.gz
cmpress Rfam.cm# RNAcentral
cp [your folder]/RoseTTAFold2NA/RNA/RNAcentral/rfam_annotations.tsv.gz .
cp [your folder]/RoseTTAFold2NA/RNA/RNAcentral/id_mapping.tsv.gz .
cp [your folder]/RoseTTAFold2NA/RNA/RNAcentral/rnacentral_species_specific_ids.fasta.gz .
../input_prep/reprocess_rnac.pl id_mapping.tsv.gz rfam_annotations.tsv.gz
gunzip -c rnacentral_species_specific_ids.fasta.gz | makeblastdb -in - -dbtype nucl -parse_seqids -out rnacentral.fasta -title "RNACentral"# nt
ln [your folder]/RoseTTAFold2NA/RNA/nt nt
模型权重:
cp [your folder]/RoseTTAFold2NA/RNA/network/RF2NA_apr23.tgz .
tar xvfz RF2NA_apr23.tgz
ll -h weights/
测试命令:
bash ../run_RF2NA.sh rna_pred rna_binding_protein.fa R:RNA.fa
# bash run_RF2NA.sh example/rna_pred example/rna_binding_protein.fa R:example/RNA.fa
执行步骤如下:
- 第1步执行:Running HHblits
- 第2步执行:Running hhsearch
- 第3步执行:Creating joint Protein/RNA MSA
- 第4步执行:Running RoseTTAFold2NA to predict structures
预测日志如下:
Running on GPUplddt best
RECYCLE 0 0.799 -1.000
RECYCLE 1 0.858 0.799
RECYCLE 2 0.870 0.858
RECYCLE 3 0.875 0.870
RECYCLE 4 0.877 0.875
RECYCLE 5 0.879 0.877
RECYCLE 6 0.877 0.879
RECYCLE 7 0.880 0.879
RECYCLE 8 0.879 0.880
RECYCLE 9 0.881 0.880
Done
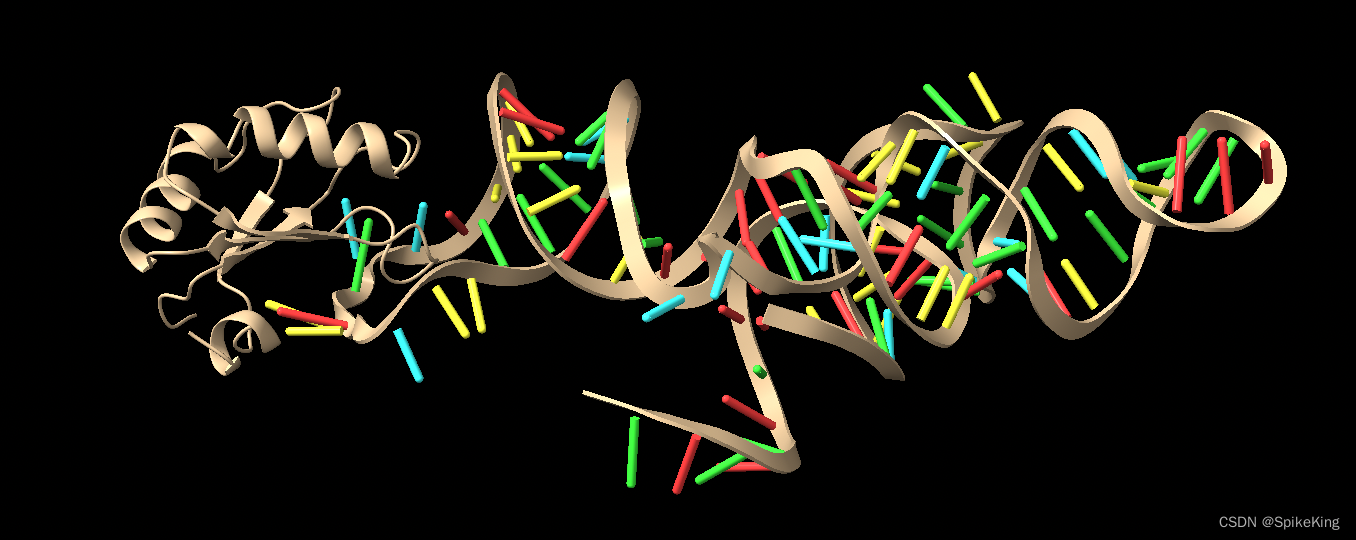
输出效果,包括 蛋白质部分与 RNA 部分,如下:
这篇关于PSP - 蛋白质-核酸复合物结构预测 RoseTTAFoldNA 算法框架 (Protein-RNA、Protein-DNA、RNA)的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!




