dna专题
OBItools:Linux下的DNA条形码分析神器
在生物信息学领域,DNA条形码分析是一种非常常见的研究方法,用于物种鉴定、生态学和进化生物学研究。今天要介绍的工具就是专为此设计的——OBItools。这个工具集专门用于处理生态学和进化生物学中的DNA条形码数据,在Linux环境下运行。无论你是本科生还是刚入门的科研人员,OBItools都能为你提供可靠的帮助。 OBItools的功能亮点 OBItools是一个强大的工具包,特别适合DNA条形
生物信息学:DNA序列的构成
DNA序列是由一串字母表示的真实的或者假设的携带基因信息的DNA分子的一级结构。 DNA序列的构成基于四种特定的碱基,分别是腺嘌呤(A)、胸腺嘧啶(T)、鸟嘌呤(G)和胞嘧啶(C)。这些碱基以特定的配对方式形成碱基对,即A与T配对,C与G配对,这是基于它们之间的氢键相互作用。每个碱基代表一个特定的遗传信息,通过这些碱基的排列顺序,DNA序列能够编码遗传信息,进而指导生物体的生长、发育和功能。
深入CSS字体的DNA:@font-face规则全解析
标题:深入CSS字体的DNA:@font-face规则全解析 CSS的@font-face规则是实现自定义字体功能的核心,它允许网页设计师使用几乎任何字体来增强网页的视觉表现力。本文将详细解释@font-face规则的工作原理,并通过代码示例展示如何在网页设计中应用这一强大的CSS特性。 1. @font-face规则简介 @font-face规则允许你定义自己的字体样式和字体来源,通过指定
重复的DNA序列(LeetCdoe)
题目 DNA序列 由一系列核苷酸组成,缩写为 'A', 'C', 'G' 和 'T'.。 例如,"ACGAATTCCG" 是一个 DNA序列 。 在研究 DNA 时,识别 DNA 中的重复序列非常有用。 给定一个表示 DNA序列 的字符串 s ,返回所有在 DNA 分子中出现不止一次的 长度为 10 的序列(子字符串)。你可以按 任意顺序 返回答案。 解题 """时间复杂度: O(n
POJ 3691 HDU 2457 DNA repair (AC自动机,DP)
http://poj.org/problem?id=3691 http://acm.hdu.edu.cn/showproblem.php?pid=2457 DNA repair Time Limit: 2000MS Memory Limit: 65536KTotal Submissions: 5690 Accepted: 2669 Description Biologists
LeetCode 187-重复的DNA序列
187. 重复的DNA序列 DNA序列 由一系列核苷酸组成,缩写为 ‘A’, ‘C’, ‘G’ 和 ‘T’.。 例如,“ACGAATTCCG” 是一个 DNA序列 。 在研究 DNA 时,识别 DNA 中的重复序列非常有用。 给定一个表示 DNA序列 的字符串 s ,返回所有在 DNA 分子中出现不止一次的 长度为 10 的序列(子字符串)。你可以按 任意顺序 返回答案。 示例 1: 输
Repeated DNA Sequences问题及解法
问题描述: All DNA is composed of a series of nucleotides abbreviated as A, C, G, and T, for example: "ACGAATTCCG". When studying DNA, it is sometimes useful to identify repeated sequences within the DNA.
POJ 1007 DNA 排序
题意:分类DNA字符串(只有ACGT四个字符)。但是分类它们的方法不是字典序,而是逆序数,排序程度从好到差。所有字符串长度相同。 解题思路:第一感觉就是用结构体数组,结构体中存字符数组和这个字符数组的逆序数。然后用两个for循环求逆序数即可。刚开始编完提交WA,仔细看题目才记得是稳定排序,所以把sort改为stable_sort即可实现稳定排序。编写一个判断的函数使逆序数从小到大排序,最后从头到
易基因:人类精子发生过程中的全基因组DNA甲基化水平变化|研究速递
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。 精子发生和精子功能需要在生殖细胞系中正确建立DNA甲基化模式。 德国明斯特大学生殖与再生生物学研究所生殖医学中心Sandra Laurentino团队分析了人类精子发生(spermatogenesis)过程中的全基因组DNA甲基化变化以及在精子发生障碍时的变化。分析结果表明精子发生与甲基化重塑有关,包括初级精母细胞中DNA甲基化的
基于动态规划算法的DNA序列比对函数,给出两条序列(v和w)的打分矩阵
一.什么是动态规划算法 1.1总体思想 ·动态规划算法与分治法类似,基本思想也是将待求解的问题分成若干个子问题 ·经过分解得到的子问题往往不是互相独立的,有些子问题被重复计算多次 ·如果能够保存已解决的子问题答案,在需要时再找出来已求得的答案,就可以避免大量重复计算,从而得到多项式时间算法(备忘录) 1.2使用动态规划求解的问题需要具备的基本要素 1)重复子问题 ·递归算法求解问题时
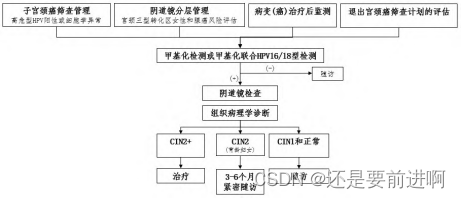
文章分享:《肿瘤DNA甲基化标志物检测及临床应用专家共识(2024版)》
本文摘自于《肿瘤DNA甲基化标志物检测及临床应用专家共识(2024版)》 目录 1. DNA甲基化标志物概述 2 DNA甲基化标志物的临床检测 2.1 临床样本前处理注意事项 2.2 DNA甲基化标志物检测技术方法 2.2.1 DNA提取与纯化 2.2.2 DNA转化 2.2.3 DNA 甲基化检测平台 3 DNA甲基化标志物用于肿瘤筛查 3.1 DNA
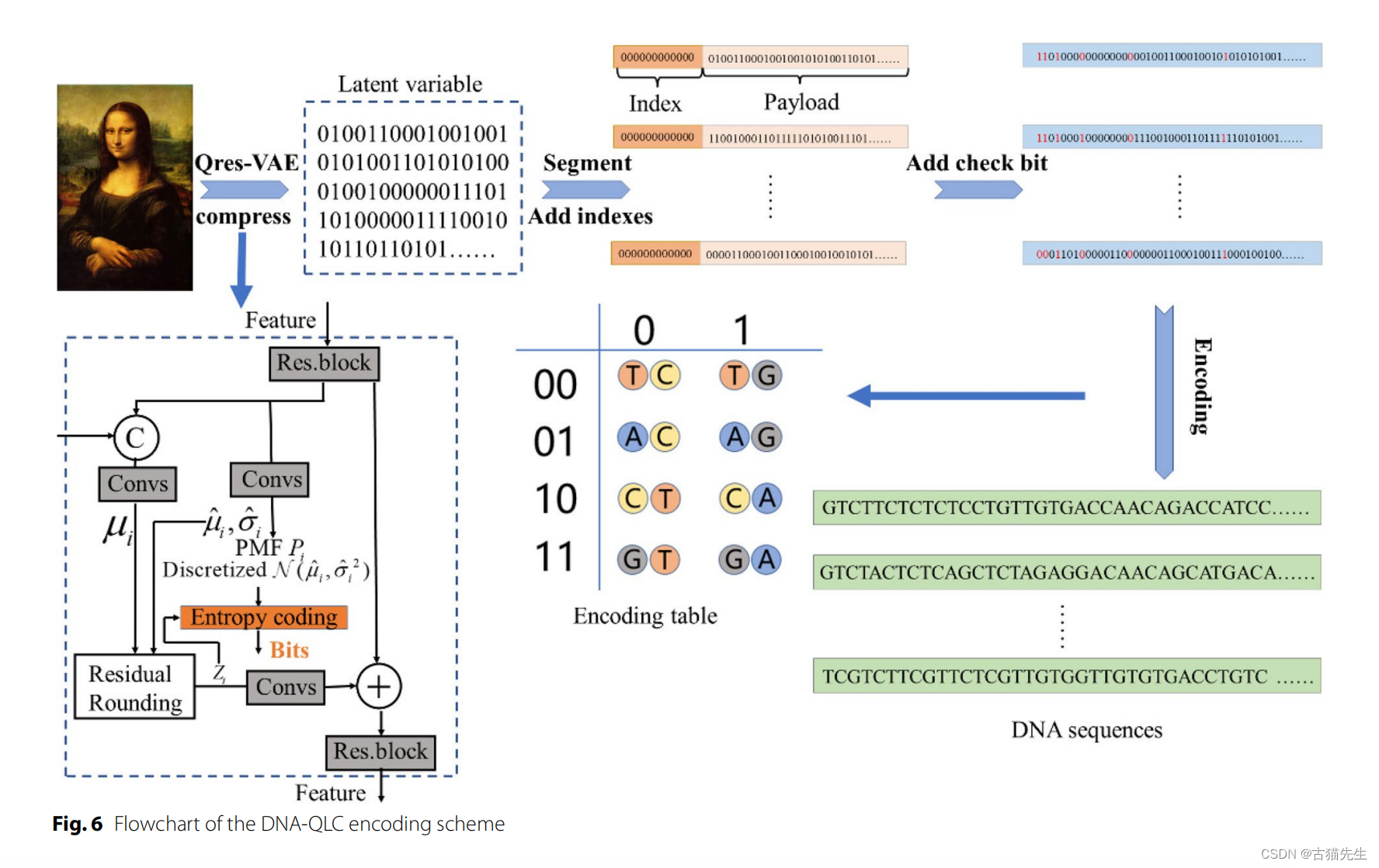
革新DNA存储:DNA-QLC编码方案高效可靠,多媒体图像存储新时代启航
在数字信息爆炸的时代,传统存储介质正面临容量、持久性和能耗的极限挑战。为此,大连理工大学计算机科学与技术学院的研究团队在《BMC基因组学》发表了一篇开创性论文,介绍了一种名为DNA-QLC的创新编码方案,为DNA存储系统的高效性和可靠性设立了新标准,极大地推进了DNA存储技术在图像等多媒体信息领域的应用潜力。 ### 背景与挑战 尽管DNA存储因巨大存储容量、长期稳定性和低能耗等优势被誉为
力扣:187. 重复的DNA序列(Java)
目录 题目描述:输入:输出:代码实现: 题目描述: DNA序列 由一系列核苷酸组成,缩写为 ‘A’, ‘C’, ‘G’ 和 ‘T’.。 例如,“ACGAATTCCG” 是一个 DNA序列 。 在研究 DNA 时,识别 DNA 中的重复序列非常有用。 给定一个表示 DNA序列 的字符串 s ,返回所有在 DNA 分子中出现不止一次的 长度为 10 的序列(子字符串)。你可以按 任
DNA序列k-mers哈希映射和相似序列查找
对DNA序列的k-mer进行哈希映射和相似序列查找是生物信息学中常见的任务之一。使用哈希函数对DNA序列的k-mer进行映射,并使用哈希表进行相似序列的查找。这种方法可以加速相似序列的搜索,并在处理大规模DNA序列数据时具有较好的性能。 哈希函数是一种将输入数据映射到固定长度的输出数据的函数。它的主要特点是对于给定的输入,能够产生唯一的输出,称为哈希值或散列值。哈希函数常用于密码学、数据完整性检
187. hash 重复的DNA序列
187. 重复的DNA序列 所有 DNA 都由一系列缩写为 A,C,G 和 T 的核苷酸组成,例如:“ACGAATTCCG”。在研究 DNA 时,识别 DNA 中的重复序列有时会对研究非常有帮助。 编写一个函数来查找 DNA 分子中所有出现超过一次的 10 个字母长的序列(子串)。 示例: 输入:s = "AAAAACCCCCAAAAACCCCCCAAAAAGGGTTT"输出:["AAA
【算法分析与设计】重复的DNA
📝个人主页:五敷有你 🔥系列专栏:算法分析与设计 ⛺️稳中求进,晒太阳 题目 DNA序列 由一系列核苷酸组成,缩写为 'A', 'C', 'G' 和 'T'.。 例如,"ACGAATTCCG" 是一个 DNA序列 。 在研究 DNA 时,识别 DNA 中的重复序列非常有用。 给定一个表示 DNA序列 的字符串 s ,返回所有在 DNA 分子中出现不
c++计算DNA探针的熔解温度
DNA探针的熔解温度(Tm)是指DNA双链在解离过程中的温度,可以用来估计DNA探针与靶序列的结合强度。 DNA探针富集实验中使用的盐浓度通常是在高盐条件下进行的,以帮助DNA与探针结合并提高富集效率。一般来说,盐浓度在0.5 M到1 M之间是常见的范围,但具体的盐浓度会根据实验的具体条件和目的而有所不同。例如,常用的盐包括NaCl和NaI。 DNA探针富集实验中常用的二价阳离子是Mg^2+。
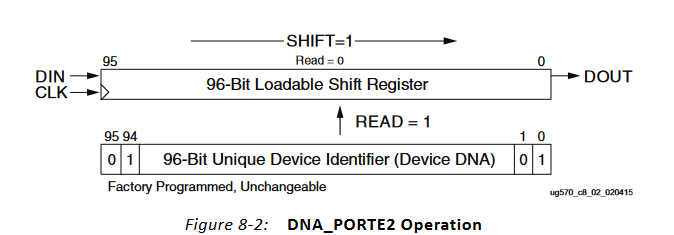
如何获取FPGA的device DNA?(含源码)
目录 一、什么是FPGA的Device DNA? 二、为什么需要获取FPGA的Device DNA? 1、验证真伪 2、追踪管理 3、技术支持 4、个性化配置 5、用户逻辑加密 三、如何获取FPGA的Device DNA? 1、通过Hardware Manager获取 2、通过verilog代码获取 四、获取Device DNA的注意事项 每个FPGA芯片都有其独特
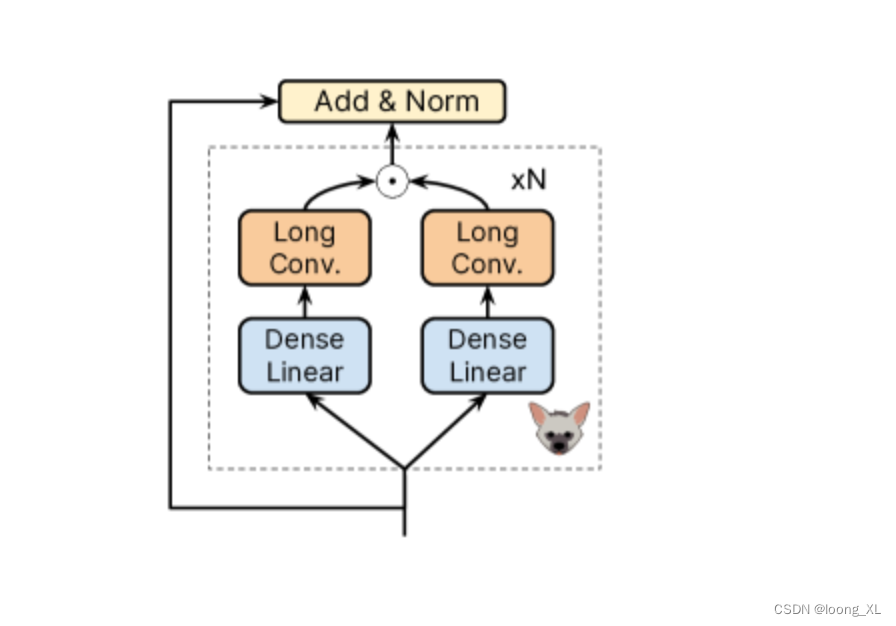
LLM生成模型在生物基因DNA应用:HyenaDNA
参考: https://github.com/HazyResearch/hyena-dna 整体框架基本就是GPT模型架构 不一样的就是𝖧𝗒𝖾𝗇𝖺𝖣𝖭𝖠 block ,主要是GPT的多重自注意力层引入了cnn 特征向量提取 # python huggingface.py #@title Single exampleimport jsonimport osim
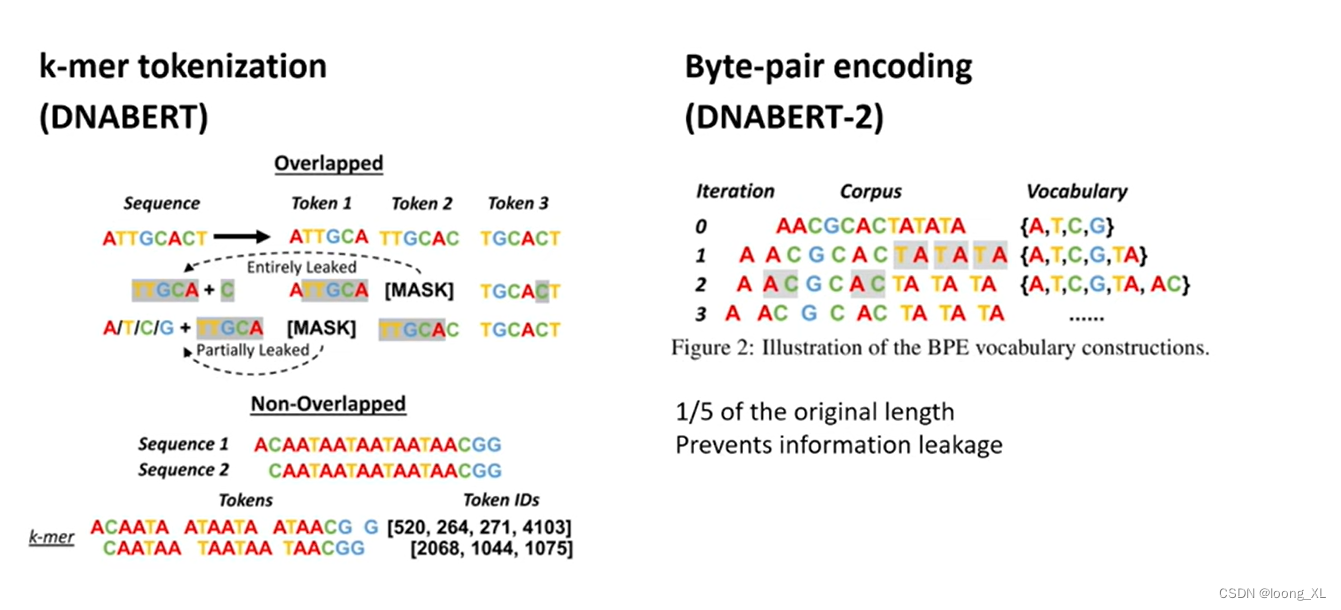
transformer在生物基因DNA的应用:DNABERT、DNABERT-2
参考: https://www.youtube.com/watch?v=mk-Se29QPBA&t=1388s 写明这些训练模型可以最终训练好可以进行DNA特征向量的提取,应用与后续 1、DNABERT https://github.com/jerryji1993/DNABERT 主要思路就是把DNA序列当成连续文本数据,直接用成熟的自然语言训练模型transformer进行生物D
Nature和Science同时报道,新疆出土四千年前遗骸完成DNA测序,证实并非移民而是土著...
来源:大数据文摘本文约1700字,建议阅读5分钟 这些埋藏在塔里木盆地上千年的遗骸,究竟来自哪里? 一个关于中国塔里木盆出土的数百具自然保存的遗骸考古研究,同时登上了著名学术期刊《自然》和《科学》的官网首页。 《自然》官网更是把这一研究放在了新闻头条上。 这一研究解决了困扰考古学界很长时间的问题,这些埋藏在塔里木盆地上千年的遗骸,究竟来自哪里? 一些科学家认为他们是来自俄罗斯黑海地区草原
MIT设计深度学习框架登Nature封面,预测非编码区DNA突变
来源:机器之心本文约2300字,建议阅读9分钟本文介绍了MIT和哈佛大学博德研究所等机构的最新研究。 来自 MIT 和哈佛大学博德研究所等机构的一项研究刚刚登上了 Nature 封面。他们创建了一个数学框架来预测基因组中非编码序列的突变及其对基因表达的影响。研究人员将能够利用这些模型来设计细胞、研发新药、寻找包括癌症和自身免疫性疾病在内的疾病新疗法。 尽管每个人类细胞都包含大量基因,但所谓
Windows DNA应用程序数据访问组件的强度测试
http://www.microsoft.com/china/MSDN/library/archives/others/vStudio/DNAstress.asp Windows DNA应用程序数据访问组件的强度测试 Mike Schelstrate 摘要:本文论述了对Microsoft Windows DNA应用程序的数据访问组件进行强度测试的重要性,以及如何更加简便地执行测试过程 目录
习题 3-7 DNA序列(DNA Consesus String) UVa 1368
题目大意: 给m个长度为n的DNA序列,求一个DNA序列到所有序列的总汉明距离最小,汉明距离为不同字符的位置的个数。如果有多个满足题意得序列则取字典序最小的那个。 Input 2 5 8 TATGATAC TAAGCTAC AAAGATCC TGAGATAC TAAGATGT 4 10 ACGTACGTAC CCGTACGTAG GCGTACGTAT TC