本文主要是介绍transformer在生物基因DNA的应用:DNABERT、DNABERT-2,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
参考:
https://www.youtube.com/watch?v=mk-Se29QPBA&t=1388s
写明这些训练模型可以最终训练好可以进行DNA特征向量的提取,应用与后续

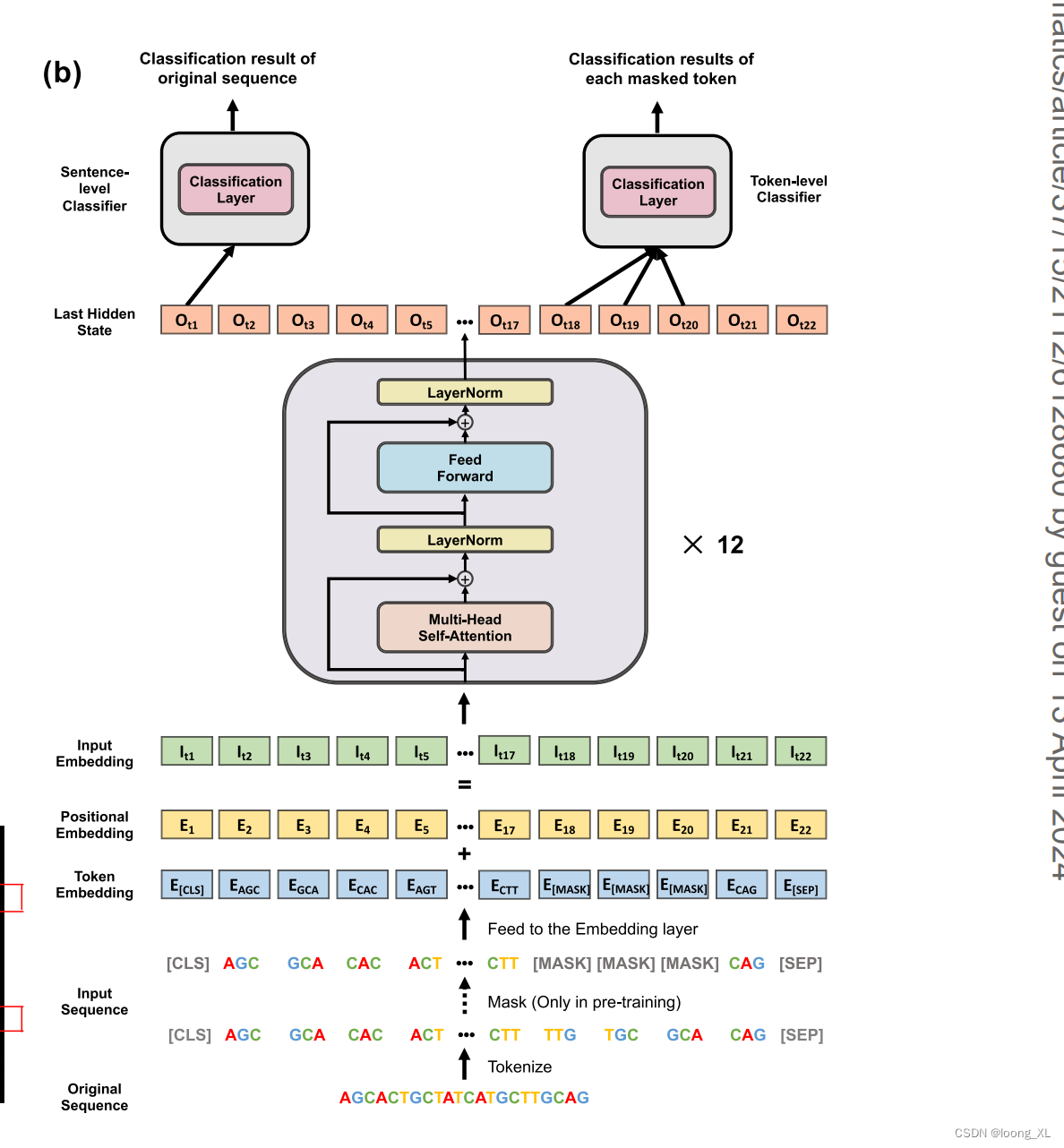
1、DNABERT
https://github.com/jerryji1993/DNABERT
主要思路就是把DNA序列当成连续文本数据,直接用成熟的自然语言训练模型transformer进行生物DNA序列数据的训练
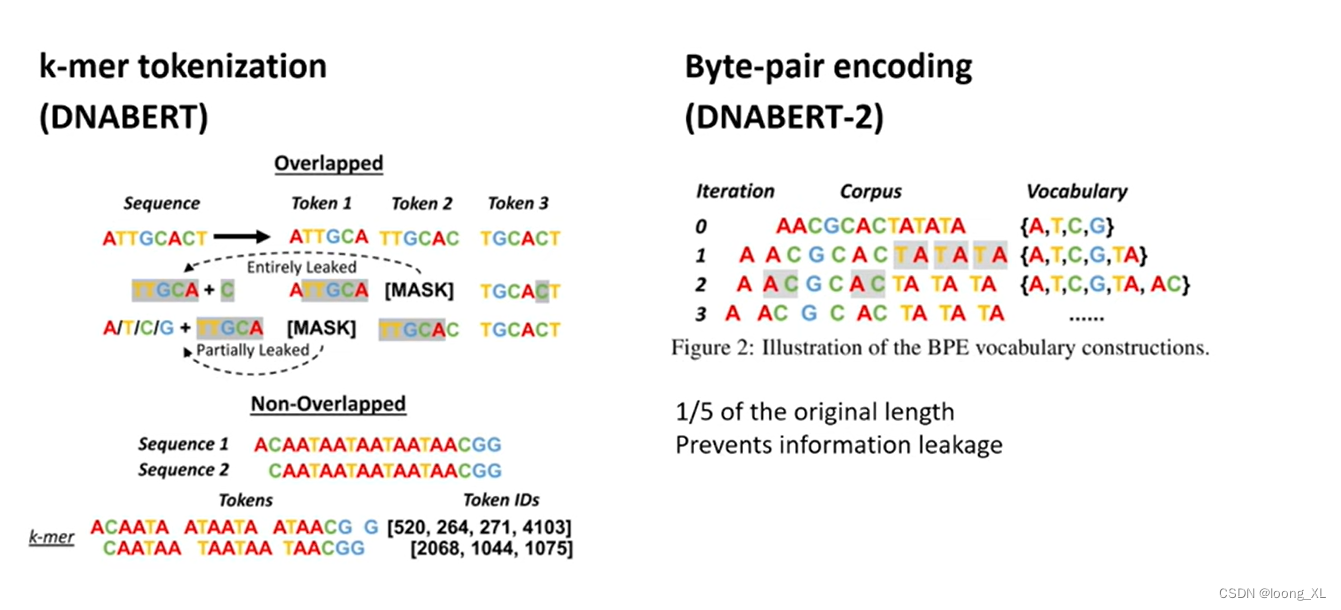
不同点主要就是ATCG序列切分token,DNA序创造了k-mer切分方法
3k-mer切分下图:

2、DNABERT-2
https://github.com/MAGICS-LAB/DNABERT_2
DNABERT-2主要是切次方法的改进
向量特征提前:
import torch
from transformers import AutoTokenizer, AutoModeltokenizer = AutoTokenizer.from_pretrained("zhihan1996/DNABERT-2-117M", trust_remote_code=True)
model = AutoModel.from_pretrained("zhihan1996/DNABERT-2-117M", trust_remote_code=True)dna = "ACGTAGCATCGGATCTATCTATCGACACTTGGTTATCGATCTACGAGCATCTCGTTAGC"
inputs = tokenizer(dna, return_tensors = 'pt')["input_ids"]
hidden_states = model(inputs)[0] # [1, sequence_length, 768]# embedding with mean pooling
embedding_mean = torch.mean(hidden_states[0], dim=0)
print(embedding_mean.shape) # expect to be 768# embedding with max pooling
embedding_max = torch.max(hidden_states[0], dim=0)[0]
print(embedding_max.shape) # expect to be 768
这篇关于transformer在生物基因DNA的应用:DNABERT、DNABERT-2的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







