本文主要是介绍ONE TRANSFORMER CAN UNDERSTAND BOTH 2D 3D MOLECULAR DATA,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
目录
摘要:
1 介绍:
2 相关工作:
3 TRANSFORMER-M
3.1符号和主干Transformer
3.2Transformer-M和培训策略
4 实验
4.1大规模预训练
4.2 pcqm4mv2性能(2d)
4.3 pdbind性能(2d & 3d)
4.4 qm9性能(3d)
4.5消融实验
5 结论
摘要:
与通常具有独特格式的视觉和语言数据不同,分子可以自然地使用不同的化学配方来表征。人们可以将分子看作二维图形,也可以将其定义为位于三维空间中的原子集合。对于分子表示学习,大多数先前的工作只针对特定的数据格式设计了神经网络,这使得学习到的模型可能无法适用于其他数据格式。我们相信一个通用的化学神经网络模型应该能够处理跨数据模式的分子任务。为了实现这一目标,在这项工作中,我们开发了一种新的基于transformer的分子模型,称为Transformer-M,它可以将2D或3D格式的分子数据作为输入,并生成有意义的语义表示。Transformer- m使用标准的Transformer作为主干架构,开发了两个独立的通道来编码二维和三维结构信息,并将其与网络模块中的原子特征结合起来。当输入的数据是特定格式时,相应的通道将被激活,另一个通道将被禁用。通过使用适当设计的监督信号对2D和3D分子数据进行训练,Transformer-M自动学习利用来自不同数据模式的知识并正确捕获表征。我们对变压器m进行了大量的实验。所有的实证结果表明,Transformer-M可以同时在二维和三维任务上取得较强的性能,表明其广泛的适用性。代码和模型将在https://github.com/lsj2408/Transformer-M上公开提供。
1 介绍:
深度学习方法已经彻底改变了许多领域,包括计算机视觉(He等人,2016)、自然语言处理(Devlin等人,2019;Brown et al, 2020)和游戏(Mnih et al, 2013;Silver et al, 2016)。最近,研究人员开始研究神经网络的力量是否可以帮助解决化学中的重要科学问题,例如,预测分子的性质并从大规模训练数据中模拟分子动力学(Hu et al ., 2020a;2021;Zhang et al ., 2018;Chanussot et al, 2020)。
化学与视觉和语言等传统领域的一个关键区别是数据的多模态。在视觉和语言中,数据实例通常具有特定形式的特征。例如,将图像定义为像素网格中的RGB值,而将句子定义为序列中的标记。相反,分子自然具有不同的化学配方。一个分子可以表示为一个序列(Weininger, 1988),一个二维图形(Wiswesser, 1985),或者一个位于三维空间的原子集合。2D和3D结构是最常用的公式,因为可以从中获得许多有价值的属性和统计数据(Chmiela等人,2017;Stokes et al, 2020)。然而,据我们所知,以往的工作大多集中在设计二维或三维结构的神经网络模型,使得以一种形式学习的模型无法应用于另一种形式的任务。
我们认为,化学中的通用神经网络模型至少应该能够处理跨数据模式的分子任务。在本文中,我们通过开发Transformer-M迈出了实现这一目标的第一步,Transformer-M是一种基于transformer的多功能分子模型,在2D和3D分子表示学习中都表现良好。请注意,对于一个分子,它的2D和3D形式描述了相同的原子集合,但使用了不同的结构表征。因此,关键的挑战是设计一个表达和兼容的模型,以捕获不同公式的结构知识,并训练参数从这两种信息中学习。transformer比其他架构更有利,因为它可以显式地将结构信号插入模型中作为偏置项(例如,位置编码(V aswani等人,2017;拉斐尔等人,2020))。我们可以方便地将二维和三维结构信息通过分离的通道设置为不同的偏置项,并将其与注意层中的原子特征结合起来。
体系结构。我们的Transformer- m的骨干网络由标准Transformer模块组成。我们开发了两个独立的通道来编码二维和三维结构信息。
2D通道采用度编码、最短路径距离编码和从2D图结构中提取的边缘编码,遵循Ying等(2021a)。最短路径距离编码和边缘编码反映了一对原子的空间关系和键合特征,并作为softmax最大注意中的偏置项。度编码被添加到输入层的原子特征中。
对于3D通道,我们遵循Shi等人(2022)使用3D距离编码对3D几何结构中原子之间的空间距离进行编码。每个原子对的欧几里得距离通过高斯基核函数(Scholkopf et al, 1997)进行编码,并将用作softmax注意中的偏差项。对于每个原子,我们将其与所有其他原子之间的三维距离编码相加,并将其添加到输入层的原子特征中。图1给出了一个说明。
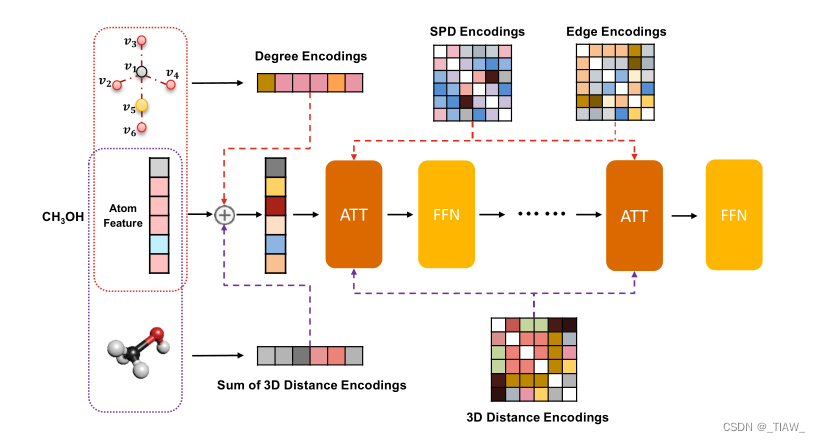
图1:我们的Transformer-M模型体系结构的插图。我们在主干变压器上建立了两个通道。红色通道用于包含度、最短路径距离和边缘信息的2D图形结构的数据。紫色通道为3D几何结构数据激活,以利用欧几里得距离信息。不同的编码位于相应的模块中。
训练。除了两个结构通道中的参数外,Transformer-M中的所有其他参数(例如自关注和前馈网络)对于不同的数据模式都是共享的。我们为Transformer-M设计了一种联合训练方法来学习其参数。在训练过程中,当批处理中的实例仅与2D图结构相关联时,2D通道将被激活,3D通道将被禁用。类似地,当批处理中的实例使用3D几何结构时,3D通道将被激活,2D通道将被禁用。当同时给出二维和三维信息时,两个通道都会被激活。这样,我们可以从单独的数据库中收集2D和3D数据,并根据不同的训练目标训练Transformer-M,使训练过程更加灵活。我们期望单个模型能够学习识别和合并来自不同模态的信息,并有效地利用这些参数,从而获得更好的泛化性能。
实验结果。我们在OGB大规模挑战(OGBLSC)中使用PCQM4Mv2数据集(Hu等人,2021)来训练我们的Transformer-M,它由340万个2D和3D形式的分子组成。通过针对3D数据的预文本3D去噪任务,训练模型预测不同格式的每个数据实例的预先计算的HOMO-LUMO间隙。通过预训练模型,我们可以直接使用或微调不同数据格式的各种分子任务的参数。首先,我们证明了在PCQM4Mv2任务的验证集上,仅包含二维分子图,我们的Transformer-M大大超过了以前的所有作品。这种改进归功于联合训练,它有效地缓解了过拟合问题。其次,在pdbbind上(Wang et al ., 2004;2005b) (2D&3D),与强大的基线相比,经过微调的Transformer-M实现了最先进的性能。最后,在QM9 (Ramakrishnan et al, 2014) (3D)基准上,与最近的方法相比,微调后的Transformer-M模型实现了具有竞争力的性能。所有结果表明,我们的Transformer-M具有在化学领域广泛应用的通用模型的潜力。
2 相关工作:
用于学习二维分子表征的神经网络。图神经网络(GNN)广泛用于分子图表示学习(Kipf & Welling, 2016;Hamilton等人,2017;Gilmer等人,2017;Xu et al ., 2019;[5] [c] .李晓明等人,2018]。GNN通过递归聚合(即消息传递)和从邻居表示中更新节点表示来学习节点和图表示。通过使用不同的聚合和更新策略来开发不同的体系结构。我们建议读者参考Wu et al(2020)进行全面调查。
最近,许多作品将Transformer模型扩展到图形任务(Dwivedi & Bresson, 2020;Kreuzer等,2021;瑛等,2021a;罗等人,2022;Kim et al, 2022;Rampášek等,2022;Park et al, 2022;Hussain et al, 2022;Zhang et al, 2023)。开创性的作品包括graphhormer (Ying等人,2021a),它开发了图形结构编码,并在标准Transformer模型中使用它们。
用于学习三维分子表征的神经网络。用三维几何信息学习分子表示在许多应用中是必不可少的,例如分子动力学模拟。最近,研究人员设计了一些架构来保持一些必要的变换(如旋转和平移)的不变和等变性质。sch等人(2017)使用连续滤波卷积层来模拟分子中的量子相互作用。Thomas等人(2018)使用球面谐波构建的滤波器来构建旋转和平移等变神经网络。Klicpera等(2020)提出了定向消息传递,保证了它们的嵌入是旋转等变的。Liu et al . (2022);Wang et al .(2022)利用球坐标捕捉几何信息,实现等方差。Hutchinson等人(2021);Thölke和De Fabritiis(2021)建立了变压器模型,保留了等变特性。Shi等人(2022)将Ying等人(2021a)扩展到3D Transformer模型,该模型在大规模分子建模挑战中获得了更好的结果(Chanussot等人,2020)。
分子的多视角学习。二维图形结构和三维几何结构可以看作是同一分子的不同视图。受视觉中的对比预训练方法的启发(Chen et al ., 2020;他等人,2020;Radford et al ., 2021),许多作品研究了联合使用二维和三维信息的分子预训练方法。Stärk等人(2022)使用两个编码器分别对二维和三维分子信息进行编码,同时最大限度地提高表征之间的互信息。Liu等人(2021a)推导了GraphMVP框架,该框架使用对比学习和重建来预训练2D编码器和3D编码器。Zhu et al .(2022)统一了上述二维和三维预训练方法,提出了一种可以通过三维几何特征增强的二维GNN模型。与这些工作不同的是,我们的目标是开发一个兼容二维和三维分子任务的单一模型。此外,上述所有工作都是使用成对的2D和3D数据来训练模型,而这种成对的数据对于训练我们的模型来说并不是很强的要求。
通用模型。构建一个可以处理多个任务的单一代理,甚至是跨模式的代理,是深度学习领域的最新发现。在早期,研究人员发现,一个单一的多语言翻译模型可以使用相同的权重翻译数十种语言,并且比罕见语言的双语翻译模型表现更好(Lample & Conneau, 2019;Conneau等人,2019;薛等,2020;Liu et al, 2020)。大规模语言模型(Devlin et al ., 2019;Brown等人,2020)是另一个可以应用于使用上下文学习或微调的不同下游任务的例子。Reed等人(2022)通过构建单一的通才代理Gato进一步突破了边界。这个智能体使用相同的网络,具有相同的权重,但可以玩Atari,字幕图像,并像人类一样进行对话。我们的工作也在这个方向上。我们专注于开发一种通用的化学模型,该模型可以将不同格式的分子作为输入,并在少量额外的训练数据的情况下,在各种分子任务中表现良好。
3 TRANSFORMER-M
在本节中,我们将介绍Transformer- m,这是一个通用的Transformer,可作为2D和3D分子表示学习的通用架构。首先,我们介绍了一些符号,并概述了主干Transformer体系结构的基础知识(第3.1节)。之后,我们提出了具有不同数据模式的两个结构通道的Transformer-M模型(第3.2节)。
3.1符号和主干Transformer
一个分子M是由一组原子在引力作用下聚集在一起的。我们记X∈R^n×d为具有特征的原子,其中n为原子数,d为特征维数。M的结构可以用不同的形式表示,如二维图形结构和三维几何结构。对于二维图结构,原子是通过化学键显式连接的,我们定义M^2D = (X, E),其中e (i,j)∈E表示原子i与j之间存在边的边特征(即键的类型)。对于三维几何结构,给出了每个原子i在笛卡尔坐标系中的位置ri。定义M^3D = (X, R),其中R = {r1,…, rn}, ri∈R3。我们的目标是设计一个参数化模型,它可以将M^2D或M^3D(或两者都)作为输入,获得上下文表示,并对下游任务进行预测。
Transformer层。我们在这项工作中使用的主干架构是Transformer模型(V aswani et al, 2017)。Transformer由堆叠的Transformer块组成。Transformer块由两层组成:自关注层,然后是前馈层,两层都具有规范化(例如,LayerNorm (Ba等人,2016))和跳过连接(He等人,2016)。表示X(l)作为第(l + 1)块的输入,并定义X(0) = X。对于输入X(l),第(l + 1)块的工作方式如下:
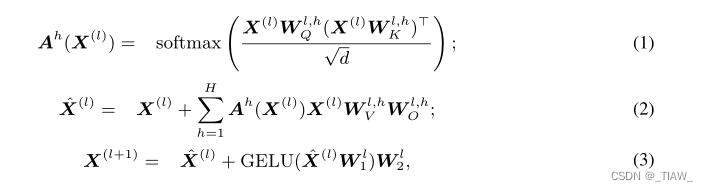
![]()
H为注意头的个数,dH为每个注意头的维度,r为隐藏层的维度。Ah(X)通常被称为注意矩阵。
位置编码。Transformer中的另一个重要组件是位置编码。请注意,自关注层和前馈层不使用输入元素的顺序(例如,单词标记),这使得模型无法捕获结构信息。原始论文(V aswani et al, 2017)开发了有效的位置编码来编码句子结构信息,并将其作为偏见项明确地集成到模型中。简而言之,许多工作都意识到位置编码在将标准Transformer扩展到语言之外的更复杂的数据结构方面起着至关重要的作用。通过使用领域知识精心设计结构编码,Transformer已成功应用于图像和图形领域,并取得了令人印象深刻的性能(Dosovitskiy等人,2020;刘等,2021b;Ying et al ., 2021a)。
3.2Transformer-M和培训策略
我们可以看到,3.1节定义的两种分子式使用了相同的原子特征空间,但结构的表征不同(图形结构E vs几何结构R)。因此,关键的挑战是设计一个兼容的体系结构,该体系结构可以利用E或R(或两者)中的结构信息,并以一种有原则的方式将它们与原子特性结合起来。
Transformer是实现这一目标的合适支柱,因为我们可以将结构信息编码为偏置项,并将它们适当地插入不同的模块中。此外,使用Transformer,我们可以通过将结构信息分解为成对编码和原子编码,以统一的方式处理E和R。在不损失通用性的情况下,我们选择使用Ying等人(2021a)提出的图形和几何转换器中的编码策略;Shi et al(2022)。为了完整起见,我们简要介绍这些结构编码,并展示如何在Transformer-M中利用它们。请注意,我们的设计方法也适用于其他编码策略(Hussain et al, 2022;Park et al, 2022;Thölke & De Fabritiis, 2021)。具体结果见附录B.5。
在E中编码成对关系。我们使用两个术语来编码图中任意原子对之间的结构关系。首先,我们对两个原子之间的最短路径距离(SPD)进行编码,以反映它们的空间关系。设ΦSPDij表示原子i和j之间的SPD编码,它是由i和j之间最短路径的距离决定的可学习标量。其次,我们沿着i和j之间的最短路径编码边缘特征(例如化学键类型),以反映键信息。对于大多数分子来说,任意两个原子之间只存在一条明显的最短路径。表示从i到j的最短路径中的边为SPij = (e1,e2,…,eN),定义i和j之间的边缘编码为ΦEdge ij =1/N*(求和n从1到N =1) en (wn)T,其中wn为与边缘特征相同维数的可学习向量。将ΦSPD和ΦEdge表示为形状为n × n的SPD编码和边缘编码的矩阵形式。
在R中编码成对关系。我们对欧几里得距离进行编码,以反映三维空间中任意一对原子之间的空间关系。对于每个原子对(i, j),我们首先用高斯基核函数处理它们的欧几里得距离(Scholkopf et al, 1997);
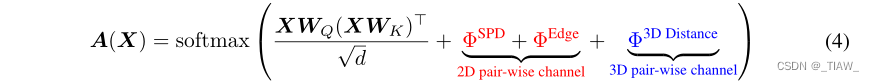
用E编码原子结构信息。对于原子i, Eqn。(4)根据I与其他原子之间的语义(第一项)和空间关系(后三项)计算归一化权重。然而,这些信息仍然不充分。例如,每个原子的重要性(即中心性)在注意力中是缺失的。对于每个原子i,我们用它的度作为中心性信息。形式上,设ΨDegree i表示原子i的度编码,这是一个由原子度决定的d维可学习向量。表示ΨDegree = [ΨDegree 1, ΨDegree 2,…][ΨDegreen]作为所有原子的中心性编码,其形状为n × d。
在R中编码原子的结构信息。类似于二维原子的中心性编码,对于几何数据,我们在三维空间中编码每个原子的中心性。对于每个原子i,我们将其与所有其他原子之间的三维距离编码相加。设ΨSum of 3D Distancei表示原子i的中心性编码,则ΨSum of 3D Distancei = P j∈[n] ψ(i,j)W 3D,其中W 3D∈RK×d是一个可学习的权重矩阵。同样,我们定义三维距离的ΨSum为所有原子的编码,其形状为n × d。

通过这种简单的方法,分子的二维和三维结构信息被整合到一个Transformer模型中。很容易检查Transformer-M是否保留了两种数据格式的等变属性。
训练。下一步是学习Transformer-M中的参数,以便从每种数据格式中捕获有意义的表示。为了实现这一目标,我们开发了一种简单灵活的联合训练方法来学习Transformer-M。我们首先收集不同格式(2D/3D)的数据集,并在每种格式上定义监督/自监督任务(例如,能量回归),并分别针对每个目标在所有数据上训练模型。具体来说,在训练期间,如果数据实例来自2D格式的数据集,则2D通道被激活,3D通道被禁用。模型参数将被优化以最小化相应的(即2D)目标。当数据实例来自3D格式的数据集时,只有3D通道被激活,模型将学习最小化3D目标。如果模型以2D和3D两种格式的分子作为输入,这两个通道都会被激活。与多视图学习方法相比,我们可以使用未配对的2D和3D数据来训练Transformer-M,使训练过程更加灵活。
由于联合训练,transformer- m可以更好地泛化。之前的一些研究(Liu et al ., 2021a)发现,二维图形结构和三维几何结构包含互补的化学知识。例如,二维图形结构只包含具有键类型的键,而三维几何结构包含长度和角度等细粒度信息。又如,三维几何结构通常通过密度泛函理论(DFT) (Burke, 2012)等计算模拟获得,这可能存在近似误差。二维图形由领域专家构建,在一定程度上为三维结构提供参考。通过参数共享的二维和三维数据联合训练,我们的模型可以学习更多的化学知识,而不是对数据噪声过拟合,并且在二维和三维任务上都有更好的表现。
未来的发展方向。作为最初的尝试,我们的Transformer-M开辟了一种开发通用分子模型的方法,以处理不同数据格式的各种化学任务。我们相信这是一个起点,未来有更多的可能性可以探索。例如,在这项工作中,我们使用了一种简单的方法,将二维和三维结构的结构信息线性组合,我们认为应该有其他有效的方法来融合这种编码。我们的模型还可以与以前的多视图对比学习方法相结合。如何使用这些方法预训练我们的模型是值得研究的。
4 实验
在本节中,我们对transformer-m的性能进行了实证研究。首先,我们在OGB大规模挑战(Hu et al, 2021)的PCQM4Mv2训练集上预训练我们的模型(第4.1节)。利用预训练模型,我们对不同数据格式的分子任务进行了实验,并评估了我们的Transformer-M的通用性和有效性。由于篇幅限制,我们研究了三个具有代表性的任务,PCQM4Mv2 (2D,章节4.2),PDBBind (2D和3D,章节4.3)和QM9 (3D,章节4.4)。消融研究见第4.5节。所有代码都是基于graphhormer (Ying等人,2021a)在PyTorch (Paszke等人,2019)中的官方代码库实现的。
4.1大规模预训练
我们的Transformer-M是在OGB大规模挑战赛的PCQM4Mv2训练集上进行预训练的(Hu et al ., 2021)。训练样本总数为337万。每个分子都与其二维图形结构和三维几何结构相关联。每个分子的HOMO-LUMO能隙作为标签,通过基于dft的方法获得(Burke, 2012)。
我们遵循Ying等人(2021a),采用12层Transformer-M模型。隐藏层和前馈层的维数设置为768。注意头的数量设置为32。高斯基核数设置为128。为了训练Transformer-M,我们为每个数据实例提供三种模式:(1)激活2D通道并禁用3D通道(2D模式);(2)激活3D通道,禁用2D通道(3D模式);(3)激活两个通道(2D+3D模式)。训练过程中每个数据实例的模式是根据预定义的分布随机绘制的,实现类似于Dropout (Srivastava et al, 2014)。在这项工作中,我们使用了两个训练目标。第一个是监督学习目标,旨在预测每个分子的HOMO-LUMO能隙。此外,我们还使用了一个名为3D位置去噪的自监督学习目标(Godwin et al, 2022;Zaidi et al, 2022),这是特别有效的。在训练过程中,如果数据实例处于3D模式,我们在每个原子的位置上添加高斯噪声,并要求模型从噪声输入中预测噪声。该模型被优化为最小化上述两个目标的线性组合。详细设置请参见附录B.1。
4.2 pcqm4mv2性能(2d)
在对模型进行预训练后,我们在PCQM4Mv2的验证集上对我们的Transformer-M进行了评估。
请注意,PCQM4Mv2的验证集仅由2D格式的分子组成。因此,我们可以用它来评估Transformer-M在二维分子数据上的表现。该任务的目标是预测HOMU-LUMO能量差,评估指标是平均绝对误差(MAE)。由于我们的训练目标包括HOMO-LUMO间隙预测任务,我们没有对任何数据的模型参数进行微调。在推理过程中,只有2D通道被激活。
我们选择了几个涵盖消息传递神经网络(MPNN)变体和图转换器的强基线。有关基线的详细说明载于附录B.2。
结果如表1所示。可以很容易地看到,我们的Transformer-M大大超过了所有基线,例如,与之前的最佳模型相比,相对MAE降低了8.2% (Rampášek等人,2022),在PCQM4Mv2数据集上建立了一个新的最先进的技术。请注意,我们的总体架构与graphhormer模型相同(Ying et al, 2021a)。Transformer-M和graphhormer基线之间的唯一区别是graphhormer仅使用2D数据进行训练,而Transformer-M使用2D和3D结构信息进行训练。因此,我们可以得出结论,Transformer-M在二维分子数据上表现良好,共享参数的二维-三维联合训练确实有助于模型学习更多的化学知识。
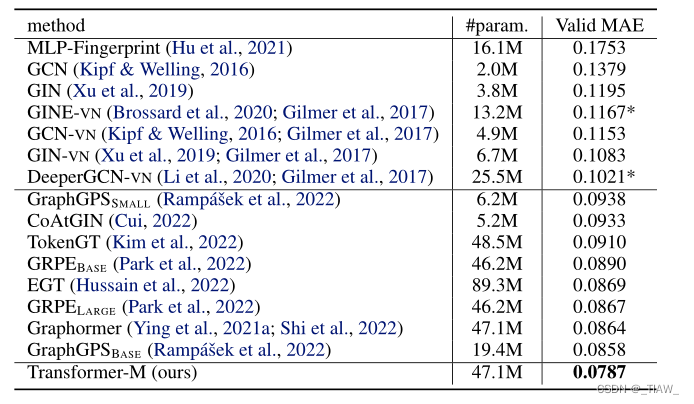
表1:OGB大规模挑战中PCQM4Mv2验证集的结果(Hu et al ., 2021)。评价指标为平均绝对误差(MAE) [eV]。我们报告OGB基线的官方结果,并使用*来表示我们的实施结果。粗体值表示最佳性能。
4.3 pdbind性能(2d & 3d)
为了验证Transformer-M的兼容性,我们在PDBBind数据集(version 2016, Wang et al (2004;2005b)),是基于结构的虚拟筛选中使用最广泛的数据集之一(jimsamnez等人,2018;Stepniewska-Dziubinska等,2018;Zheng et al ., 2019)。PDBBind数据集由蛋白质配体复合物作为数据实例组成,这些数据实例是在与pKa(或- log Kd, - log Ki)亲和值相关的生物测定实验中获得的。对于每个数据实例,提供了三维几何结构,并通过预定义的规则构建了二维图形结构。这项任务需要模型来预测蛋白质-配体复合物的结合亲和力,这对药物发现至关重要。在PCQM4Mv2训练集上进行预训练后,我们的Transformer-M模型在PDBBind数据集的核心集上进行微调和评估。
我们将我们的模型与包括经典方法、基于cnn的方法和gnn的竞争性基线进行了比较。所有实验用不同的种子重复五次。报告平均性能。由于篇幅限制,我们在附录B.3中详细介绍了基线和实验设置。结果如表2所示。我们的Transformer-M在所有评估指标上的表现都明显优于所有基线,例如Pearson相关系数(R)有3.3%的绝对提高。值得注意的是,PDBBind数据集的数据实例是蛋白质配体复合物,而我们的模型是在简单分子上进行预训练的,这证明了Transformer-M的可转移性。
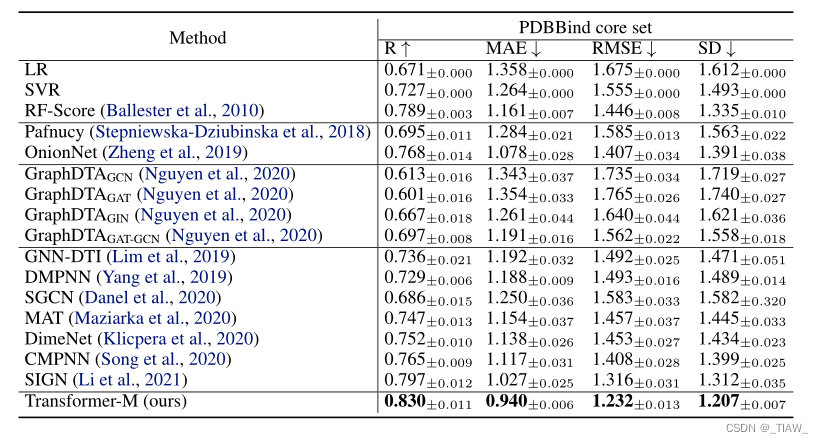
表2:PDBBind核心集(2016版)的结果(Wang et al ., 2004;2005 b)。评价指标包括皮尔逊相关系数(R)、平均绝对误差(MAE)、均方根误差(RMSE)和标准差(SD)。我们报告了Li等人(2021)的官方基线结果。粗体值表示最佳性能。
4.4 qm9性能(3d)
我们使用QM9数据集(Ramakrishnan等人,2014)以3D数据格式评估我们的Transformer-M在分子任务上的表现。QM9是由134k个稳定的有机小分子组成的量子化学基准。这些分子对应于由1660亿个有机分子组成的GDB-17化学宇宙中的133885个物种。每个分子都与12个靶标相关联,包括其能量、电子和热力学性质。分子的三维几何结构被用作输入。根据Thölke和De Fabritiis(2021),我们分别随机选择10,000和10,831个分子进行验证和测试评估。剩下的分子被用来微调我们的Transformer-M模型。我们观察到,之前的一些研究使用了不同的数据分割比率,或者没有描述评估细节。为了公平比较,我们选择了原始论文中使用相似分割比率的基线。详细的基线和实验设置见附录B.4。
结果如表3所示。可以看出,与这些基线相比,我们的Transformer-M取得了具有竞争力的性能,这表明该模型与3D分子数据兼容。特别是,Transformer-M在HUMO、LUMO和HUMO-LUMO预测上表现最好。这表明在预训练任务中学到的知识可以更好地转移到类似的任务中。请注意,该模型在其他一些任务上执行得不是很好。我们认为Transformer-M可以在几个方面进行改进,包括采用精心设计的输出层(Thölke & De Fabritiis, 2021)或使用更多自监督训练信号进行预训练。
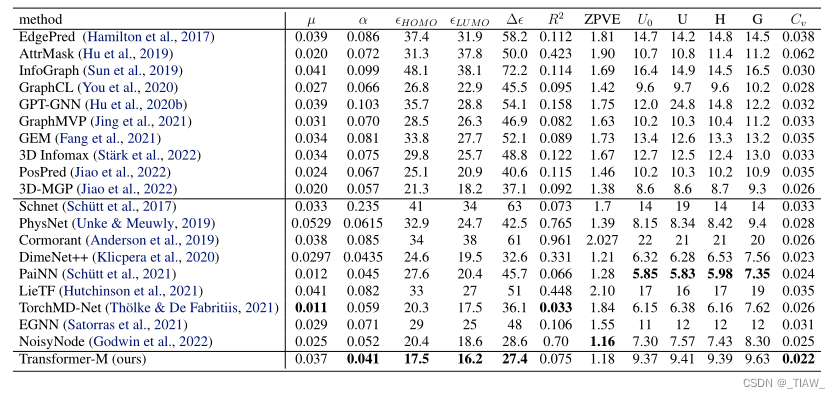
表3:QM9的结果(Ramakrishnan et al ., 2014)。评估指标是平均绝对误差(MAE)。我们报告了Thölke & De Fabritiis(2021)的官方基线结果;Godwin et al (2022);Jiao等(2022)。粗体值表示最佳性能。
4.5消融实验
在本节中,我们进行了一系列实验来研究我们的变压器m的关键设计。在本文中,我们使用两个训练目标来训练模型,并将两个训练目标的效果进行对比。此外,我们使用三种模式来激活具有预定义分布的不同通道,我们将研究分布对最终性能的影响。
由于篇幅限制,我们将在附录B.5中对我们的Transformer-M模型进行更多的分析。
训练前任务的影响。如第4.1节所述,我们的Transformer-M模型通过两个任务在PCQM4Mv2训练集上进行预训练:(1)预测分子在2D和3D格式下的HOMO-LUMO间隙。(2)三维位置去噪。我们对PCQM4Mv2和QM9数据集进行消融研究,以检查这两个目标是否有利于下游任务。具体来说,我们进行了两个附加实验。第一个实验是在PCQM4Mv2上从头开始训练Transformer-M模型,使用其2D图形数据,QM9使用其3D几何数据,以检查整体预训练方法的好处。第二个实验是在不使用3D去噪任务的情况下对Transformer-M进行预训练,研究提出的2D-3D联合预训练方法的有效性。结果如表4所示。可以看出,联合预训练在PCQM4Mv2和QM9数据集上都显著提高了性能。此外,3D位置去噪任务也是有益的,特别是在3D格式的QM9数据集上。
模式分布的影响。表示(p2D, p3D, p2D&3D)为4.1节中提到的模态的概率。我们通过实验研究了不同分布对模型性能的影响。我们选择三个分布(p2D, p3D, p2D&3D)分别为:1:1:1,1:2:2和1:2:1。结果如表4所示。我们在PCQM4Mv2和QM9数据集上得到了一致的结论:1)对于所有三种配置,我们的Transformer-M模型都取得了较强的性能,这表明我们的联合训练对超参数选择具有鲁棒性;2)在3D模式上使用稍大的概率可以获得最佳效果。
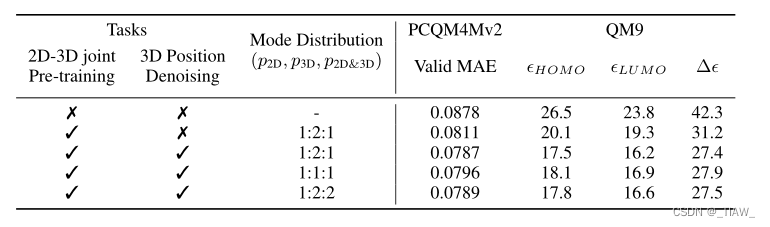
表4:预训练任务和模式分布对Transformer-M的影响。为了公平比较,所有其他超参数保持不变。
5 结论
在这项工作中,我们向通用分子模型迈出了第一步。提出的Transformer-M提供了一种很有前途的方法来处理2D和3D格式的分子任务。我们使用两个独立的通道对二维和三维结构信息进行编码,并将其集成到主干Transformer中。当输入的数据是特定格式时,相应的通道将被激活,另一个通道将被禁用。通过对2D和3D分子数据的简单训练任务,我们的模型自动学习利用不同数据格式的化学知识并正确捕获表征。进行了大量的实验,所有的实证结果都表明,我们的Transformer-M可以同时在2D和3D任务上取得较强的性能。我们的transformer- m的潜力可以在化学领域的广泛应用中得到进一步的探索。
这篇关于ONE TRANSFORMER CAN UNDERSTAND BOTH 2D 3D MOLECULAR DATA的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!