nanopore专题
2024.09.04【读书笔记】|如何使用Tombo进行Nanopore Direct RNA-seq(DRS)分析
文章目录 Tombo快速使用介绍模型介绍RNA修饰分析步骤特异性替代碱基检测(推荐)De novo canonical model comparison ONT全长转录组分析步骤疑难解答Minimap2在比对nanopore直接RNA-seq数据时的最佳实践和参数设置有哪些?featureCounts在进行RNA-seq定量分析时,如何选择最合适的参考基因组注释文件?Tombo序列重校正过程
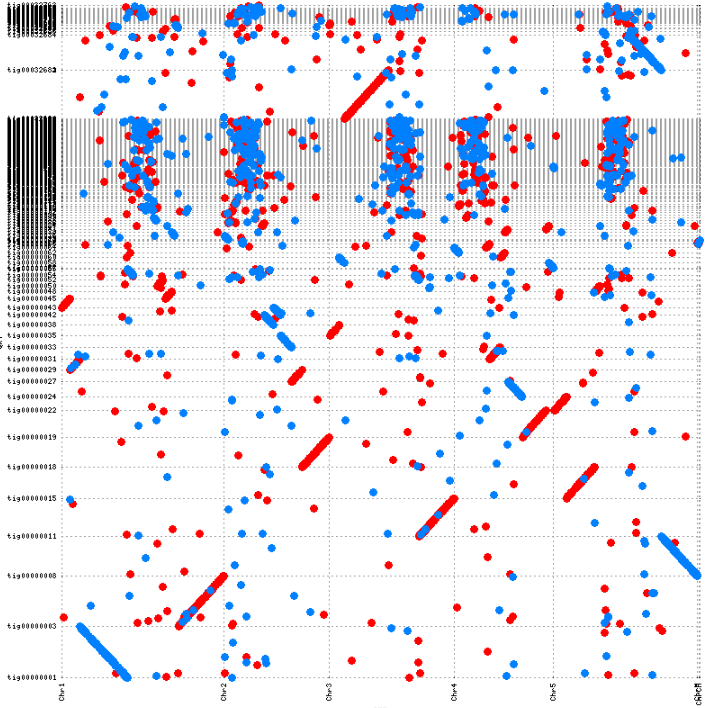
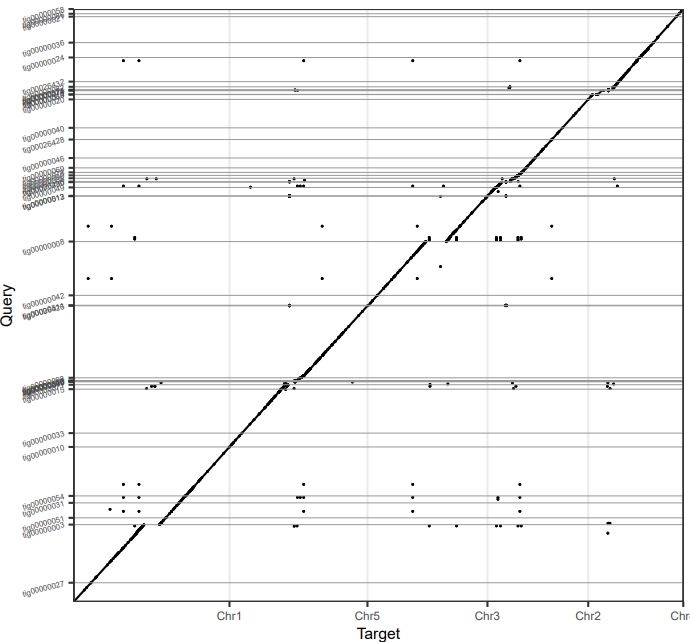
「杂谈」Nanopore组装的拟南芥基因组效果如何?
使用的数据来自于一篇发在NC的拟南芥的基因组文章,文章用了minimap/miniasm 进行组装,然后用racon和Pilon进行polish, 最后拼接处62 contigs 且N50 = 12.3 Mb。 wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/ERR217/003/ERR2173373/ERR2173373.fastq.gzseqkit seqk
NECAT: Nanopore数据的高效组装工具
NECAT是肖传乐老师团队开发的一个针对Nanopore数据组装的软件,目前该工具尚未发表,除了https://github.com/xiaochuanle/NECAT有软件的介绍外,暂时没有中文资料介绍NECAT的使用。 太长不看的结论: Nanopore的组装推荐用下NECAT。组装之后是先用MEDAKA做一遍三代polish,然后用NextPolish默认参数做二代polish。 这篇将
Nanopore宏基因组分析永久冻土融化过程中微生物群落
Devin Drown和他在美国阿拉斯加大学费尔班克斯分校(University of Alaska Fairbanks)的团队一直在利用OxfordNanopore纳米孔技术带来的长读长测序的优势,致力于研究永久冻土的融化对土壤微生物群落造成的影响1。永久冻土是土壤、砾石和沙子的永久冻结层,位于地球表面或地表以下。 永久冻土指的是至少连续冻结两年的地区,而大多数的永久冻土已经冻结了数百甚至
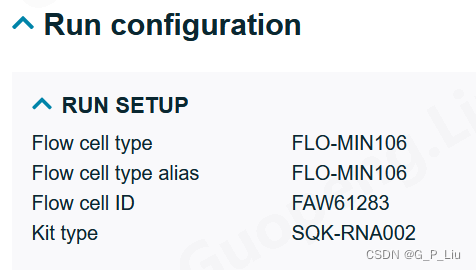
Guppy从nanopore下机fast5文件中进行basecalling时所需config文件的确定
使用Guppy进行basecalling时需要指定与芯片类型匹配的config文件。所有config文件均在Guppy软件的data文件夹下。如果已知所用芯片型号,则直接选择对应的config即可。若不知道(比如第三方测序的或者其他来源的),可以根据下机报告(一个***Run Report***.html文件)中的“Run configuration”部分查看: 可以看到这批数据用的flow
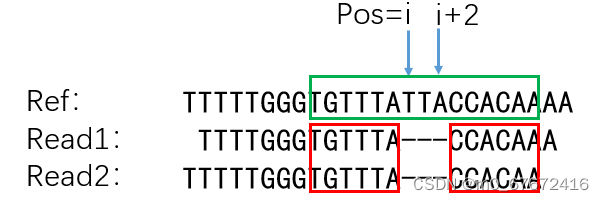
Oxford Nanopore Technologies实战应用之电信号比对工具Tombo
目录 一、前言 二、Tombo快速使用 1.安装 2.快速使用 3.比对结果解析 4.高阶应用 三、总结 一、前言 对于纳米孔测序常规用途,如基因组比对、SNV鉴别、宏基因组物种分析而言,用户一般使用碱基识别软件输出的fastq文件即可展开相应分析。但对于某些特殊用途,如甲基化鉴别、碱基识别算法开发,则需要深入分析纳米孔测序产生的fast5文件中原始电信号与碱基(