本文主要是介绍Oxford Nanopore Technologies实战应用之电信号比对工具Tombo,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
目录
一、前言
二、Tombo快速使用
1.安装
2.快速使用
3.比对结果解析
4.高阶应用
三、总结
一、前言
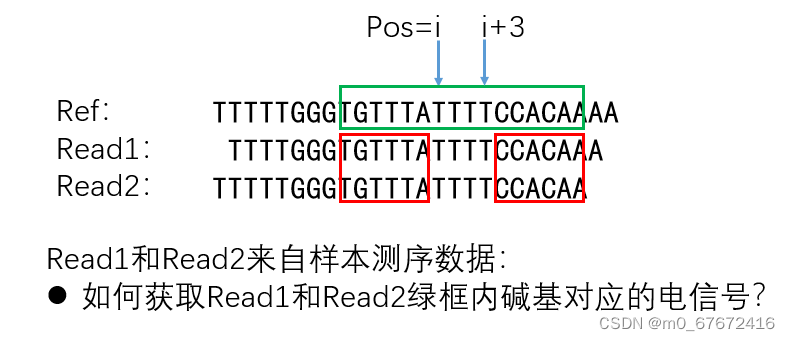
对于纳米孔测序常规用途,如基因组比对、SNV鉴别、宏基因组物种分析而言,用户一般使用碱基识别软件输出的fastq文件即可展开相应分析。但对于某些特殊用途,如甲基化鉴别、碱基识别算法开发,则需要深入分析纳米孔测序产生的fast5文件中原始电信号与碱基(A/T/G/C)之间的对应关系。那么,有没有软件可以实现这种功能呢?答案是ONT官方发布的软件Tombo。
二、Tombo快速使用
1.安装
使用conda进行快速安装。
# install via bioconda environment (https://bioconda.github.io/#set-up-channels)
conda install -c bioconda ont-tombo2.快速使用
假定fast5文件中已经有basecalls(即ATGC碱基),且Reference序列已知,那么最快速使用Tombo进行电信号比对的命令如下。输出的比对结果会写入到fast5文件中。
tombo resquiggle path/to/fast5s/ genome.fasta --processes 4 --num-most-common-errors 53.比对结果解析
首先要读入fast5文件,可以使用h5py中的h5py.File函数读入,也可以使用其他第三方fast5分析软件。
import h5py
fast5_info = h5py.File(fast5_path, 'r')然后,就可以在fast5_info['Analyses']['RawGenomeCorrected_000']['BaseCalled_template']['Events'][:]中找到碱基与电信号的对应关系。
并且,可以通过fast5_info['Analyses']['RawGenomeCorrected_000']['BaseCalled_template']['Alignment']的attrs来查看每条fast5与参考序列的比对信息,如比对起止位点、正负链信息等。
4.高阶应用
随着ONT测序技术的更新,并非所有fast5文件都可以直接被Tombo读入,此时需要考虑2个问题:
(1)fast5中是否有basecalls?某些用户在设置测序控制软件时,产生的fast5并不一定含有basecalls,此时就需要re-basecalling。
(2)fast5可能使用了VBZ压缩,此时就需要先安装hdf5插件(参考https://github.com/nanoporetech/vbz_compression/),然后才能继续使用h5py读取fast5。
三、总结
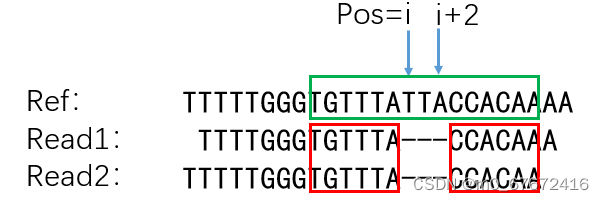
其实使用tombo resquiggle进行电信号比对还有许多细节需要注意,有兴趣的读者可以参考其说明书(https://nanoporetech.github.io/tombo/resquiggle.html)。比如,reads相对于reference序列出现了Indel(下图所示)要怎么获取电信号,Tombo也给出了解决方案。
这篇关于Oxford Nanopore Technologies实战应用之电信号比对工具Tombo的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







