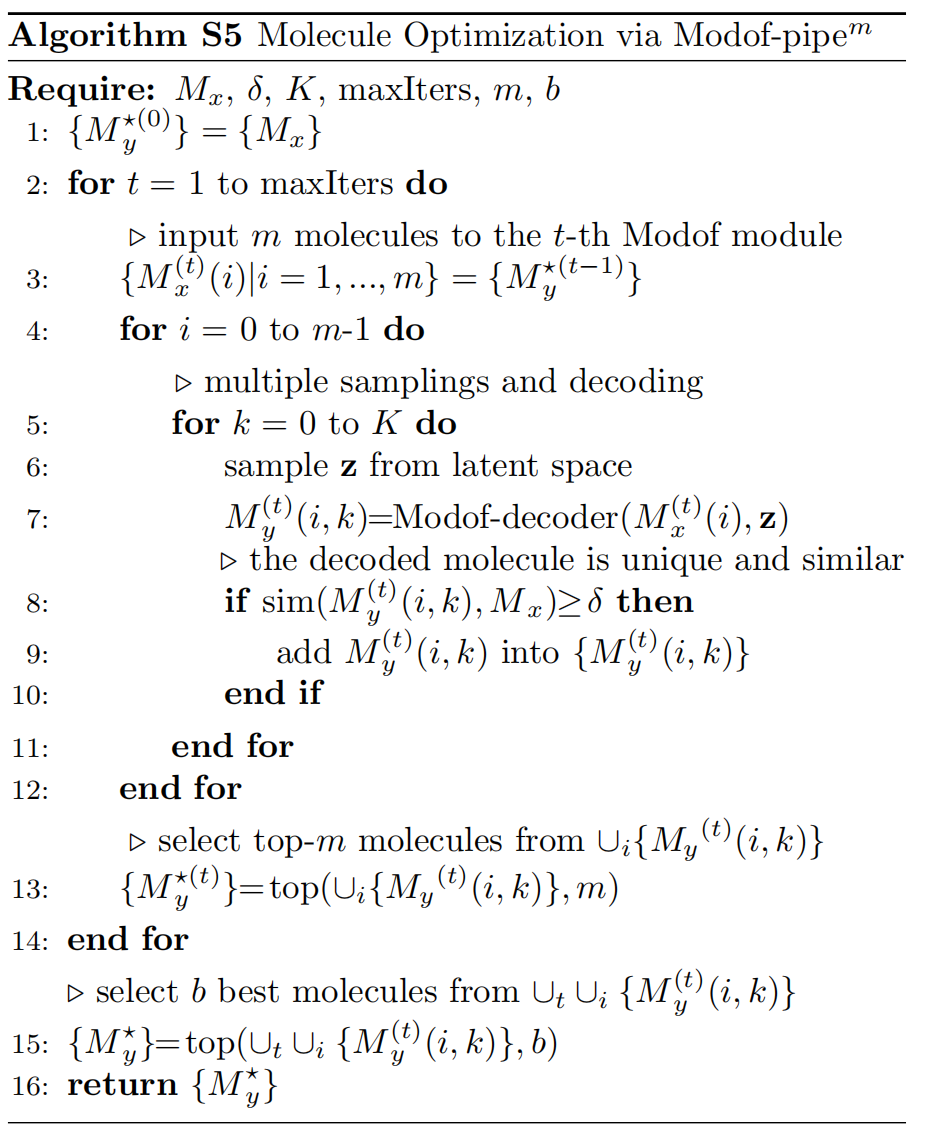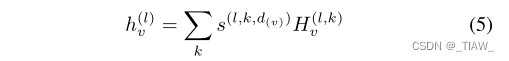molecule专题
2024-python字典-报错compounds.iloc[0].molecule_structures.keys()
2024-python字典-报错compounds.iloc[0].molecule_structures.keys() .keys()拿不到 需要如何解决呢 import ast# 假设 "molecular_structures "是一个看起来像 Python 字典的字符串molecule_structures = df_VEGFR2_compounds.iloc[0].molecul
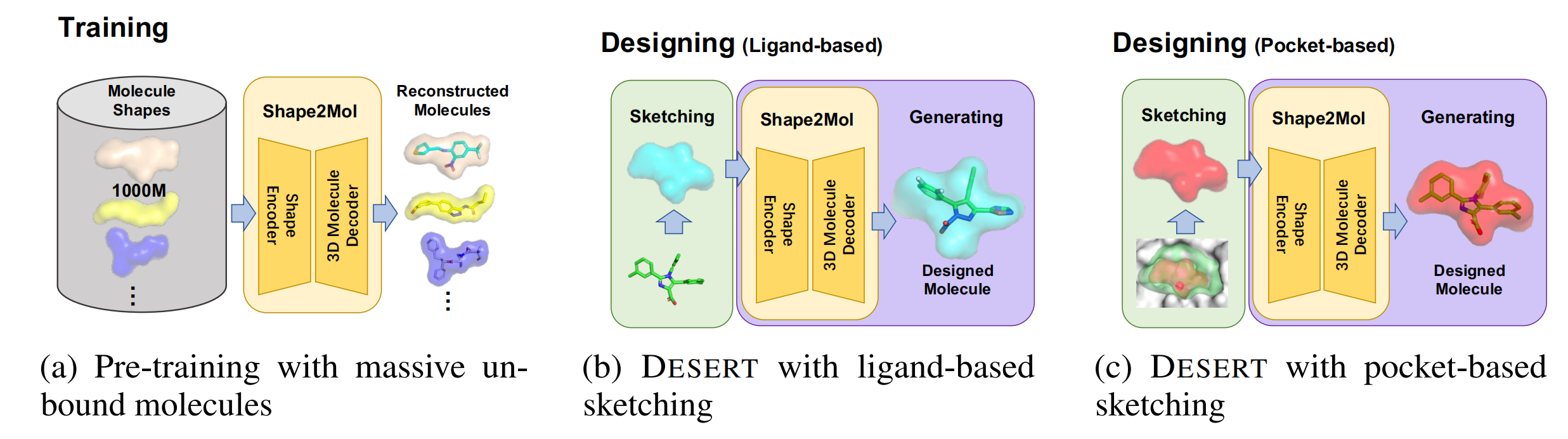
“bound drug/molecule”or “unbound drug/molecule”、molecule shape、sketching是什么?
“bound drug/molecule”or “unbound drug/molecule” For clarity, the following terms will be used throughout this study: “bound drug/molecule” (or “unbound drug/molecule”) refers to the drug/molecule th
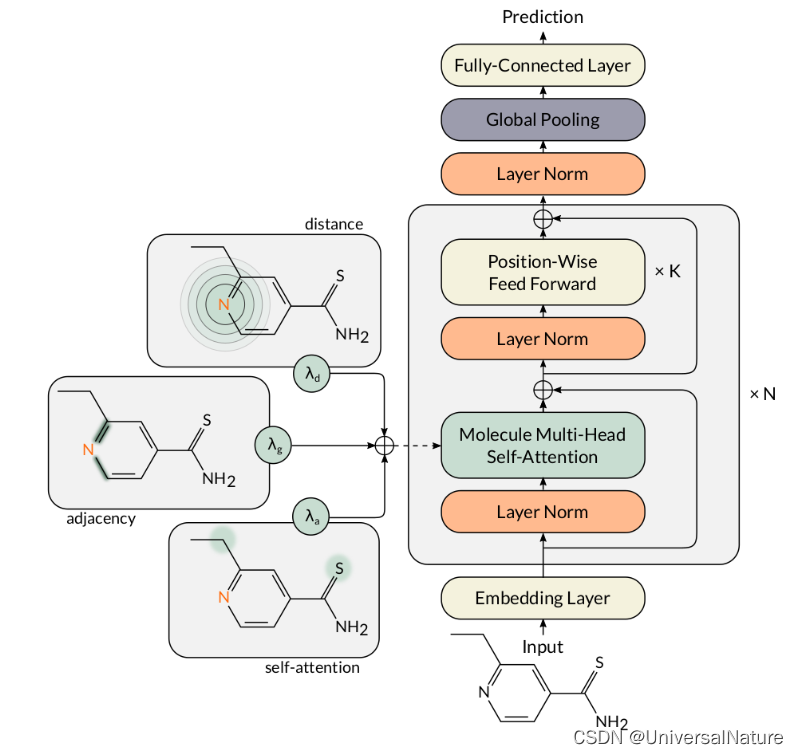
Molecule Attention Transformer(一)
应用Transformer框架对分子属性进行预测,代码:MAT,原文:Molecule Attention Transformer。变量名,函数名很多来自The Annotated Transformer,在《深入浅出Embedding》一书中也做了讲解。本文主要从实例运行开始一步步看代码具体内容,整体模型如下: 文章目录 1.数据准备1.1.load_data_from_df1.
【AAAI 2021】Multi-constraint Molecule Sampling for Molecule Optimization
多限制分子采样分子优化 Multi-constraint Molecule Sampling for Molecule Optimization 摘要 挑战:face difficulties in simultaneously optimizing multiple drug properties. 方法:To address such challenges, we propose
Molecule Attention Transformer(二)
应用Transformer框架对分子属性进行预测,代码:MAT,原文:Molecule Attention Transformer。变量名,函数名很多来自The Annotated Transformer,在《深入浅出Embedding》一书中也做了讲解。本文主要从实例运行开始一步步看代码具体内容,整体模型如下: 文章目录 2.模型构建2.1.make_model & run2.2.
Multi-Objective Molecule Generation using Interpretable Substructures
2021/7/22 摘要:药物设计问题旨在发现满足目标化学性质要求的新分子结构,当面对多性质优化问题时这项工作便变得极其困难。本文提出了一种解决这种复杂性的方法,通过一个子结构语料库来组成分子,称之为分子基本准则,这些子结构是从分子中确定的可能对某个属性有贡献的子结构,这样的方法属于“基于分子碎片”的设计方法。 实现自动生成分子的模型包括两个步骤: 在性质预测模型的帮助下提取出用于构建分子的分
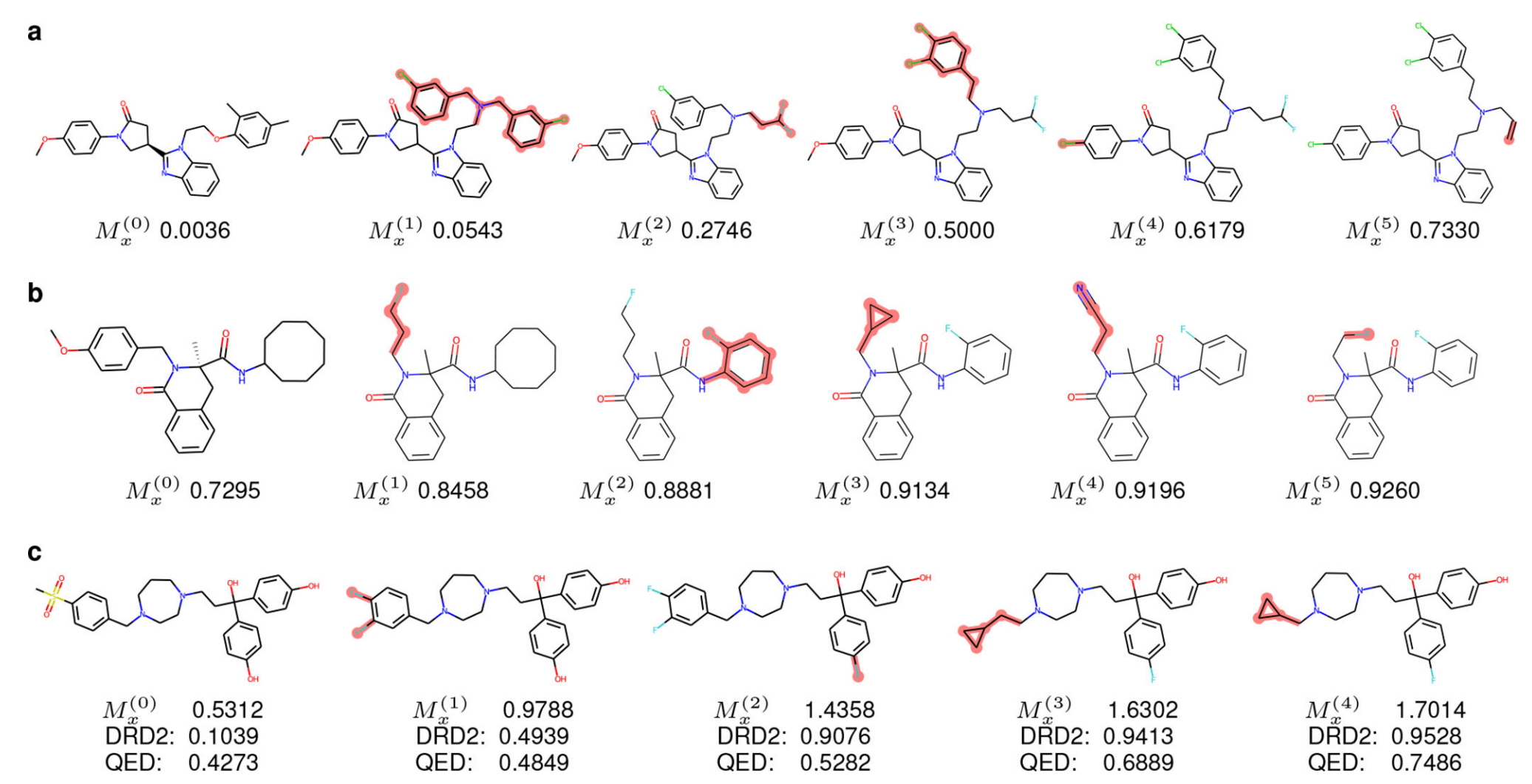
论文:A Deep Generative Model for Molecule Optimization via One FragmentModification
A Deep Generative Model for Molecule Optimization via One FragmentModification 利用变分自编码器的生成模型 训练:输入两个对比的分子,生成断点,需要删除增加的片段 生成:从隐向量空间中采样 z , 和Mx一起计算出断点,和需要删除或添加的片段。 We first quantified the difference
A Deep Generative Model for Molecule Optimization via One FragmentModification 1 【整体理解】
Modof:A Deep Generative Model for Molecule Optimization via One FragmentModification 基于单片段修改的分子优化深度生成模型 paper and supplement metrics : A deep generative model for molecule optimization via one fragm
Paper reading (三十七):Generating focused molecule libraries for drug discovery with RNN
论文题目:Generating focused molecule libraries for drug discovery with recurrent neural networks scholar 引用:203 页数:12 发表时间:2017.12 发表刊物:ASC(American Chemical Society) Central Science 作者:Marwin H. S.
12月16日Molecule在Github、Gitee正式开源
随着全球开源生态的持续性发展,开源项目数量呈现指数级的增长,并逐渐覆盖全栈技术领域。袋鼠云数栈技术开源团队一直秉承着“源于开源 回馈开源”的理念,坚持以技术为核心,开源开放、不断地将数字化技术红利回馈社区,为大家分享成果。截至目前数栈开源技术团队已在Github上开源了FlinkX、FlinkStreamSQL、EasyAgent等项目,取得了超过5000+star数和2000+Fork数。 作
CORE: Automatic Molecule Optimization using Copy and Refine Strategy(论文解读)
CORE: Automatic Molecule Optimization using Copy and Refine Strategy(AAAI 2020) CORE:使用复制和改进策略自动进行分子优化 CORE: Automatic Molecule Optimization Using Copy & Refine Strategy | Papers With Code paired d
A Deep Generative Model for Molecule Optimization via One FragmentModification 2 【encoder和decoder详解】
Decoder整体流程: DSP预测在哪个节点应该断开,并预测在该断开位点 n d n_d nd 是否应该有一个child node 或者多个 child node n c n_c nc 再预测这些child node的类型 x c x_c xc连接断开位点 n ∗ ( t ) n^{*}(t) n∗(t) 和这些 child node n c n_c nc【连接的键的选择、连接
Molecular Simulation:Chain-Molecule Sampling Techniques
转载于:https://www.cnblogs.com/Simulation-Campus/p/8794374.html
Molecule Property Prediction based on Spatial Graph Embedding | 基于空间图嵌入的分子性质预测 | Graph | GNN
文章在2019.8发表在JCIM上,化学二区 1 Introduction 1.1 背景 目前深度学习方法 过度依赖手工设计的特征,这可能会限制模型 潜在表征 的搜索空间,那么如何设计卷积方法 来提高分子性质预测的性能 是需要解决问题。 1.2 本文工作 本文提出了一种 卷积空间图嵌入层(C-SGEL),使卷积运算能够应用于 分子图数据,将多个嵌入层堆叠起来 构造 卷积空间图嵌入网络(C
CoAtGIN: Marrying Convolution and Attentionfor Graph-based Molecule Property Prediction
目录 摘要 1 介绍 2 方法 A. 预赛(Preliminaries) B.局部卷积 C.全局卷积 摘要 基于计算策略的分子性质预测在药物发现和设计过程中起着关键作用,如DFT。然而,这些传统的方法耗时长,劳动强度大,不能满足生物医学的需要。由于深度学习的发展,出现了许多用于分子表示学习的图神经网络(GNN)变体。然而,现有的性能良好的基于图的方法具有大量的参数或轻模
![2024-python字典-报错compounds.iloc[0].molecule_structures.keys()](/front/images/it_default.jpg)