本文主要是介绍【AAAI 2021】Multi-constraint Molecule Sampling for Molecule Optimization,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
多限制分子采样分子优化
Multi-constraint Molecule Sampling for Molecule Optimization
摘要
挑战:face difficulties in simultaneously optimizing multiple drug properties.
方法:To address such challenges, we propose the MultI-constraint MOlecule SAmpling (MIMOSA) approach, a sampling framework to use input molecule as an initial guess and sample molecules from the target distribution.
具体来说:MIMOSA首先对两个属性不可知图神经网络(GNN)进行预训练,用于分子拓扑和子结构类型预测,其中子结构可以是原子或单环。对于每次迭代,MIMOSA使用GNN的预测并使用三种基本的子结构操作(添加,替换,删除)来生成新的分子和相关的权重(associated weights)。权重可以编码多个约束,包括相似性约束和药物性质约束,在此基础上我们选择有希望的分子进行下一次迭代。
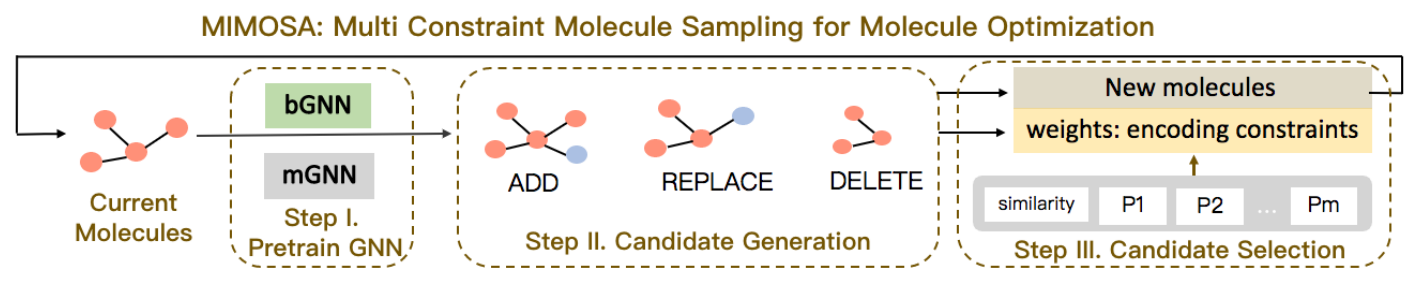
算法示意图:
1、(Pretrain GNN),MIMOSA使用大量未标记的分子对两个图形神经网络(GNN)进行预训练,这些分子将在采样过程中使用。 MIMOSA预训练两个属性不可知(property-agnostic)的GNN(bGNN、mGNN)用于分子拓扑和子结构类型预测。
2、(Candidate Generation,生成很多候选分子),MIMOSA根据 bGNN、mGNN 的预测结果,采用三种基本的子结构操作(ADD、REPLACE和DELETE)生成新的候选分子并评分。
3、 (Candidate Selection,从步骤 II 生成的新分子候选中使用MCMC sampling选择出最有潜力的分子进行下一轮的迭代),MIMOSA为新分子的每个性质分配权重。权重可以理解为:权重编码为多个约束,包括相似性约束和药物性质约束,在此基础上我们接受有希望的分子进行下一次迭代。MIMOSA对分子进行迭代编辑,可以高效地绘制分子样本。【其实就是将step2得到的分子求出性质,然后根据性质的重要性不同,为每个性质分配不同的权重,最后求出综合性质最优的进行下一次的迭代】
1、用于子结构类型和分子拓扑预测的预训练GNN(Pretrain GNN)
为了准确地表示分子,在大分子数据集上预先训练分子嵌入。用图表示分子,其中每个子结构都是一个节点,作者建立了两个基于GNN的预训练任务来辅助分子修饰,这两个GNN将评估每个子结构受分子图中所有其他子结构制约的概率。选择训练两个单独的GNN是因为存在很多未标记分子样本,而且这两个任务在本质上差异很大。两个GNN模型中,一个用于子结构类型预测,称为mGNN,另一个用于分子拓扑预测,称为bGNN。
mGNN模型(子结构类型预测):以多类分类为目标,用于预测掩码节点的子结构类型。mGNN模型根据其他子结构和连接来输出单个子结构的类型。用一个特殊的掩码指示器单独掩码子结构。
bGNN模型(分子拓扑预测):旨在对分子拓扑结构进行二分类预测。bGNN的目标是预测节点是否会扩展。
2、通过子结构修改操作来生成候选对象(Candidate Generation)
借助于mGNN和bGNN定义子结构修饰操作,即对输入分子Y进行替换、添加或删除操作。
【采样候选分子是通过给“替换”、“添加”、“删除”不同的权重,从而用不同概率采样不同的分子,这是否可以看成一种“变异”操作?这样就可以使变异变的有意义,而不是单纯的通过概率进行变异】
生成的候选分子集合可以根据它们接受的子结构修饰的类型被分组为三个集合(three sets),即替换集合Splace、添加集合Sadd和删除集合Sdelete。MIMOSA使用MCMC的一种特殊类型Gibbs采样,用于候选分子选择。Gibbs采样算法根据其他变量的当前值,按顺序或随机顺序从每个变量的分布中生成一个实例(见算法1)。在这里,这三组分子将以不同的采样权重进行采样【这些权重代表了不同子结构修改类型的候选分子集合(如替换集S_replace、添加集S_add和删除集S_delete)被选择成为下一步迭代中的分子的概率。这里是通过Gibbs采样完成的采样】。它们的权重被设计为满足详细的平衡条件(Brooks et al. 2011)。
(1)采样替换Sreplace。对于“replace”操作产生的分子,采样wr中的权重由式(13)给出:
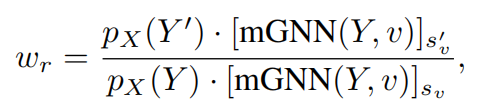
(2)采样添加Sadd。对于“加”操作产生的分子,采样的权重由等式给出 (14):

(3)采样删除Sdelete。对于这些通过“删除”操作产生的分子,采样中的权重由等式给出 (15):

3、通过MCMC采样进行候选分子选择(Candidate Selection)
算法流程:

-
输入:
- 分子 X
- 粒子数量 N
- 最大采样次数 Tmax
- 预热迭代次数 Tburnin
-
输出:
- 生成的分子集合 Φ
-
预训练GNN:
- 使用方程(6)训练mGNN
- 使用方程(9)训练bGNN
-
初始化候选集Θ为单一分子X,输出集Φ为空。
-
进行Tmax次迭代:
-
候选生成:
- 初始化候选池Ψ为空。
- 对于集合Θ中的每个分子Z:
- 通过编辑Z(如子结构操作)生成候选Z'。
- 验证Z'的有效性。
- 如果Z'有效,则将其添加到Ψ中。
-
候选选择:
- 如果当前迭代次数小于预热迭代次数Tburnin:
- 从Ψ中选择具有最高密度值的N个分子(根据方程(1))并将其加入到Θ中。
- 否则:
- 使用重要性采样从Ψ中抽取N个分子,根据方程(13)、(14)或(15)的权重,并将其加入到Θ中。
- 如果当前迭代次数小于预热迭代次数Tburnin:
-
更新生成的分子集合Φ,将Θ中的分子添加到Φ中。
-
-
迭代结束后,输出生成的分子集合Φ。
此MIMOSA算法主要通过多次迭代进行分子优化。在每次迭代中,首先基于当前的候选集合Θ生成新的分子候选,然后根据给定的条件选择新的分子并更新候选集。经过预定的迭代次数后,算法输出一个经优化的分子集合。
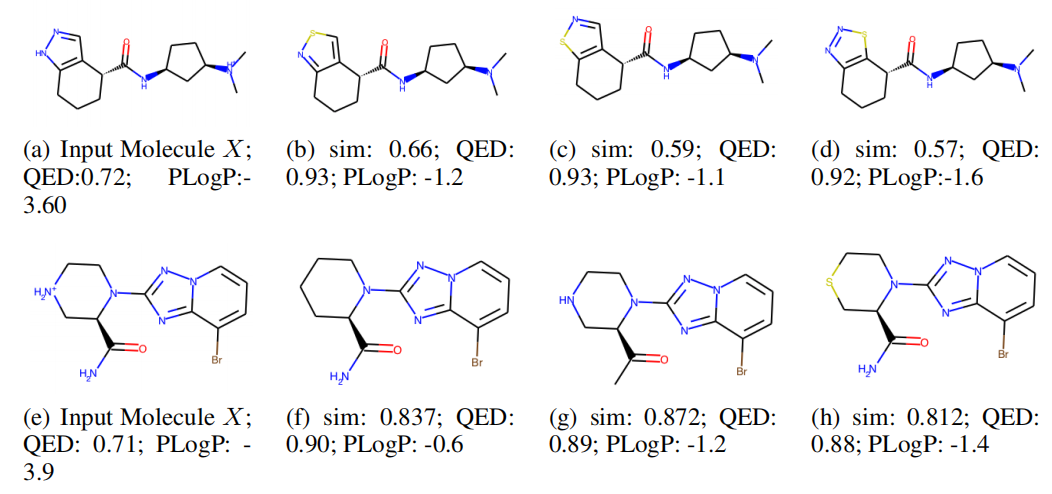
CASE
Figure 2: Exp 3. Examples of “QED & PLogP” optimization. (Upper), the imidazole ring in the input molecule:
(a) is replaced by less polar rings thiazole (b and c) and thiadiazol
(d). Since more polar indicates lower PLogP, the output molecules increase PLogP while maintaining the molecular scaffold. (Lower), the PLogP of input molecule
(e) is increased by neutralizing the ionized amine
(g) or replacing with substructures with less electronegativity (f and h). These changes improve the QED.
为了进一步探究MIMOSA如何有效改善对局部结构变化敏感的属性,例如PLogP,我们在图2中展示了两个示例。对于第一行,输入分子(a)中的咪唑环被较少极性的五元环噻唑(b和c)和噻二唑(d)所替代。由于PLogP与分子的极性相关:极性更强意味着PLogP更低。这种生成结果在保持分子骨架的同时增加了PLogP。对于第二行,通过中和离子化的胺基(g)或替换为电负性较低的亚结构(f和h),增加了输入分子(e)的PLogP。这些变化也有助于提高药物样性,即QED值。
MIMOSA: 用于分子优化的多约束分子采样
这篇关于【AAAI 2021】Multi-constraint Molecule Sampling for Molecule Optimization的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!





![[SWPUCTF 2021 新生赛]web方向(一到六题) 解题思路,实操解析,解题软件使用,解题方法教程](https://i-blog.csdnimg.cn/direct/bcfaab8e5a68426b8abfa71b5124a20d.png)
