hla专题
【Nature. Mach. Intell. 】基于Transformer的多肽-HLA I类结合预测和疫苗的新生抗原序列设计...
来源:专知本文1000字,建议阅读5分钟TransPHLA设计了Transformer衍生模型来预测pHLA的结合。 近日,国际知名期刊《Nature Machine Intelligence》在线发表了上海交通大学生命科学技术学院魏冬青团队的研究论文《A transformer-based model to predict peptide–HLA class I binding and o
HLA-A单克隆抗体参考说明书
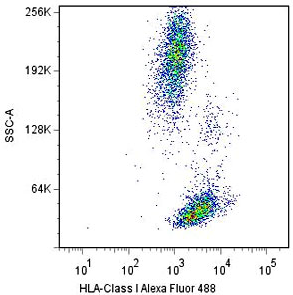
HLA-A单克隆抗体 cat#:MAB6449 描述:针对天然HLA-A的小鼠单克隆抗体。 克隆名称:W6/32 免疫原:从人类扁桃体细胞膜天然纯化的HLA-A。 牛、猫、人、非人灵长类应用:ELISA、Flow Cyt、Func、IHC Fr、IP、WB 原细胞特异性:该抗体识别所有人类有核细胞表面表达的MHC类I分子(MHC类Ia)。这种抗体也适合作为HLA组织分型和交叉配型的阳性
HLA-A单克隆抗体实验研究方案
HLA-A属于HLA I类重链同源物。该I类分子是由重链和轻链(β-2微球蛋白)组成的异二聚体。重链固定在膜上。I类分子通过提供来自内质网腔的肽在免疫系统中发挥中心作用。它们在所有细胞中表达。重链约为45kDa,其基因包含8个外显子。外显子1编码先导肽,外显子2和3编码结合肽的alpha1和Alpha2结构域,外显子4编码alpha3结构域,外显子5编码跨膜区,外显子6和7编码细胞质尾部。外显子2
KY-RTI分布仿真技术:附录2 大联邦(构建1000个成员的HLA/RTI仿真系统)
基于当前的RTI构建1000个以上成员的仿真系统在一些人看来似乎是极具挑战性的一个问题。如果你对当前的计算机技术、网络技术、仿真技术真正了解的话,其实构建这样的一个大规模仿真系统是完全可行的。本章以聊天程序为例,介绍了基于KY-RTI构建1000个仿真成员的两种方法:基于单个RTI服务器的大联邦;基于桥接程序实现多个RTI服务器的大联邦。从HLA的概念来讲,每个RTI服务器及其所有仿
Optitype进行HLA分型的尝试
Optitype 软件安装 最开始尝试使用docker,无奈悲剧的失败,发现bioconda有这个软件的,于是上conda,感觉比docker更方便呢。还有一个好处是,win10家庭版不支持docker,要想支持得修改注册表一通操作,太麻烦了。 # 下面两个命令选一就可以了conda install -c bioconda optitype conda install -c biocon
HLAreporter : HLA分型软件简介
欢迎关注"生信修炼手册"! 在前面的文章中,我们详细介绍了HLA Allel的命名格式,示意图如下 从示意图可以看出,一个HLA Allel 可以分成四个字段,在加上最后的修饰后缀,共5个字段;在定义HLA 分型结果的分辨率时,会根据分型结果的最大位数来判断,如果只给出了字段一,即血清学分类的信息,代表是2位的分型结果;如果最多给出了字段二,即对应的蛋白信息,代表是4位的分型结果;如果最多给出
HLAminer:根据NGS数据确定HLA分型结果
欢迎关注"生信修炼手册"! PCR-SBT方法是世界卫生组织WHO推崇的HLA 分型的金标准,其实就是指的直接测序,无论是WGS, WES, RNA_seq 数据都可以。近几年来涌现了很多的软件,支持从NGS测序数据直接确定HLA Allel, HLAminer 就是其中之一。 官网如下: https://github.com/warrenlr/HLAminer 安装过程如下: wget htt
HLA-VBSeq:对全基因组数据进行HLA分型
欢迎关注"生信修炼手册"! HLA-VBseq 利用全基因组测序的数据,可以提供8位的HLA分型结果,其文献链接如下 https://bmcgenomics.biomedcentral.com/articles/10.1186/1471-2164-16-S2-S7 在该文献中,利用30X的全基因组数据,对HLA-VBSeq, PHLAT, HLAminer这3款软件的分型结果进行了评估,准确
使用HLAscan进行HLA分型
欢迎关注"生信修炼手册"! HLAScan是由韩国的科研团队开发的一款HLA分型工具,可以处理WGS, WES和目标区域捕获测序的数据,将reads与IMGT/HLA数据库中的reads进行比对,然后确定HLA的基因型信息。 软件的开发团队利用测试数据,评估了以下几个软件的分型效果 可以看到HLAscan能够提供可靠的2位和4位分型的结果。官网链接如下 http://www.genomeko
HLAforest:使用RNA-seq数据进行HLA分型
欢迎关注"生信修炼手册"! HLAforest是一款针对RNA_seq数据进行HLA分型的软件,github地址如下 https://github.com/FNaveed786/hlaforest 安装过程如下 git clone https://github.com/FNaveed786/hlaforest 当我们把源代码下载到本地之后,还需要修改以下几个配置文件中的内容 1. 修改con
seq2HLA:利用RNA_seq数据进行HLA分型
欢迎关注"生信修炼手册"! 对于不同的HLA Allel来说,exon2和exon3 序列的差异性尤为明显,很多的HLA 分型软件都会根据这部分序列,整理出HLA Allel序列参考数据库。 seq2HLA也采用了类似的策略,通过HLA不同Allel的exon2和exon3的序列,整理了一份HLA参考数据库,通过将RNA_seq的reads与该数据库比对,确定HLA分型结果,原理示意图如下 迭
娱乐版HLA分型网页升级了
之前做了一个简单的网页,使用23andme格式+snp2hla软件获得hla分型数据,当然准确性不咋的,也就玩玩,上线后为大约100+人提供了服务,这是伯值得骄傲的事,因为第一次能为大家提供服务。代码我是放在了gitHub的,数据是脚本处理完后自动删除。看网页是不是有点眼熟,这是谷歌中国网页框架,直接搬来的。 2020.6.20更新了合并后的参考数据集,据论文说准确度有一定程度提高,欢迎测试
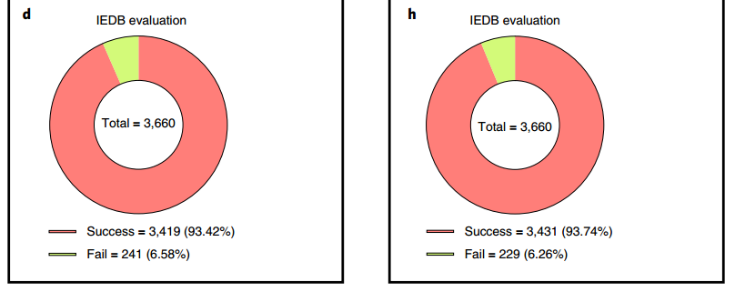
Nat. Mach. Intell 2022 | TransMut+:基于transformer的用于预测peptide–HLA class I binding 并优化疫苗设计中的突变多肽
论文标题:A transformer-based model to predict peptide–HLA class I binding and optimize mutated peptides for vaccine design 论文地址:A transformer-based model to predict peptide–HLA class I binding and opti