本文主要是介绍蛋白质治病突变的计算方法(三),希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
3 用于识别致病突变的特征
文献中使用了几种特征来识别蛋白质中的致病突变。它们大致分为三类:(1)序列,(2)结构和(3)网络,以及它们的组合。图1说明了这三组中的一些重要属性。

图1 用于识别致病突变和热点的重要特征。
基于氨基酸序列的特性包括理化特性、二级结构、位置特异性得分矩阵(PSSM)、特异性基序(motifs)和保守性得分。基于结构的性质包括界面分布(interface profiles)、残基的位置在核心和表面、相对溶剂可及面积(RSA)、体积、氢键供体和受体以及统计势能(statistical potentials)。基于网络的特征主要集中在分子相互作用网络、度、介数(betweenness)、紧密度(closeness)、特征向量和聚类系数等方面(eigen vector and clustering coefficient)。
3.1 基于序列的性质
3.1.1 理化性质
Gromiha等人(1999)收集了一组49种氨基酸特性,它们代表了物理、化学、构象(conformational)和能量特性。这些特性被广泛用于理解蛋白质的结构和功能,以及预测突变的后果。AAindexis是另一个数据库,它包含氨基酸各种理化和生化性质的数值指数。(没看懂这个数据库,但是链接是:AAindex: Amino acid index database (genome.jp))
3.1.2 预测的二级结构和溶剂可及性
蛋白质中每个残基的二级结构和可及表面面积(ASA)被用作识别致病突变的特征。二级结构主要有螺旋、股(stand)和盘管(coil)。一个残基的ASA分为暴露型(ASA > 25%)和埋型(ASA < 25%)。常用的基于序列的方法预测二级结构和溶剂可及性,包括JPred4,NetSurfP,SPIDER2 和SARPred。
3.1.3 Motifs
motif代表氨基酸序列中的特定模式,由二肽中相邻残基的连续出现(XM;X:任何残基和M:感兴趣残基)、三肽等,以及在它们之间引入间隙(例如X*M;*:差距)。motif提取细节如图2所示。
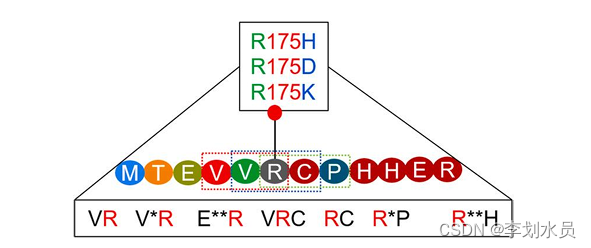
图2 从蛋白质序列中提取不同的基序以识别致病突变和热点残基。
这些motifs的偏好用“odds score”来解释,它被定义为特定基序在疾病易感位点和中性位点(突变)中的比例。它由以下公式计算:

其中NdP(disease-prone)和NnP(neutral)分别表示在疾病易感位点和中性位点(或突变)出现特定的二肽或三肽基序。Nd和Nn是数据集中易患病位点和中性位点(或突变)的总数。
(公式总体来看像是易感/中性,然后每个小的又是二/三肽比所有)

3.1.4 氨基酸组成
氨基酸组成表示从热点残基(或致病突变)开始的序列在选定的窗口长度内氨基酸残基的出现情况。窗口长度为3的残基包括中心残基,并且在中心残基的两侧各有一个(图2中的R175包括VRC)。一般情况下,计算组成时考虑3 ~ 21个残差的窗口长度。它计算公式是:
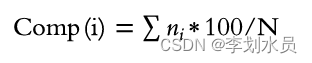
其中,i表示20种的氨基酸,ni表示其中i这种氨基酸的数量,N表示所有氨基酸的数量。
这篇关于蛋白质治病突变的计算方法(三)的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!