本文主要是介绍数学建模-最优包衣厚度终点判别法-二(K-Means聚类),希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
💞💞 前言
hello hello~ ,这里是viperrrrrrr~💖💖 ,欢迎大家点赞🥳🥳关注💥💥收藏🌹🌹🌹
💥个人主页:viperrrrrrr的博客
💥 欢迎学习数学建模算法、大数据、前端等知识,让我们一起向目标进发!
基于近红外光谱的肠溶片最优包衣厚度终点判别法
包衣是将片剂的外表面均匀地包裹上一层衣膜的过程,旨在控制药物在胃肠道中的释放部位和速度,遮盖苦味或不良气味,防潮、避光,改善外观等。然而,包衣膜太薄或太厚都不利于药效,并且包衣终点的判断方法目前存在一定的难度。近红外光谱技术(NIRS)是一种高效、无需试剂、无污染的分析方法,通过近红外光谱仪、化学计量学软件和应用模型,能快速、简便地实现多组分检测。为实现包衣终点的准确判断,对数据进行分析并完成以下问题:
问题一:对药品在不同包衣时间段包衣片剂的近红外光谱进行特征峰提取,选择具有有效信息的波长片段,即波长选择。
问题二:分析药品包衣厚度分类规律,建立合适的模型对药品包衣不同厚度进行划分,给出方法及结果,并进行灵敏度分析。
问题三:对于不同的包衣厚度,通过建立模型分析包衣之间的关联性,判别出最优的包衣厚度。
我们本次主要解决问题二。
对肠溶片包衣厚度进行分类,进一步探究包衣厚度的分类规律,分析其分类标准。包衣技术要求对药片进行多次包衣和对包衣终点并没有明确指标,本问题通过在提取的有效特征峰基础上,根据聚类汇总分析各聚类类别的频数:此步骤是对第一步的补充,旨在了解每个聚类中样本的数量和分布。这有助于更好地理解每个聚类的特征和代表性。便于问题三对肠溶片包衣终点判别作为数据基础。
针对问题二,本文通过使用聚类算法(K-Means),通过比较样本与聚类中心的距离,了解样本与该聚类的相似性或者距离程度。我们将metrics库引入python得到轮廓系数进一步确定K值的准确性。在随着聚类数目的增加,聚类内部的误差会减小,但是减小的速度会逐渐变慢,直到达到一个“肘部”点。在得到较为精确的K值后在SPSS中进行K-Means聚类分析,最终得到肠溶片的分析结果。
聚类中心是聚类算法中的重要概念,代表了该聚类的主要特征和中心思想。聚类中心坐标可以用于分析各样本与中心点的距离,我们通过比较样本与聚类中心的距离,可以了解样本与该聚类的相似性或距离程度[3]。
将metrics库引入python得到轮廓系数进一步确定K值的准确性。在随着聚类数目的增加,聚类内部的误差会逐渐减小,但是减小的速度会逐渐变慢,直到达到一个“肘部”点。
通过肘部法则SSE计算误差平方和公式如下:
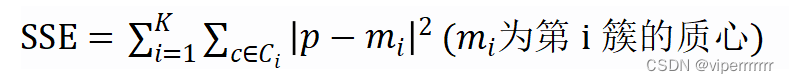
(2)
肘部的轮廓系数计算公式如下:

(3)
据图4可得,聚类中心个数为3时即K=3时聚类分析模型性能达到最佳。
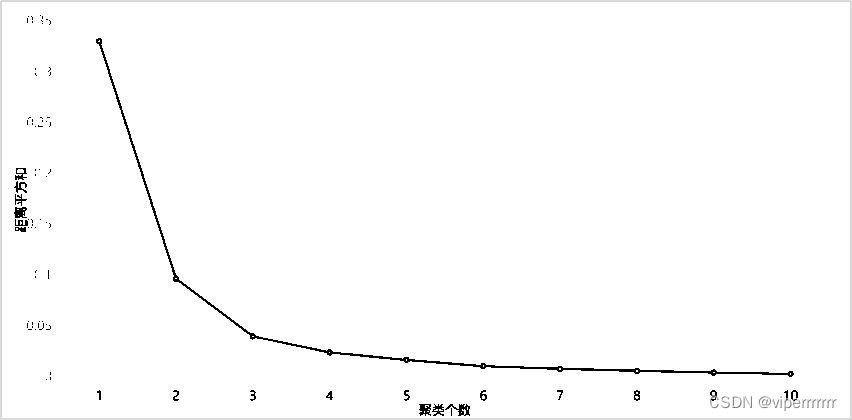
图4 聚类中心个数
确定聚类中心个数后,进行K-means聚类分析之前,先将基因表达矩阵标准化,以消除尺度差异对聚类结果的影响,再遵循如图5所示的步骤进行K-means++聚类分析。
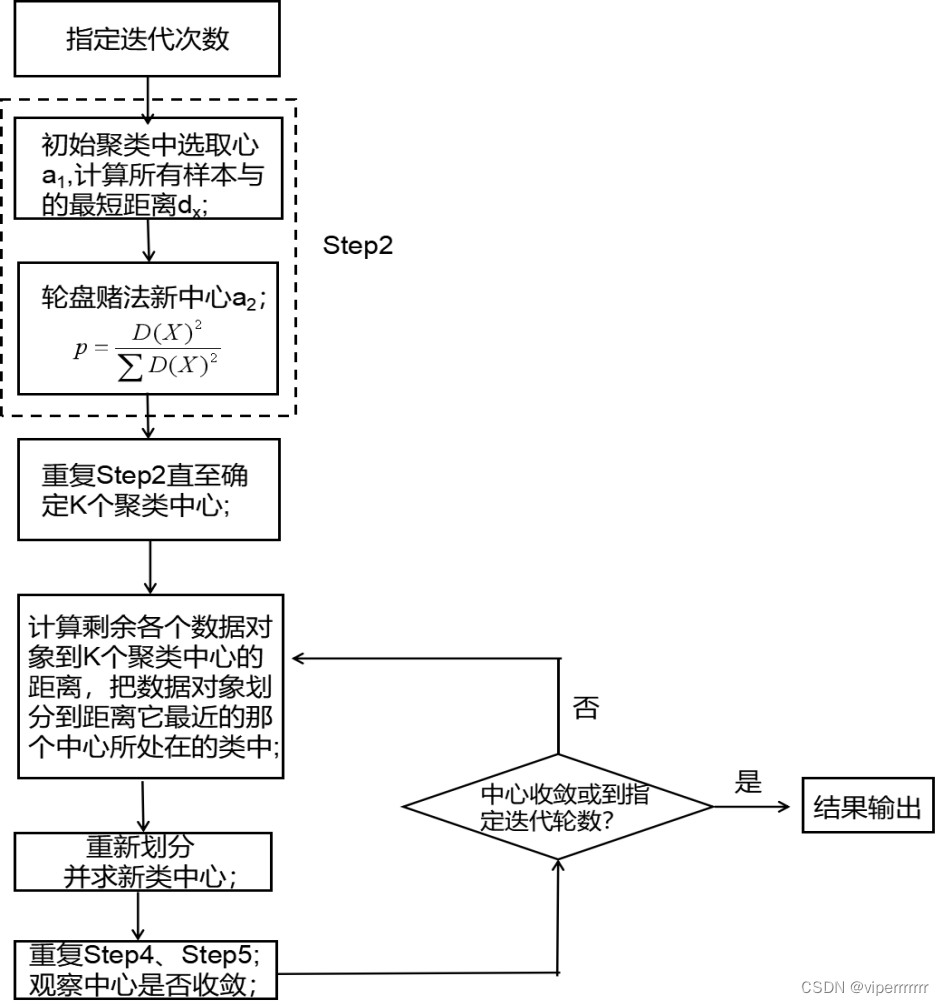
图5 K-means++聚类算法流程图
通过上述方法得到较为精确的K值(如图4)后再通过K-Means聚类分析,得到最终的分类结果如表1所示。
表1 聚类表
| 波长 | 聚类类别(平均值+/-标准差) | F | ||
| 类别1(n=47) | 类别3(n=35) | 类别2(n=18) | ||
| 3795.38 | 0.953±0.015 | 0.904±0.013 | 0.851±0.016 | 类别1(n=47) |
| 3803.1 | 0.952±0.015 | 0.903±0.013 | 0.85±0.016 | 类别1(n=47) |
通过表1可得定量字段的差异性,其中得到波长为3795.38和3803.1两组数据之间都呈现显著差异,在进行K-means聚类分析时,得到的数据类别之间也存在显著的差异。
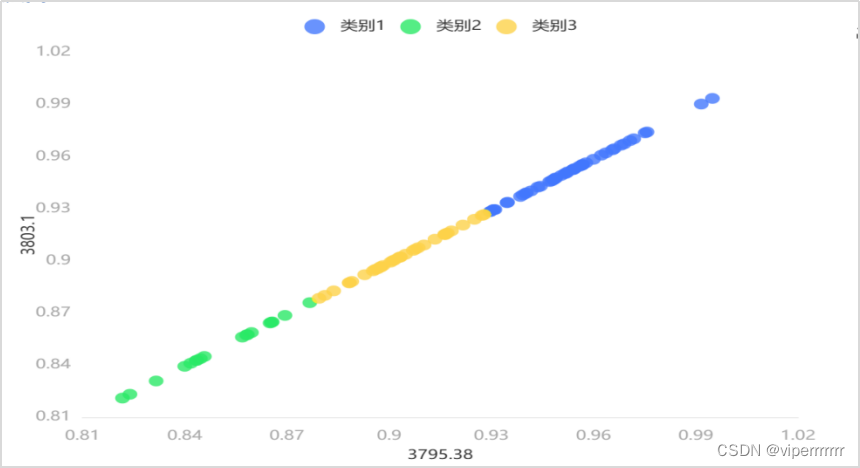
图6 聚类散点图
再在python中引入绘图包,引入使用K-Means聚类分析得到的聚类中心坐标,得到三种类别数据的聚类散点图,如图6所示。
表2 评价系数汇总表
| 评价系数 | ||
| 轮廓系数 | DBI | CH |
| 0.611 | 0.451 | 346.533 |
再对表二分析可得,DBI指标数值为0.451,代表簇间距离大,聚类效果好。CH指标数值为346.533,代表类间中心点与数据集中心点距离大,也表示聚类效果好。通过以上两个评定指标,可以验证建立的聚类分析模型较为可靠。
这篇关于数学建模-最优包衣厚度终点判别法-二(K-Means聚类)的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!








