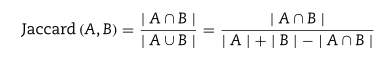本文主要是介绍基于特征融合与注意力机制的药物互作模型:MDF-SA-DDI,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
论文题目:MDF-SA-DDI: predicting drug–drug interaction events based on multi-source drug fusion, multi-source feature fusion and ransformer self-attention mechanism
论文来源: Briefings in Bioinformatics,00(00), 2021,1–13
网址:https://academic.oup.com/bib/advance-article-abstract/doi/10.1093/bib/bbab421/6406700?redirectedFrom=fulltext
代码: GitHub - ShenggengLin/MDF-SA-DDI
主要内容:使用多源药物融合,多源特征融合和Transformer进行药物互作预测
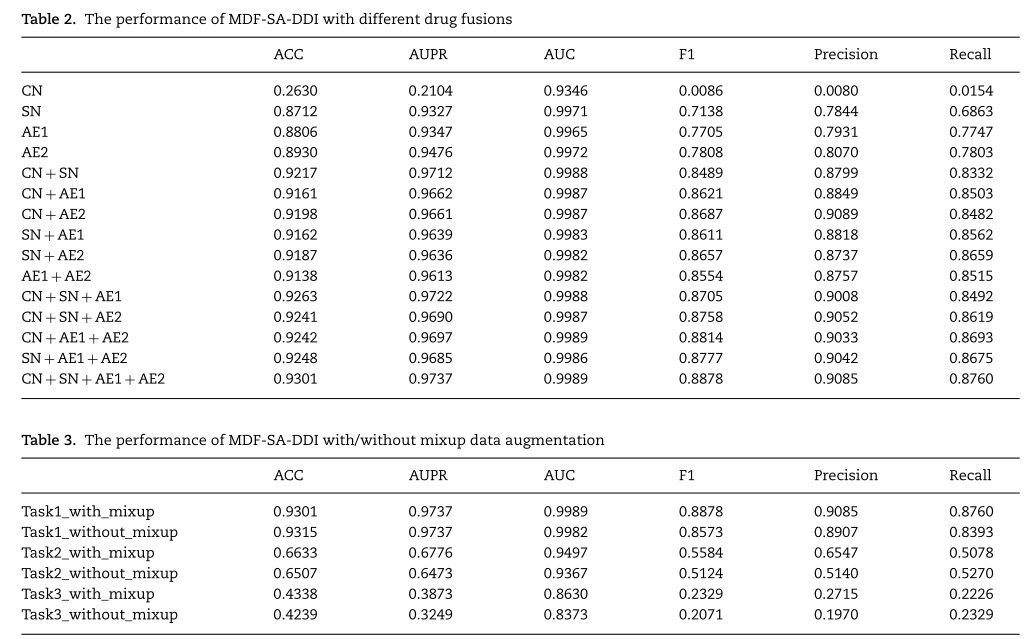
- 四个网络+多头自注意力机制
- 孪生网络
- 卷积神经网络
- 两个自动编码器
药物互作相关
- 为什么要进行药物互作的研究
- 大多数人类疾病病理复杂,对单一的药物都有抗药性。使用联合药物治疗可以有效提高药效,降低耐药性。
- 但是,不同的药物之间可能会发生相互作用(drug–drug interaction, DDI),可能会导致不良事件。
- 而每两种药都进行试验验证过于昂贵,因而使用计算机进行模拟的研究显得比较重要了。
- 主流方法:(1)基于机器学习的方法;(2)基于深度学习的方法;(3)基于矩阵分解的方法;(4)基于网络扩散的方法;(5)基于集成学习的方法;(6)基于文献或文本挖掘的方法。
- 以往方法的问题
- 大多基于深度学习技术,将两个药物向量连接在一起预测DDI事件,而没有尝试其他方法来融合药物对的信息
- 大多数方法在预测已知药物之间未观察到的相互作用方面表现良好,很难预测新药之间未被观察到的相互作用
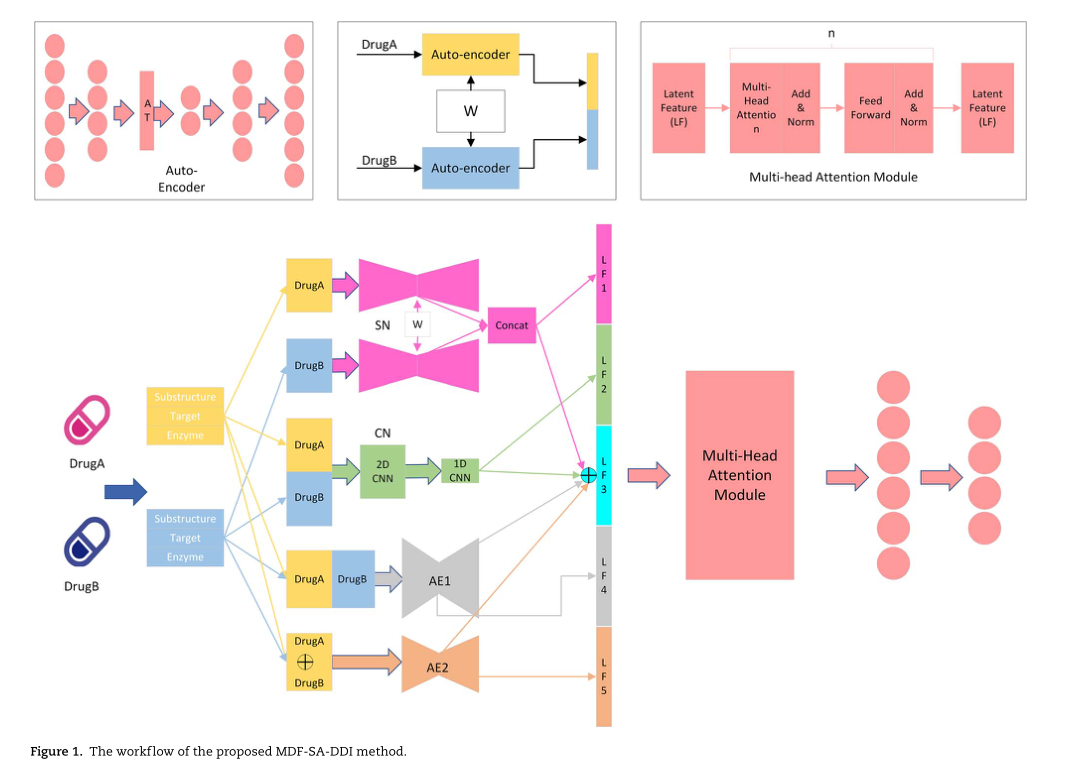
提出方法:MDF-SA-DDI
- 具体内容
- 孪生网络(Siamese Network,SN),将两种药物的特征输入其中,得到的两个向量作为药物对的新特征。需要注意的是,这两个向量表示的是每个药物各自的特征
- 卷积神经网络(CN),将两个药物特征拼接,然后将其进行卷积,得到药物对的潜在特征
- 自动编码器(AE1),将两个特征向量拼接起来,然后通过自动编码器得到特征
- 自动编码器(AE2),将两个特征向量按照元素相加,然后通过自动编码器得到特征
- 自注意力层:将上述四个向量,过一个多头自注意力模块进行特征融合
- 全连接:将上述得到的特征过一个全连接层,得到最后的药物互作事件
- 由于数据集特征过多,直接使用容易得到维数灾难,基于类似药物可能与相同的药物相互作用的假设,文章直接使用了Jaccard近似
实验及结果
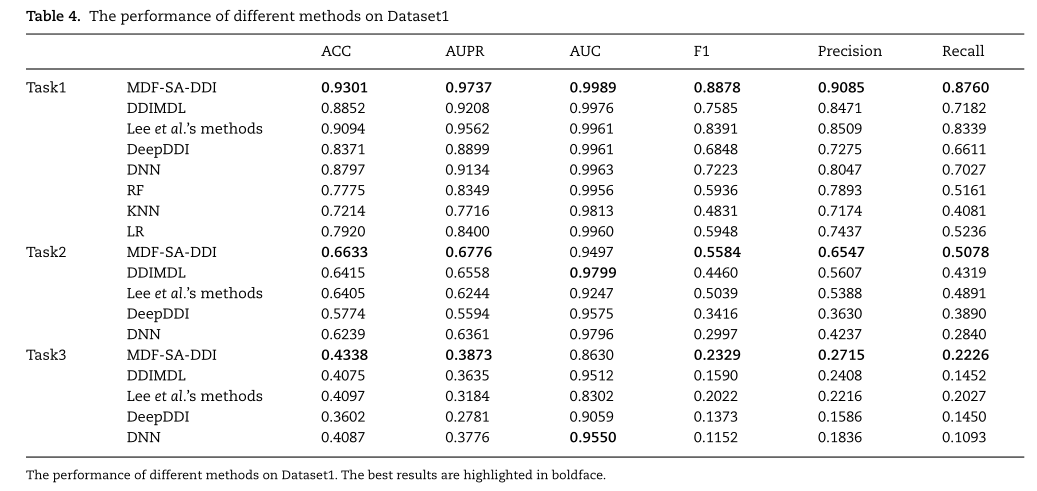
- 三种实验,相应任务中的新药表示在训练集中缺失,但在测试集中存在
- 预测已知药物之间未观察到的相互作用事件(任务1)
- 预测已知药物与新药之间的相互作用事件(任务2)
- 预测新药之间的相互作用事件(任务3)


这篇关于基于特征融合与注意力机制的药物互作模型:MDF-SA-DDI的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!