本文主要是介绍四、肺癌检测-数据集准备 dsets.py文件,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
一、目标
数据集准备需要完成以下几个工作:
1. 读取annotations.csv内容;
2. 读取candidates.csv内容;
3. 构造Ct类,用于根据输入的series_uid,获取该uid的CT数据的信息。
4. 构造Dataset类,用于加载数据集。
二、要点说明
1. SimpleITK库
读取和解析CT结果的【mhd】文件需要使用SimpleITK库,可通过【conda install simpleitk】命令安装。
其中主要用到以下几个函数说明如下:
# 读取mhd格式文件,并返回一个mhd对象。
ct_mhd = SimpleITK.ReadImage(path)# 获取ct_mhd对象的XYZ坐标相对于IRC坐标的原点偏移,类型为1x3数组。
origin_xyz = ct_mhd.GetOrigin()# 获取ct_mhd对象每个体素在xyz坐标轴的大小,用于转换为IRC坐标时进行尺度缩放。类型为1x3数组
vxSize_xyz = ct_mhd.GetSpacing()# 获取ct_mhd对象从XYZ转换为IRC坐标时的空间转换矩阵,类型为3x3的eye数组
direction_a = ct_mhd.GetDirection()).reshape(3, 3)2. functools库
代码中用到了functools库,用于将某些函数的结果缓存到内存中。
@functools.lru_cache(1):代表1次缓存。用于存放在需要缓存的函数定义的代码的开头。意义是:如果该函数之前已经输入过相同的参数,下一次再输入相同参数时,函数直接从缓存调用结果,而不会从新执行函数内部代码。
3. diskcache库
代码中用到了diskcache库,用于将CT数据解析后缓存到磁盘中,使用缓存可以较大的提高训练时数据加载速度。库的使用可参考相关文章:
【编程】Python : diskcache 本地缓存持久化,一行代码_哔哩哔哩_bilibili
Python 爬虫进阶篇——diskcache缓存_十先生(公众号:Python知识学堂)的博客-CSDN博客_diskcache python
4. CT文件信息
4.1 csv文件
annotations.csv: 记录实际结节。文件结构: uid, x, y, z, diameter candidates.csv: 记录候选结节。文件结构: uid, x, y, z, class
注意:两个文件中,相同的uid对应的xyz坐标可能有偏差,要将偏差大于半径的一半(即diameter/4)的数据的diameter强制为0,即认为这个结节异常,不处理。
5. XYZ、IRC坐标轴
5.1 坐标轴方向
CT数据中,有XYZ坐标轴,训练时需要转换为IRC坐标轴,两个坐标轴分别对应着:
xyz:各坐标轴正的方向指向的人体的方向为为:
x:左手,y:后背,z:头顶
irc:各坐标轴正的方向指向的人体的方向为为:
i:头顶,r:后背,c:左手
其中i-index,r-row, c-column
简记为:xyz-左后上,irc-上后左
5.2 坐标轴转换
5.2.1 irc转xyz
step1:将irc矩阵翻转为cri
step2:用体素大小缩放cri坐标
step3:缩放后的cri坐标与空间矩阵叉乘得到xyz坐标
step4:xyz坐标加上原点偏移量。
def irc2xyz(coord_irc, origin_xyz, vxSize_xyz, direction_a):"""irc坐标转为xyz坐标step1:将irc矩阵翻转为cristep2:用体素大小缩放cri坐标step3:缩放后的cri坐标与空间矩阵叉乘得到xyz坐标step4:xyz坐标加上原点偏移量。:param coord_irc: irc坐标:param origin_xyz: irc坐标相对于xyz的坐标偏移:param vxSize_xyz: 体素在xyz尺度的大小:param direction_a: 空间矩阵:return: """cri_a = np.array(coord_irc)[::-1]origin_a = np.array(origin_xyz)vxSize_a = np.array(vxSize_xyz)coords_xyz = (direction_a @ (cri_a * vxSize_a)) + origin_a# coords_xyz = (direction_a @ (idx * vxSize_a)) + origin_areturn XyzTuple(*coords_xyz)5.2.2 xyz转irc
def xyz2irc(coord_xyz, origin_xyz, vxSize_xyz, direction_a):origin_a = np.array(origin_xyz)vxSize_a = np.array(vxSize_xyz)coord_a = np.array(coord_xyz)cri_a = ((coord_a - origin_a) @ np.linalg.inv(direction_a)) / vxSize_acri_a = np.round(cri_a)return IrcTuple(int(cri_a[2]), int(cri_a[1]), int(cri_a[0]))
5.3 CT数据单位
CT文件中数据单位为HU(HounsField Units,亨氏单位)。其中人体各组织的HU值水平为:
空气:-1000HU,约0g/cm3
水:0HU,约1g/cm3
骨骼:1000HU,约2~3g/cm3。
因此超出-1000HU到1000HU外的数据并不是我们需要关心的数据,可强制转换为限值。
5.4 体素、结节概念
体素:可理解为CT扫描后得到的三维切片矩阵中所对应的一个点(像素),即切片后最小的人体组织,接三维的立体像素。
结节:可能为恶性也可能是良性,CT扫描后可根据体素的尺寸,结节中心坐标,结节直径截取出结节所对应的坐标值已经HU值。
| 良性结节和恶性结节的特征区别 | ||
| 特征 | 良性 | 恶性 |
| 生长速度 | 迅速 | 缓慢 |
| 查体表现 | 软,活动度大 | 硬,活动度小 |
| 超声检查 | 边界清晰,与组织分解明显 | 边界不清晰,与组织分解不明显 |
| 形态 | 光滑,圆 | 不规则,纵横比>1,直立生长 |
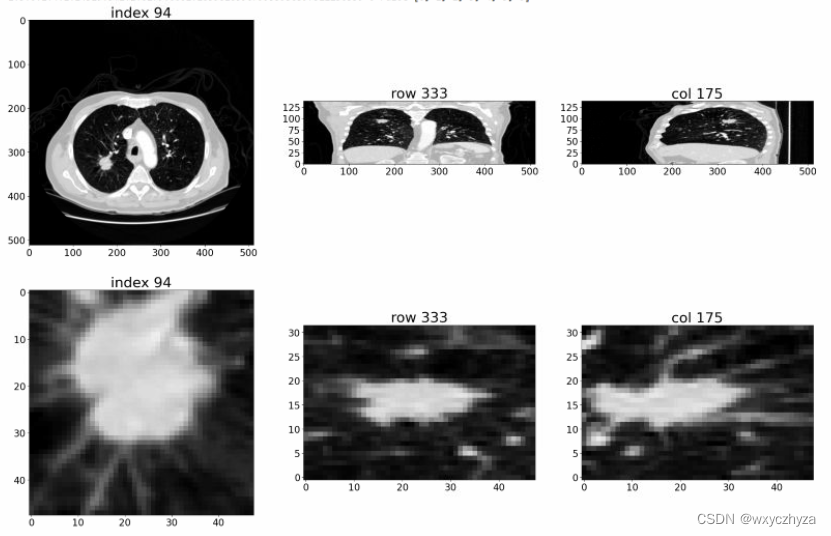
6. 数据可视化
下图第一行是对CT文件中,三维CT矩阵用不同维度索引下的结果;
下图第二行是对某个结节中,三维结节矩阵用不同维度索引下的结果。
更多可视化内容可参照原书代码的ipynb文件。
三、函数说明
1. getCandidateInfoList
candidateInfo_list = getCandidateInfoList(requireOnDisk_bool=True)
返回candidates.csv文件对应的list,其中每个元素为名称为candidateInfoTuple的元组,元组有如下节点:
class, diameter, id, xyz
2. Ct类
属性如下:
CT.hu_a:以HU为单位的三维array,存储的是CT的所有体素数据。
CT.origin_xyz:xyz坐标和irc坐标的原点偏移量
CT.vzSize_xyz:体素在xyz坐标轴的尺度大小
CT.direction_a:体素的空间矩阵
CT.getRawCandidate函数:
ct_chunk, center_irc = getRawCandidate(center_xyz, width_irc)
center_xyz:结节在xyz坐标系的坐标值。
width_irc:结节在irc坐标系的尺寸大小。也是数据集输入到模型的input_size
ct_chunk:结节在irc坐标轴的HU值的三维矩阵。
center_irc:结节中心在irc坐标系的坐标值。
3. LunaDataset类
ds = LunaDataset(val_stride=0, isValSet_bool=False, series_uid=None)
val_stride:作为验证集时,从数据集中抽取样本作为验证集样本的步长。即每隔val_stride抽取一个样本作为验证集样本。
isValSet_bool:是否作为验证集。
series_uid:获取某个uid对应的所有样本。
四、代码
1. 原书代码
书中代码【dsets.py】如下:
import copy
import csv
import functools
import glob
import osfrom collections import namedtupleimport SimpleITK as sitk
import numpy as npimport torch
import torch.cuda
from torch.utils.data import Datasetfrom util.disk import getCache
from util.util import XyzTuple, xyz2irc
from util.logconf import logginglog = logging.getLogger(__name__)
# log.setLevel(logging.WARN)
# log.setLevel(logging.INFO)
log.setLevel(logging.DEBUG)raw_cache = getCache('part2ch10_raw')CandidateInfoTuple = namedtuple('CandidateInfoTuple','isNodule_bool, diameter_mm, series_uid, center_xyz',
)@functools.lru_cache(1)
def getCandidateInfoList(requireOnDisk_bool=True):# We construct a set with all series_uids that are present on disk.# This will let us use the data, even if we haven't downloaded all of# the subsets yet.mhd_list = glob.glob('data-unversioned/part2/luna/subset*/*.mhd')presentOnDisk_set = {os.path.split(p)[-1][:-4] for p in mhd_list}diameter_dict = {}with open('data/part2/luna/annotations.csv', "r") as f:for row in list(csv.reader(f))[1:]:series_uid = row[0]annotationCenter_xyz = tuple([float(x) for x in row[1:4]])annotationDiameter_mm = float(row[4])diameter_dict.setdefault(series_uid, []).append((annotationCenter_xyz, annotationDiameter_mm))candidateInfo_list = []with open('data/part2/luna/candidates.csv', "r") as f:for row in list(csv.reader(f))[1:]:series_uid = row[0]if series_uid not in presentOnDisk_set and requireOnDisk_bool:continueisNodule_bool = bool(int(row[4]))candidateCenter_xyz = tuple([float(x) for x in row[1:4]])candidateDiameter_mm = 0.0for annotation_tup in diameter_dict.get(series_uid, []):annotationCenter_xyz, annotationDiameter_mm = annotation_tupfor i in range(3):delta_mm = abs(candidateCenter_xyz[i] - annotationCenter_xyz[i])if delta_mm > annotationDiameter_mm / 4:breakelse:candidateDiameter_mm = annotationDiameter_mmbreakcandidateInfo_list.append(CandidateInfoTuple(isNodule_bool,candidateDiameter_mm,series_uid,candidateCenter_xyz,))candidateInfo_list.sort(reverse=True)return candidateInfo_listclass Ct:def __init__(self, series_uid):mhd_path = glob.glob('data-unversioned/part2/luna/subset*/{}.mhd'.format(series_uid))[0]ct_mhd = sitk.ReadImage(mhd_path)ct_a = np.array(sitk.GetArrayFromImage(ct_mhd), dtype=np.float32)# CTs are natively expressed in https://en.wikipedia.org/wiki/Hounsfield_scale# HU are scaled oddly, with 0 g/cc (air, approximately) being -1000 and 1 g/cc (water) being 0.# The lower bound gets rid of negative density stuff used to indicate out-of-FOV# The upper bound nukes any weird hotspots and clamps bone downct_a.clip(-1000, 1000, ct_a)self.series_uid = series_uidself.hu_a = ct_aself.origin_xyz = XyzTuple(*ct_mhd.GetOrigin())self.vxSize_xyz = XyzTuple(*ct_mhd.GetSpacing())self.direction_a = np.array(ct_mhd.GetDirection()).reshape(3, 3)def getRawCandidate(self, center_xyz, width_irc):center_irc = xyz2irc(center_xyz,self.origin_xyz,self.vxSize_xyz,self.direction_a,)slice_list = []for axis, center_val in enumerate(center_irc):start_ndx = int(round(center_val - width_irc[axis]/2))end_ndx = int(start_ndx + width_irc[axis])assert center_val >= 0 and center_val < self.hu_a.shape[axis], repr([self.series_uid, center_xyz, self.origin_xyz, self.vxSize_xyz, center_irc, axis])if start_ndx < 0:# log.warning("Crop outside of CT array: {} {}, center:{} shape:{} width:{}".format(# self.series_uid, center_xyz, center_irc, self.hu_a.shape, width_irc))start_ndx = 0end_ndx = int(width_irc[axis])if end_ndx > self.hu_a.shape[axis]:# log.warning("Crop outside of CT array: {} {}, center:{} shape:{} width:{}".format(# self.series_uid, center_xyz, center_irc, self.hu_a.shape, width_irc))end_ndx = self.hu_a.shape[axis]start_ndx = int(self.hu_a.shape[axis] - width_irc[axis])slice_list.append(slice(start_ndx, end_ndx))ct_chunk = self.hu_a[tuple(slice_list)]return ct_chunk, center_irc@functools.lru_cache(1, typed=True)
def getCt(series_uid):return Ct(series_uid)@raw_cache.memoize(typed=True)
def getCtRawCandidate(series_uid, center_xyz, width_irc):ct = getCt(series_uid)ct_chunk, center_irc = ct.getRawCandidate(center_xyz, width_irc)return ct_chunk, center_ircclass LunaDataset(Dataset):def __init__(self,val_stride=0,isValSet_bool=None,series_uid=None,):self.candidateInfo_list = copy.copy(getCandidateInfoList())if series_uid:self.candidateInfo_list = [x for x in self.candidateInfo_list if x.series_uid == series_uid]if isValSet_bool:assert val_stride > 0, val_strideself.candidateInfo_list = self.candidateInfo_list[::val_stride]assert self.candidateInfo_listelif val_stride > 0:del self.candidateInfo_list[::val_stride]assert self.candidateInfo_listlog.info("{!r}: {} {} samples".format(self,len(self.candidateInfo_list),"validation" if isValSet_bool else "training",))def __len__(self):return len(self.candidateInfo_list)def __getitem__(self, ndx):candidateInfo_tup = self.candidateInfo_list[ndx]width_irc = (32, 48, 48)candidate_a, center_irc = getCtRawCandidate(candidateInfo_tup.series_uid,candidateInfo_tup.center_xyz,width_irc,)candidate_t = torch.from_numpy(candidate_a)candidate_t = candidate_t.to(torch.float32)candidate_t = candidate_t.unsqueeze(0)pos_t = torch.tensor([not candidateInfo_tup.isNodule_bool,candidateInfo_tup.isNodule_bool],dtype=torch.long,)return (candidate_t,pos_t,candidateInfo_tup.series_uid,torch.tensor(center_irc),)
2. 我注释的代码
import functools
import glob
import os.path
import csv
import SimpleITK as sitk
import numpy as np
import copyimport torch
import torch.cuda
from torch.utils.data import Datasetfrom collections import namedtuplefrom util.disk import getCache
from util.util import XyzTuple, xyz2irc
from util.logconf import logginglog = logging.getLogger(__name__)
log.setLevel(logging.DEBUG)# annotations.csv: 记录实际结节。文件结构: uid, x, y, z, diameter
# candidates.csv: 记录候选结节。文件结构: uid, x, y, z, classraw_cache = getCache('part2ch10_raw')# 构建用于存储候选结节的元组, 结构: class, diameter, id, xyz
candidateInfoTuple = namedtuple('candidateInfoTuple','isNodule_bool, diameter_mm, series_uid, center_xyz')@functools.lru_cache(1) # 缓存一次调用结果
def getCandidateInfoList(requireOnDisk_bool=True):"""加载annotations.csv和candidates.csv,分别存到diameter_list和candidateInfo_list:param requireOnDisk_bool. 如果文件不存在,是否跳过:return candidateInfo_list. 由candidateInfoTuple构成的list"""mhd_list = glob.glob('data-unversioned/part2/luna/subset*/*.mhd')presentOnDisk_set = {os.path.split(p)[-1][:-4] for p in mhd_list} # 提取所有文件名,即uiddiameter_dict= {}with open('data/part2/luna/annotations.csv', 'r') as f:for row in list(csv.reader(f))[1:]:series_uid = row[0]annotationCenter_xyz = tuple([float(x) for x in row[1:4]])annotationDiameter_mm = float(row[4])diameter_dict.setdefault(series_uid, []).append((annotationCenter_xyz, annotationDiameter_mm))candidateInfo_list = []with open('data/part2/luna/candidates.csv', 'r') as f:for row in list(csv.reader(f))[1:]:series_uid = row[0]# 如果annotations.csv中找不到这个id,则跳过if series_uid not in presentOnDisk_set and requireOnDisk_bool:continuecandidateDiameter_xyz = tuple([float(x) for x in row[1:4]])isNodule_bool = bool(int(row[4]))# 如果candidate中的xyz坐标和annotation中的xyz坐标偏差大于半径的一半,# 则认为它们不是同一个节点,将直接用零代替,即认为这不是结节candidateDiameter_mm = 0.0for annotation_tup in diameter_dict.get(series_uid, []):annotation_xyz, annotationDiameter_mm = annotation_tupfor i in range(3):delta_mm = abs(candidateDiameter_xyz[i] - annotation_xyz[i])if delta_mm > annotationDiameter_mm/4:breakelse:candidateDiameter_mm = annotationDiameter_mmbreakcandidateInfo_list.append(candidateInfoTuple(isNodule_bool,candidateDiameter_mm,series_uid,candidateDiameter_xyz,))candidateInfo_list.sort(reverse=True)return candidateInfo_listclass Ct:def __init__(self, series_uid):mhd_path = glob.glob(r'data-unversioned/part2/luna/subset*/{}.mhd'.format(series_uid))[0]# 用SampleSTK包可直接读取CT扫描数据ct_mhd = sitk.ReadImage(mhd_path)# HU: 亨氏单位,Hounsfield Unit.# 空气为-1000 HU,约等于0 g/cm3. 水为0 HU,约等于1 g/cm3, 骨骼至少时1000HU,约等于2~3g/cm3ct_a = np.array(sitk.GetArrayFromImage(ct_mhd), dtype=np.float32) # 读取到的数据单位为HU# 将数据限定再-1000~1000 HUct_a.clip(-1000, 1000, ct_a)self.series_uid = series_uidself.hu_a = ct_aself.origin_xyz = XyzTuple(*ct_mhd.GetOrigin()) # xyz坐标和irc坐标的原点偏移量self.vxSize_xyz = XyzTuple(*ct_mhd.GetSpacing()) # 体素在xyz坐标轴的大小self.direction_a = np.array(ct_mhd.GetDirection()).reshape(3, 3) # 体素方向矩阵,等于eye(3)def getRawCandidate(self, center_xyz, width_irc):"""根据xyz坐标算出病人坐标irc。然后根据每个结节的irc和体素宽度,算出结节包含的体素块数据:param center_xyz: 结节的xyz坐标:param width_irc: 体素宽度,也是数据集输入到模型的输入尺寸:return ct_chunk: 结节包含的体素块的HU值,array:return center_irc: 结节的病人坐标信息"""center_irc = xyz2irc(center_xyz,self.origin_xyz,self.vxSize_xyz,self.direction_a)slice_list = []for axis, center_val in enumerate(center_irc):start_ndx = int(round(center_val - width_irc[axis]/2))end_ndx = int(start_ndx + width_irc[axis])assert center_val >= 0 and center_val < self.hu_a.shape[axis], repr([self.series_uid, center_xyz, self.origin_xyz, self.vxSize_xyz, center_irc, axis])if start_ndx < 0:# log.warning("Crop outside of CT array: {} {}, center:{} shape:{} width:{}".format(# self.series_uid, center_xyz, center_irc, self.hu_a.shape, width_irc))start_ndx = 0end_ndx = int(width_irc[axis])if end_ndx > self.hu_a.shape[axis]:# log.warning("Crop outside of CT array: {} {}, center:{} shape:{} width:{}".format(# self.series_uid, center_xyz, center_irc, self.hu_a.shape, width_irc))end_ndx = self.hu_a.shape[axis]start_ndx = int(self.hu_a.shape[axis] - width_irc[axis])slice_list.append(slice(start_ndx, end_ndx))ct_chunk = self.hu_a[tuple(slice_list)]return ct_chunk, center_irc@functools.lru_cache(1, typed=True) # 保留一次缓存结果
def getCt(series_uid):return Ct(series_uid)@raw_cache.memoize(typed=True) # 数据缓存到同路径的cache文件夹下
def getCtRawCandidate(series_uid, center_xyz, width_irc):ct = getCt(series_uid)ct_chunk, center_irc = ct.getRawCandidate(center_xyz, width_irc)return ct_chunk, center_ircclass LunaDataset(Dataset):def __init__(self, val_stride=0, isValSet_bool=False, series_uid=None):"""val_stride:作为验证集时,从数据集中抽取样本作为验证集样本的步长。即每隔val_stride抽取一个样本作为验证集样本。isValSet_bool:是否作为验证集。series_uid:获取某个uid对应的所有样本。 """self.candidateInfo_list = copy.copy(getCandidateInfoList())if series_uid:self.candidateInfo_list = [x for x in self.candidateInfo_list if x.series_uid==series_uid]if isValSet_bool:assert val_stride > 0, val_strideself.candidateInfo_list = self.candidateInfo_list[::val_stride]assert self.candidateInfo_listelif val_stride > 0:del self.candidateInfo_list[::val_stride]assert self.candidateInfo_listlog.info("(!r): {} {} samples".format(self,len(self.candidateInfo_list),"validation" if isValSet_bool else "training",))def __len__(self):return len(self.candidateInfo_list)def __getitem__(self, ndx):"""返回指定索引对应的结节信息:param ndx: 某个ct数据中的第ndx个结节索引:return: candidate_t. 结节所包含的所有体素的三位数组。t代表数组时个tensor:return: post_t. 结节是否为肿瘤。0代表不是,1代表肿瘤。:return: series_uid. ndx所对应的结节uid:return: center_irc. 结节的重心坐标。类型为tensor"""candidateInfo_tup = self.candidateInfo_list[ndx]width_irc = (32, 48, 48)candidate_a, center_irc = getCtRawCandidate(candidateInfo_tup.series_uid,candidateInfo_tup.center_xyz,width_irc,)candidate_t = torch.from_numpy(candidate_a)candidate_t = candidate_t.to(torch.float32)candidate_t = candidate_t.unsqueeze(0)post_t = torch.tensor([not candidateInfo_tup.isNodule_bool,candidateInfo_tup.isNodule_bool],dtype=torch.long,)return (candidate_t,post_t,candidateInfo_tup.series_uid,torch.tensor(center_irc))
这篇关于四、肺癌检测-数据集准备 dsets.py文件的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!





