本文主要是介绍CHAPERONg:基于GROMACS的分子动力学模拟和轨迹分析的自动化工具,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!

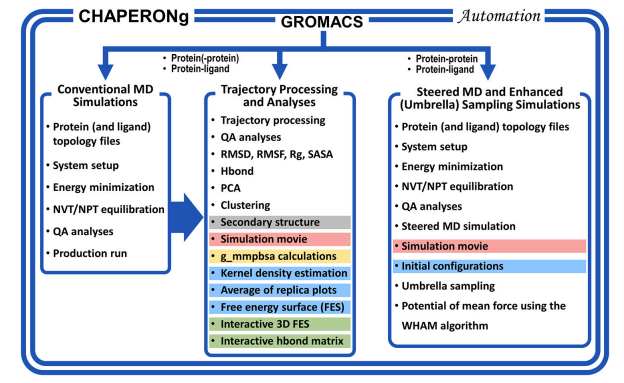
此工作介绍了一个名为CHAPERONg的工具,它是一个自动化的GROMACS(一种广泛使用的分子动力学模拟软件)模拟和轨迹分析工具。CHAPERONg旨在简化和自动化蛋白质和蛋白质-配体系统的GROMACS分子动力学(MD)模拟流程,包括常规MD模拟、拉伸MD(steered MD)和增强型伞采样(umbrella sampling)模拟。这个工具通过集成GROMACS模块和第三方工具,提供了多达20种后模拟处理和轨迹分析功能,使得MD模拟对初学者更加友好,同时为研究人员提供了专注于数据解释和其他非编程方面的平台。
项目地址:https://github.com/abeebyekeen/CHAPERONg
CHAPERONg可以处理含有配体分子的蛋白-配体复合物的自动建模。根据提供的文件内容,CHAPERONg支持蛋白质-配体系统的GROMACS分子动力学(MD)模拟。它能够识别通过流行的配体参数化程序和网络服务器生成的小分子配体拓扑结构,这些程序包括:
- CGenFF(用于CHARMM力场)
- ACPYPE(用于AMBER力场)
- PRODRG2(用于GROMOS力场)
- LigParGen(用于OPLS-AA力场)
在进行模拟之前,CHAPERONg会自动执行以下步骤:
系统准备:生成蛋白质和配体的拓扑结构。
模拟盒定义:创建一个占位的立方单元格,并引导用户交互式地调整盒子和质心尺寸。
溶剂化和离子添加:在系统中添加溶剂和离子,以模拟生物环境。
能量最小化和平衡:进行能量最小化和NVT/NPT平衡步骤,以确保系统的稳定性。
这些步骤为蛋白质-配体复合物的MD模拟提供了必要的前期准备,使得用户可以专注于模拟的执行和后续分析。通过这种方式,CHAPERONg简化了蛋白质-配体复合物的建模过程,使得即使是没有丰富经验的用户也能够进行有效的模拟研究。
安装流程
同名公众号后台回复 “240213” 自取安装包
-
- 配置conda环境
unzip CHAPERONg-main.zip
cd CHAPERONg-main
chmod +x conda_env_setup.sh && bash ./conda_env_setup.sh
-
- 安装CHAPERONg
chmod +x install_CHAPERONg.sh && ./install_CHAPERONg.sh
安装完成的界面:
-
- 使用
source ~/.bashrc
conda activate chaperong
./run_CHAPERONg.sh -i inputStructure_filename [more options]
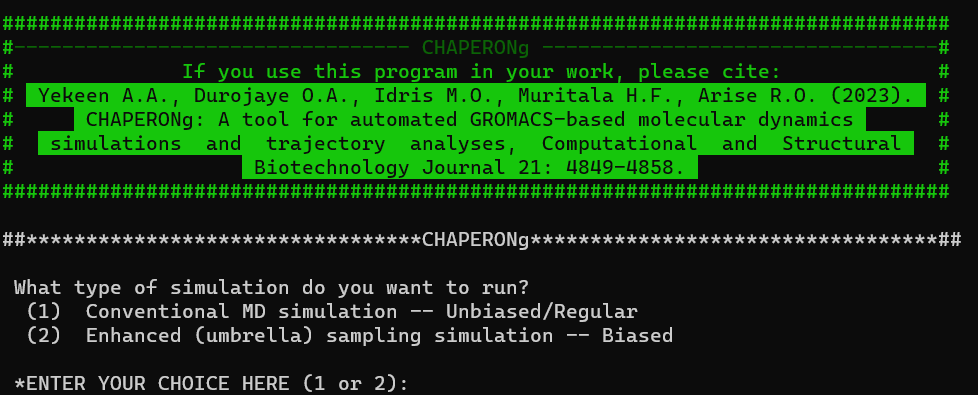
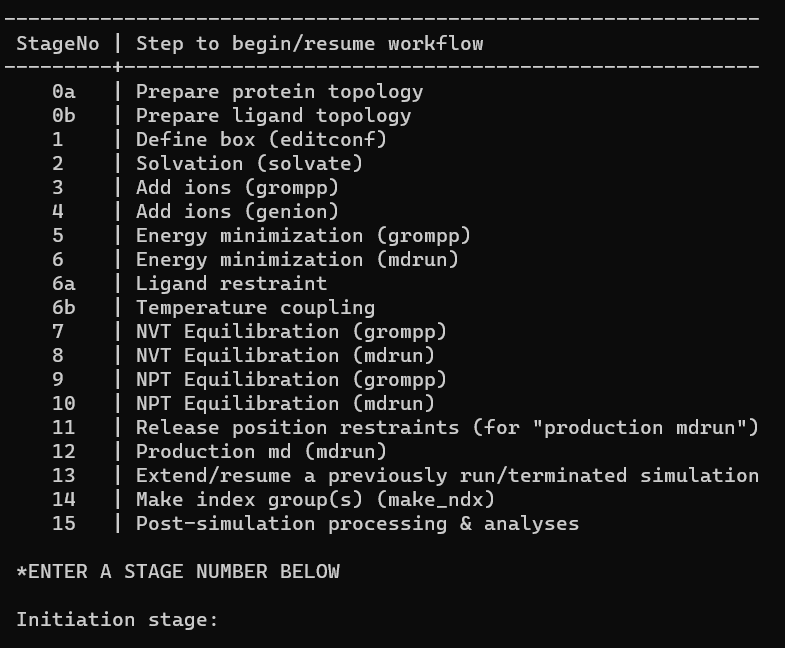
交互界面如下:
这篇关于CHAPERONg:基于GROMACS的分子动力学模拟和轨迹分析的自动化工具的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!








