本文主要是介绍【plink】如何把基因型数据ATCG格式转换为012之一 --recodeA,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
plink官网:Data management - PLINK 1.9
命令:将基因型数据转化为012的raw格式
plink --file DD --recodeA --out DD_test_A#结果文件 DD_test_A.raw查看具体说明:
--recodeA : snp的major变为了0, snp的minor变为了2, 杂合变为了1.

具体查看:
plink --bfile DD --recodeA --out DD_test
plink --bfile DD --recodeAD --out DD_test_AD
原始ped
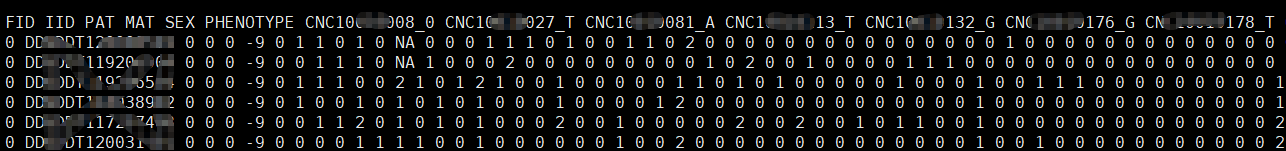
--recodeA : 一个SNP标记只有1列,纯合转换为02,杂合为1(如TT 替换为0, 0是major ; AA 替换为2 ; AT 替换为1)
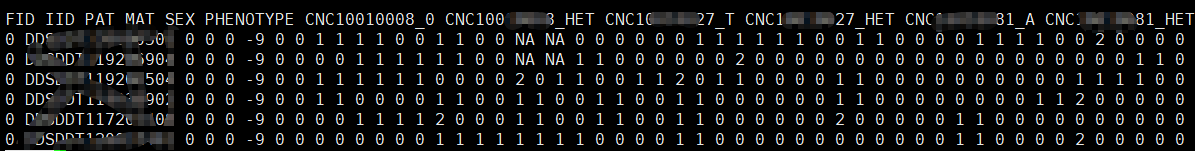
--recodeAD:一个标记有2列 (TT 替换为00,AA替换为20 ;TC 替换为11)
本文章仅作为个人笔记与大家分享,希望大家少走弯路,过程中参考了一系列大神的文章,如有侵犯劳烦联系删除。
这篇关于【plink】如何把基因型数据ATCG格式转换为012之一 --recodeA的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







