本文主要是介绍综述 2019-Genome Biology:非比对方法的benchmark,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
Zielezinski, Andrzej, et al. "Benchmarking of alignment-free sequence comparison methods." Genome biology 20.1 (2019): 1-18. Benchmarking of alignment-free sequence comparison methods | Genome Biology | Full Text
- 被引次数:170;
- 研究背景:DNA 和氨基酸序列的比较分析在生物学研究中至关重要,特别是在分子生物学和基因组学中。它是分子进化分析、基因功能和调控区预测、序列组装、同源性搜索、分子结构预测、基因发现、蛋白质结构与功能关系分析的第一步和关键步骤。
一、项目地址
AF | HomeAFproject is a free service for objective performance benchmark of alignment-free sequence comparison tools.
http://afproject.org/
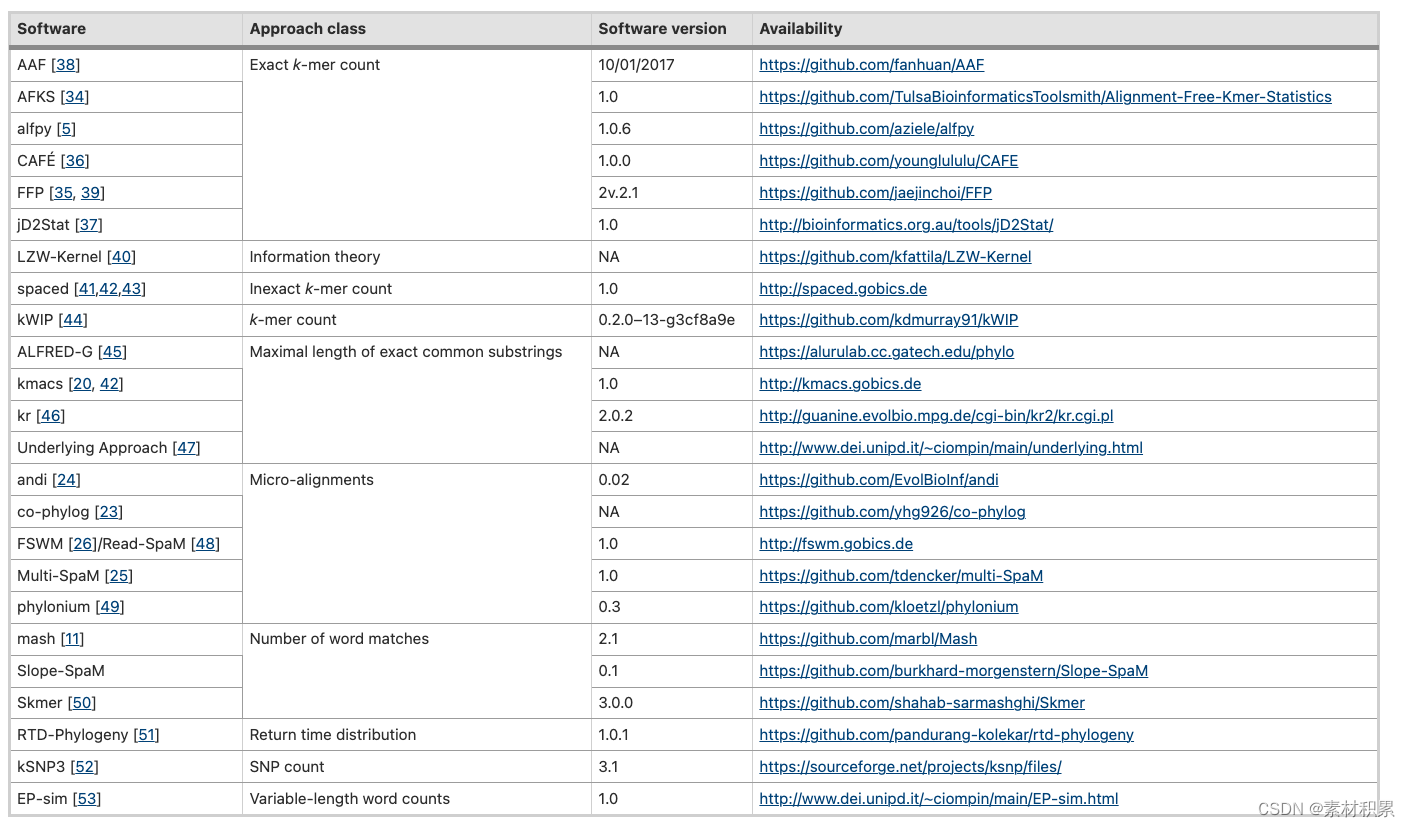
项目服务流程:
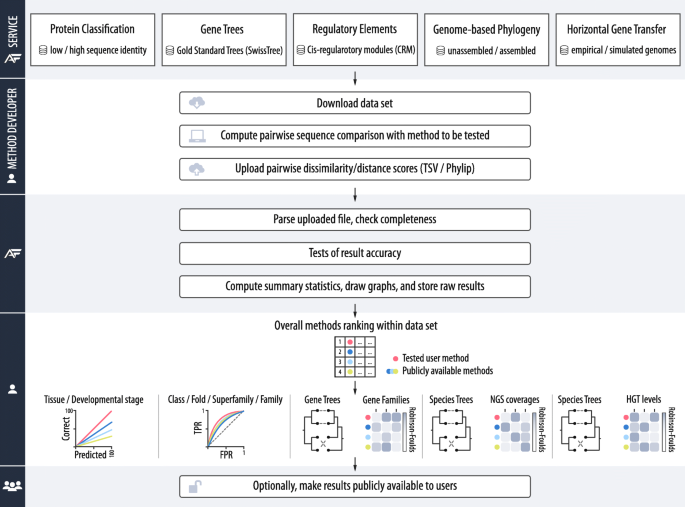
二、24 个软件
包含了 74 种 AF 方法
三、5 种应用场景
- 蛋白质序列分类 protein sequence classification
- 基因树推断 gene tree inference
- 调控元件检测 regulatory element detection
- 基因组的系统发育推断 genome-based phylogenetic inference
- 水平基因转移和重组事件下物种树的重建 reconstruction of species trees under horizontal gene transfer and recombination events
四、数据集
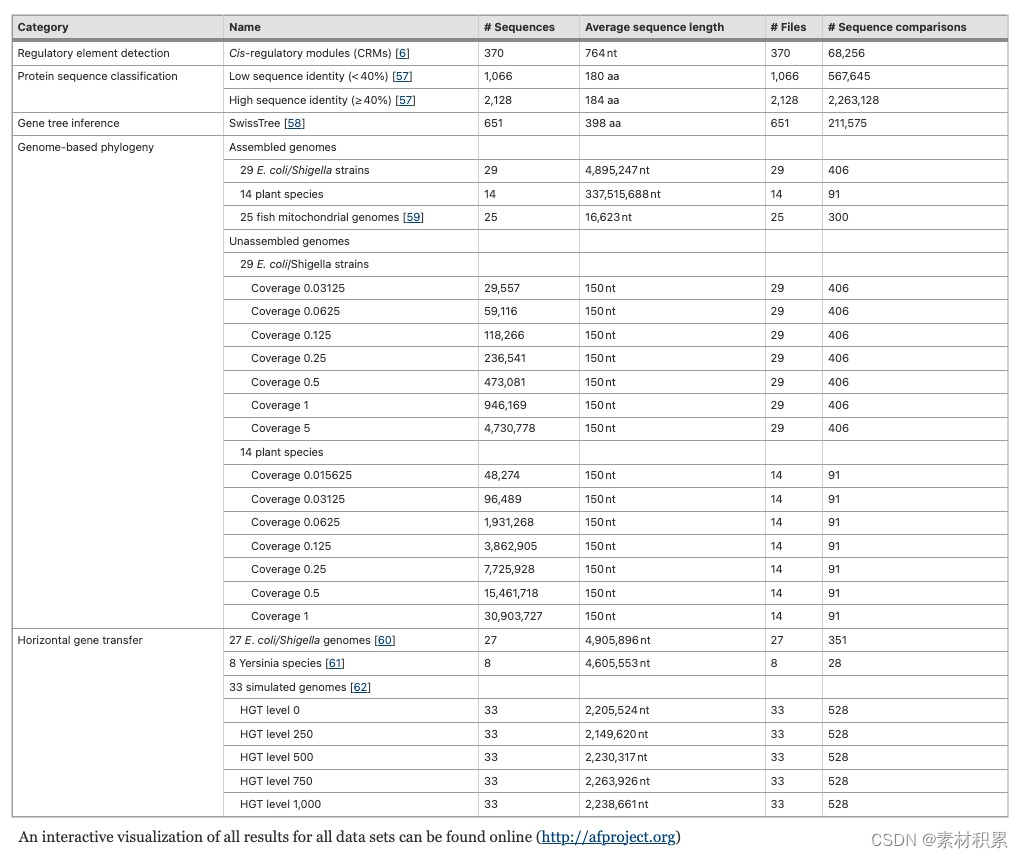
这篇关于综述 2019-Genome Biology:非比对方法的benchmark的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!