本文主要是介绍从NBCI上下载大量细菌的SRA测序文件及其对应的fasta文件,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
第一步 从genome数据库中下载对应信息
- 打开NCBI,进入genome数据库
- 从Custom resources下进入Microbes,再进入Browse microbial genomes
- 输入需要的物种,可以进一步筛选(filter),然后download
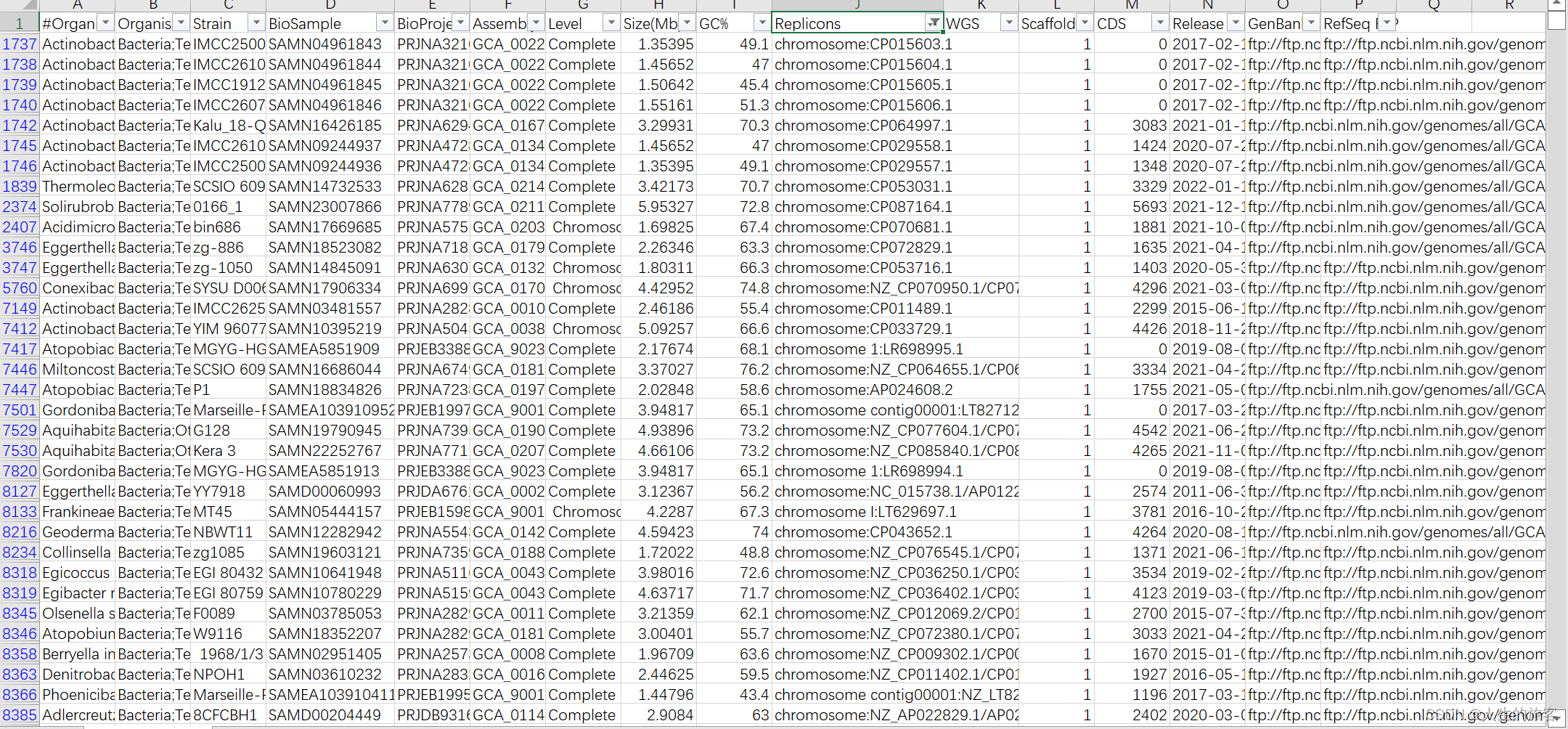
第二步 提取csv结果文件中的biosmple列的biosample号和replicon列的chromesome号
- replicon,有一些是空的,需要筛选;chromesome号有CP、NZ、NC三种,按需提取
- 分别将biosample号、chromesome号存为文件
第三步 batch entrez下载数据
- nucleotide数据库下载chromesome号的文件,已经就是fasta文件了
- biosample数据库下载biosample的summary文件;如需更多信息,可以选择full下载样本的所有信息,包括environment等;下图为选择full之后的下载结果,可能存在部分样本不含SRA文件的情况,可以进一步筛选(也可以直接在搜索页面进行筛选)

- 提取上一步下载的文件的sra号,再去选择sra数据库下载SRA文件
这篇关于从NBCI上下载大量细菌的SRA测序文件及其对应的fasta文件的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!





