本文主要是介绍BWA-MEM算法结构分析,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
一、BWA-MEM函数框架
软件位置:https://sourceforge.net/projects/bio-bwa/
1 读入 bwt、options、reads;
2 利用mem_chain生成chain;
3 利用mem_chain_flt过滤掉部分chain;
4 利用mem_chain2aln生成比对结果数据。
1.第一步:数据输入
加载已经生成的bwt表。接口的参数文件名为:xx.fasta;实际中包含具有以下几个后缀名的文件.amb,.ann,.bwt,.fai,.pac,.sa。注意后面几个文件的名字要和接口参数的文件名一致。
其中这几个文件分别为:
.bwt文件:重新排列的bwt表,大小与原reference相同。存储的是o表。
.sa文件:sa_intv
.pac文件:原始reference的压缩文件,原来ACTG用ASCIIS码表示,一个字符占八位,在.pac文件中,用00~11的2bit重新编码bwt表,使得.pac文件成为原来.fasta文件大小的1/4.
.ann文件:reference分组的划分信息
.amb文件:
2.第二步:
mem_align1()函数功能:给一个read匹配一块reference区域
输入:const mem_opt_t *opt~运算中的匹配数据。alignment parameters
const bwt_t *bwt~ bwt表
const bntseq_t *bns ~reference的信息。Information of the reference
const uint8_t *pac~压缩后的reference表
int l_seq ~read 的长度
char *seq~read(待匹配片段)返回值:mem_alnreg_v regs~描述确定reads下匹配到的在reference上的位置信息
3. 第三步:
mem_reg2aln():功能:根据reads和reference的匹配结果,生成CIGAR(一种描述匹配情况的信息)
输入:
const mem_opt_t * opt~alignment parameters
int64_t l_pac~length of concatenated reference sequence
int n~number of query sequences;must be an even number
const mem_alnreg_v *regs ~region array of size $n; 2i-th and (2i+1)-th elements constitute a pair
mem_pestat_t pes[4])~inferred insert size distribution (output)
返回:
mem_aln_t a ~CIGAR, strand, mapping quality and forward-strand position
1.bwa_idx_load()//加载bwt表for(every reads){ 2.ar = mem_align1();//找到每个reads 的匹配区域for (i = 0; i < ar.n; ++i){ // traverse each hit3.a = mem_reg2aln()// get forward-strand position and CIGARfor (k = 0; k < a.n_cigar; ++k) // print CIGARprintf("%d%c", a.cigar[k]>>4, "MIDSH"[a.cigar[k]&0xf]);printf("\t%d\n", a.NM); // print edit distance}
}
二、各函数概括
2.1bwa_idx_load()//加载bwt表
- bwt表是如何由reference重构组成的?
长为n的reference右移->在n×n的空间里从左向右排序->保留最后一列的数据作为bwt
typedef struct {
bwtint_t primary; // S^{-1}(0), or the primary index of BWT
bwtint_t L2[5]; // C(), cumulative coun~L2[0]]--L2[4]分别对应初始bwt表中,以ACGT为首的interval的索引
bwtint_t seq_len; // sequence length~bwt表总长度
bwtint_t bwt_size; // size of bwt, about seq_len/4~该表的总比特数
uint32_t *bwt; // BWT表
// occurance array, separated to two parts
uint32_t cnt_table[256];
// suffix array
int sa_intv;
bwtint_t n_sa;
bwtint_t *sa;
} bwt_t;
2.2mem_align1():找匹配区域函数
2.2.1mem_align1():
- 功能:给一个read匹配一块reference区域
- 结构:mem_align1()=mem_align1_core()+mem_mark_primary_se()
- 代码:
mem_alnreg_v mem_align1(const mem_opt_t *opt, const bwt_t *bwt, const bntseq_t *bns, const uint8_t *pac, int l_seq, const char *seq_)
{ mem_alnreg_v ar;char *seq;seq = malloc(l_seq);memcpy(seq, seq_, l_seq); // makes a copy of seq_ar = mem_align1_core(opt, bwt, bns, pac, l_seq, seq, 0);mem_mark_primary_se(opt, ar.n, ar.a, lrand48());free(seq);return ar;
}2.2.2 mem_align1_core()
- 结构:mem_align1_core=mem_chain+mem_chain_flt+mem_flt_chained_seeds+mem_chain2aln+mem_sort_dedup_patch
- 代码:
mem_alnreg_v mem_align1_core(const mem_opt_t *opt, const bwt_t *bwt, const bntseq_t *bns, const uint8_t *pac, int l_seq, char *seq, void *buf)
{int i;mem_chain_v chn;mem_alnreg_v regs;for (i = 0; i < l_seq; ++i) // convert to 2-bit encoding if we have not done soseq[i] = seq[i] < 4? seq[i] : nst_nt4_table[(int)seq[i]];chn = mem_chain(opt, bwt, bns, l_seq, (uint8_t*)seq, buf);chn.n = mem_chain_flt(opt, chn.n, chn.a);mem_flt_chained_seeds(opt, bns, pac, l_seq, (uint8_t*)seq, chn.n, chn.a);if (bwa_verbose >= 4) mem_print_chain(bns, &chn);kv_init(regs);for (i = 0; i < chn.n; ++i) {mem_chain_t *p = &chn.a[i];if (bwa_verbose >= 4) err_printf("* ---> Processing chain(%d) <---\n", i);mem_chain2aln(opt, bns, pac, l_seq, (uint8_t*)seq, p, ®s);free(chn.a[i].seeds);}free(chn.a);regs.n = mem_sort_dedup_patch(opt, bns, pac, (uint8_t*)seq, regs.n, regs.a);if (bwa_verbose >= 4) {err_printf("* %ld chains remain after removing duplicated chains\n", regs.n);for (i = 0; i < regs.n; ++i) {mem_alnreg_t *p = ®s.a[i];printf("** %d, [%d,%d) <=> [%ld,%ld)\n", p->score, p->qb, p->qe, (long)p->rb, (long)p->re);}}for (i = 0; i < regs.n; ++i) {mem_alnreg_t *p = ®s.a[i];if (p->rid >= 0 && bns->anns[p->rid].is_alt)p->is_alt = 1;}return regs;
}三.核心逻辑:
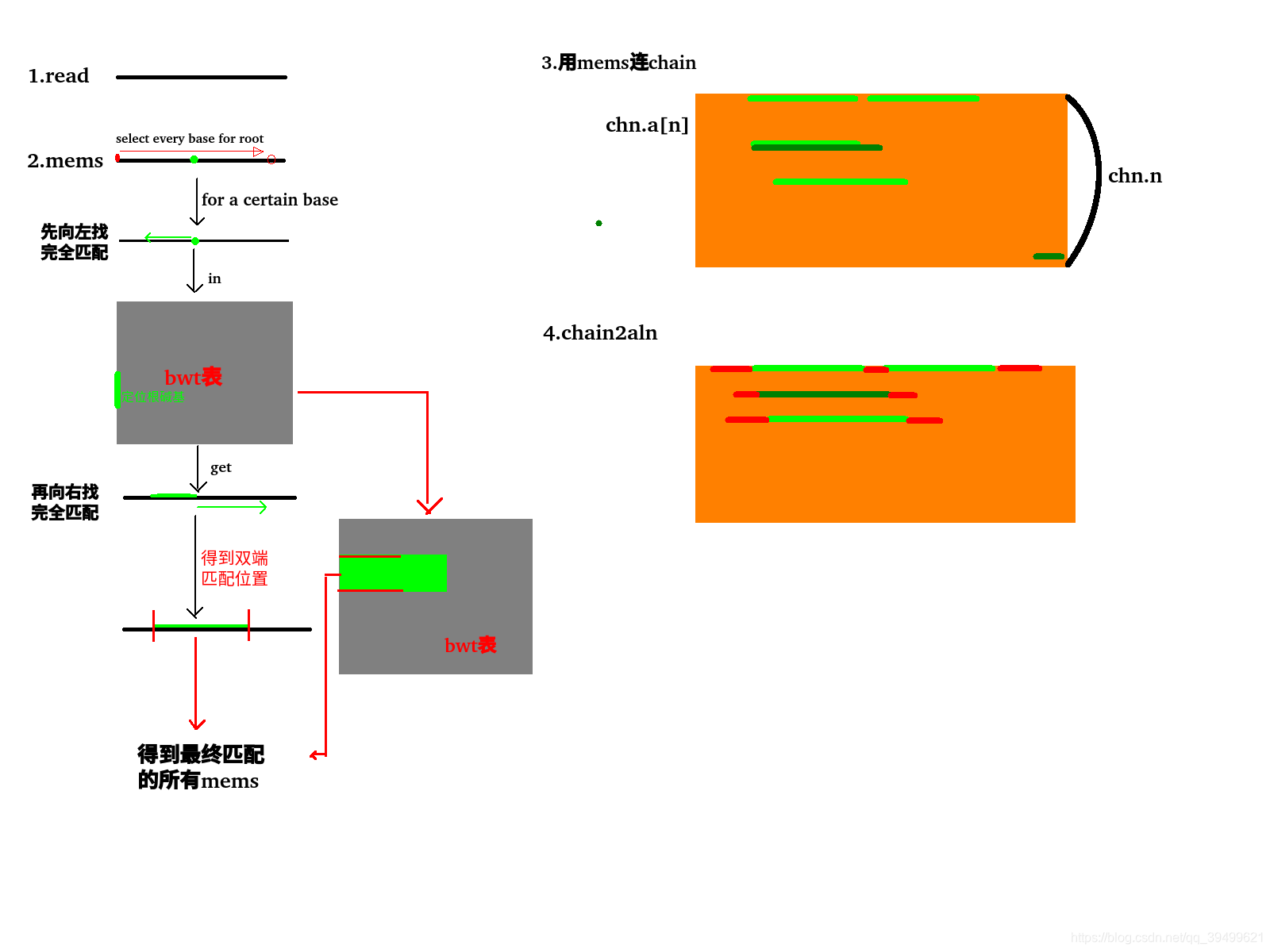
3.1mem_chain()
1.寻找SMEMS:mem_collect_intv()
a. find all SMEMS——bwt_smem1()。
b.re-seeding
对于reads上的每个碱基,我们做bwt_smem1(),先向左找完全匹配,再向右找完全匹配。找到所有的SMEMS。为防止遗漏个别最长敬佩匹配,采用re-seeding进行后期纠错。
a.bwt_smem1()分析:
bwtintv_t ik, ok[4];
ik:输入的bi-interval。x[0]:正向interval,x[1]反向interval,x[2]interval的个数。
ok[4]:ik前面是ACGT的bi-interval。
重点分析一下反向匹配的过程:
1.反相向左匹配遵循以下几点原则:
a.在向左匹配后x[2]<1,则考虑是否存为最终结果
b.左端相同的mem,仅存最长的一个(排序后的第一个)
for (i = x - 1; i >= -1; --i) { // backward search for MEMs
c = i < 0? -1 : q[i] < 4? q[i] : -1; // c==-1 if i<0 or q[i] is an ambiguous base
for (j = 0, curr->n = 0; j < prev->n; ++j) {
bwtintv_t *p = &prev->a[j];
if (c >= 0 && ik.x[2] >= max_intv)
bwt_extend(bwt, p, ok, 1);
if (c < 0 || ik.x[2] < max_intv || ok[c].x[2] < min_intv) { //如果向左匹配ok->x[2]没有数据了,
if (curr->n == 0) { //
if (mem->n == 0 || i + 1 < mem->a[mem->n-1].info>>32) { //
ik = *p; ik.info |= (uint64_t)(i + 1)<<32;
kv_push(bwtintv_t, *mem, ik);
}
} // otherwise the match is contained in another longer match
} else if (curr->n == 0 || ok[c].x[2] != curr->a[curr->n-1].x[2]) {//向左有戏还得看:1.curr是否为空;2.ok->x[2]是否更新ok[c].info = p->info;
kv_push(bwtintv_t, *curr, ok[c]);
}
}
if (curr->n == 0) break;
swap = curr; curr = prev; prev = swap;
}
b.re-seeding。未防止选中含错配的最长匹配SMEM,通过re-seeding进行处理。即认定在某些SMEM中,为追求某个最长匹配,在众多准确匹配关系中,选择了其中某几条。是一种以SMEM长度换个数的算法实现。其目的是防止遗漏种子。
3.2mem_chain():将read连成chain
输入:
const mem_opt_t *opt
const bwt_t *bwt
const bntseq_t *bns
int len
const uint8_t *seq
void *buf
输出:
mem_chain_v chain ~链接过后的chain链
- 函数流程描述:
先将读取得到的read在bwt表上进行完全匹配,即mem_collect_intv,其返回一个aux类型的指针,指针里面存储着该read在bwt表中的全部smems,然后
- 1.mem_chain()子函数:
- mem_collect_intv()
功能:在bwt上找到seq对应的所有的MEMS
输入:const mem_opt_t *opt
const bwt_t *bwt
int len~sequence的长度
const uint8_t *seq
输出:
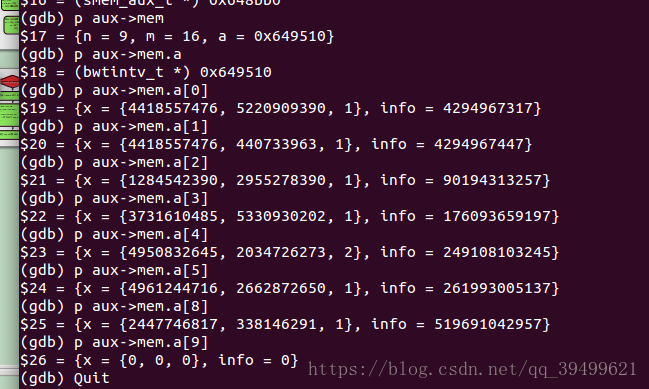
smem_aux_t *a ~存储smems的指针,描述的是该sequence的所有mems信息,具体描述如下。a->mem显示处n=9,表示该sequence在reference上有9个Mems,m=16未知,a=0x629510是指针地址。
a->mem.a[]只有9个内容,x的第一个值是interval的forward索引起始位置,第二个是backward索引位置,第三个值是interval的个数。info是40bit的数据,例如info=519691042957,化成16进制数是:790000008d(000000790000008d),指的是从sequence的左端第79个碱基开始,到第8×16+14=142个碱基具有这个Smem。即高32位存在sequence的左端索引,低32位是在sequence的右端索引。
第一步:对读进来的sequence,从左边第一个碱基开始,作为起点,做bwt_smem1a(),bwtsmem1a是以单个碱基为输入参数在bwt表对interval进行匹配的函数,以bwt_smem1a()的返回值作为下一个开始位置的碱基
第二步:在较长的SMEM里面继续找MEMS(Re-seeding)
第三步:在read里面从左到右遍历一次,逐个找到不重叠Mems
将以上三步找到的MEMs作为最终的结果,存给smem_aux_t类型的a~aux
aux类型是:smem_aux_t ,smem_aux_t是bwtintv_v类型的结构体。bwtintv_v这个结构体是64位的无符号立即数。
smem_aux_t: { bwtintv_v mem(入栈参数), mem1(参与运算的中间参数), *tmpv[2];} smem_aux_t;
bwtintv_v: { size_t n(bwtintv_t的个数), m; bwtintv_t *a; } bwtintv_v;
bwtintv_t: {
bwtint_t x[3](其中x[0]:向前匹配的interval的起点~x[1]:向后匹配的interval起点~x[2]:interval的长度(个数)),
info(info的低32位存Mems右端在sequence上的索引位置,高32位存Mems左端的索引位置);}
bwtint_t:uint64_t
1.1.bwt_smem1a():
功能:针对一个固定的sequence,以它的一个碱基位置为起始点,先向前(右)匹配,找到最右端无法匹配的碱基索引,再向左匹配,最终将最右端的索引返回,该函数以返回值作为新的起点,继续匹配。具体匹配规则后续写。
3.2.2mem_chain_flt()
输入:
const mem_opt_t *opt,
int n_chn,~mem_chain()生成的chain
mem_chain_t *a
返回值:
int chn.n chian的个数
-
对于每个read的所有chain进行过滤。
n_chn:总的chain个数
chn.n:该条chain的seeds个数
kvec_t(int) chains = {0,0,0};用来存储非重叠链的int索引
2.1对每个chain,做初步筛选,通过mem_chain_weight函数得到每条seed的weight,若weight小于最小可行seed的weight,则剔除(free seed)。
2.1.1mem_chain_weight()
为什么遍历两次所有seeds?一遍是在query上的weight,另一遍是在reference上的weight。
2.2对每个chain的每个seed判断是否需要剔除:
遍历chain中的所有seeds,让它和kvec_t(int) chains 中的非重叠链比对,判断其是否是重叠链。
若重叠,则对比前面的kvec_t(int) chains,判断是否有overlap,和是否是有意义的overlap。(how?取第i个chain和第j个chain的最大begin位置,和最小end位置)。
a[j]的三个seeds和a[i]的一个seed:

-
由图可得,i chian的seed和j的chain有overlap。
再判断a.若重叠部分>0.5*最短chain的总长 and 最短chain的总长<10000
我们则判断其为significant overlap。对于这类chain,可理解为直接舍弃。
若不重叠,则将i push进chains,并且将a[i].kept置为2or3(2:significant overlap;3:not overlap)
接着对舍弃的chain我们将它的.kept置为1。
a[c->first].kept = 1;
接着将所有.kept<3的chain都置为0,认为其都是与.kept=3的chain有overlap的chain,free掉这些,并将结果认为成最终return输出。
至此,我们认为由seed连成的几条chain之间,不存在overlap。
- 子函数:mem_seed_sw(opt, bns, pac, l_query, query, s)
3.2.3mem_flt_chained_seeds():过滤chained过的seeds
功能:
在过滤完chain的基础上,我们对seeds进行逐个过滤。
进入条件:if (min_l > MEM_SEEDSW_COEF * l_query) return;
按照给定的数据可知:MEM_MINSC_COEF * log(l_query)> MEM_SEEDSW_COEF * l_query
即:5.5*log(l)>0.05f*l; →> l>725
结构:对于每个chain,都有c→n个seeds,对于每个seeds我们做:mem_seed_sw(),计算该seeds的得分。
但实际上,基本进不到mem_seed_sw()中。
在mem_seed_sw()中,做了这些事情:
a.判断seed长度是否已经>MEM_SHORT_LEN,若大于,则直接返回这是一个好的seed,保留。
b.若不是足够好的seed,我们在进行sw算法计算具体的匹配得分。从而在外面断定其是否能够保留。
3.1mem_seed_sw():计算seed得分。
3.1.1bns_fetch_seq(bns, pac, &rb, mid, &re, &rid)在reference的某个位置取出一段碱基序列的头指针,该位置由rb、mid、re、ri、所决定。
3.1.2ksw_align2(qe - qb, (uint8_t*)query + qb, re - rb, rseq, 5, opt->mat, opt->o_del, opt->e_del, opt->o_ins, opt->e_ins, KSW_XSTART, 0):比较query和reference的匹配质量。返回给x,我们将x.score作为最终的输出。
2.输入参数:
const mem_opt_t *opt,
const bntseq_t *bns,
const uint8_t *pac,
int l_query,
const uint8_t *query,
int n_chn,
mem_chain_t *a
3.输出
mem_chain_v chn
3.2.4mem_chain2aln():将chain映射到真实的reference区域region
包含了基于动态规划匹配的ksw算法。
输入:
const mem_opt_t *opt,~运算中的匹配数据。alignment parameters
const bntseq_t *bns,~Information of the reference
const uint8_t *pac,~2-bit encoded reference
int l_query,~length of query sequence
const uint8_t *query,~query sequence
const mem_chain_t *c,
mem_alnreg_v *av~返回的是描述该read在reference真实位置的对应信息
typedef struct {
int n, m, first, rid;//seed的个数
uint32_t w:29, kept:2, is_alt:1;
float frac_rep;
int64_t pos;
mem_seed_t *seeds;
} mem_chain_t;
4.mem_reg2aln():生成CIGAR信息和forward-strand position
从上面生成的alignment region里面生成CIGAR信息和forward-strand position,并打印到屏幕上,其中没有太多实现逻辑。
* @param opt alignment parameters
* @param bns Information of the reference
* @param pac 2-bit encoded reference
* @param l_seq length of query sequence
* @param seq query sequence
* @param ar one alignment region
*
* @return CIGAR, strand, mapping quality and forward-strand position
*/
算法结构基本上就是这些。
总结一些问题,将代码从CPU转移到GPU的过程中,存在内省排序的调整限制性问题。
1.内省排序:introsort
内省排序遵循先快排,稍微有序之后堆排序,最后基本有序插入排序的策略。这样排序的优点在于效率最高,但是并不适合在GPU上进行运算,但是该排序属于不稳定排序,直接在GPU上替换成比较排序并不可行。因此可以考虑仍然在CPU上进行运算,将运算结果进行积攒并合并拷贝。
这篇关于BWA-MEM算法结构分析的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!