本文主要是介绍[生物医学文本挖掘]利用文本特征用于提取文献中药物之间的关系,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
背景
今年三月份准备投个会议文章,参考了一篇生物医学文本挖掘(BioNLP) 的文章,非教育网的朋友请点击百度云查看。这篇文章在之前实验室的journal club 论文交流的时候讲过,这里把ppt贴上,ppt总结了文章的主要内容,欢迎大家批评指正。
第一页:文章标题
姓名信息啥的删了 #(^_^)#
第二页:文章基本信息
发表不到一年,引用29次,文章还可以,但是仔细读完后发现文章还是能进行改进的。
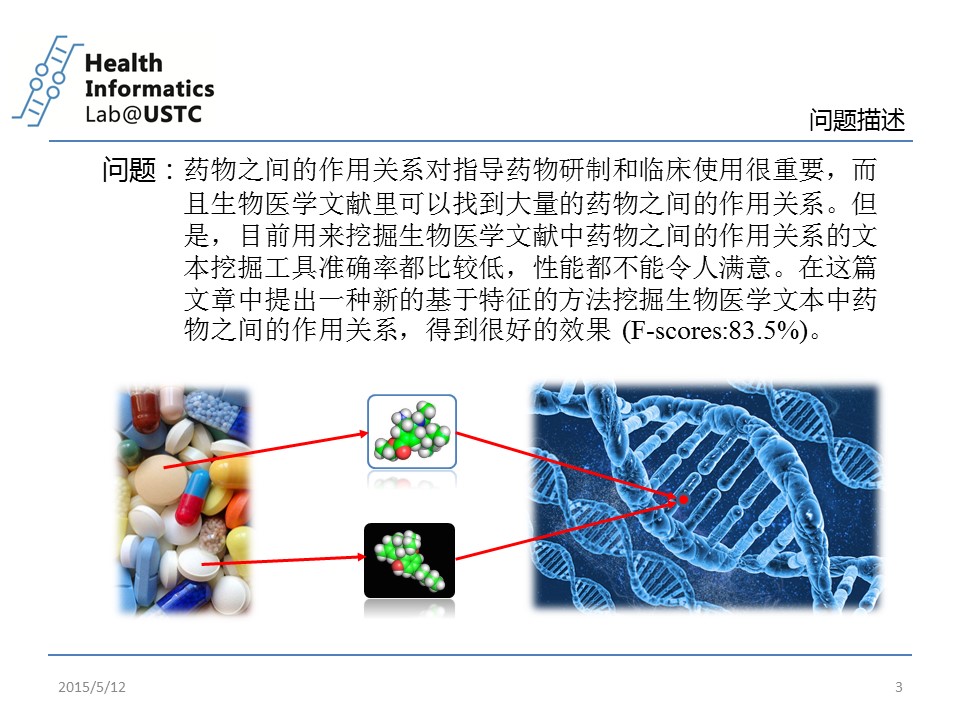
第三页:问题描述+背景
介绍了一点生物背景,本科学微电子的,现在转做生物大数据处理,没生物背景,简单介绍下,有错误欢迎指正。
第四页:研究现状
就是文章里introduction的主要东西。
第五页:文章的转折点
讲文章为什么会提出这种新的基于特征的方法,思路就来自这里。
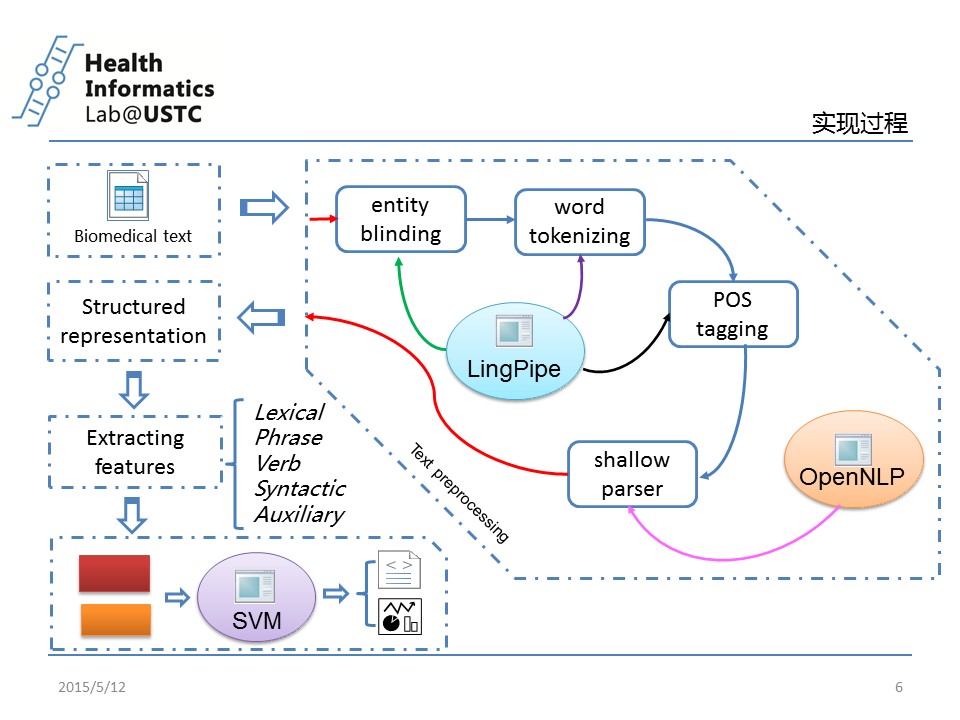
第六页:workflow
文章的主要流程,看了几遍文章画出来的。
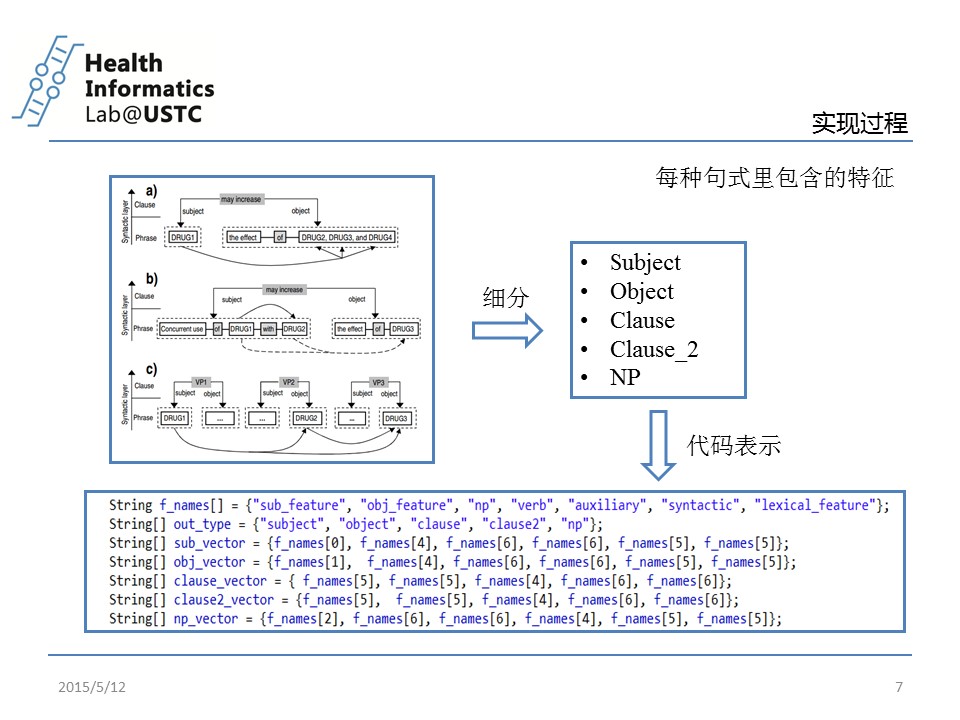
第七页:文章最重要的地方之一
用5种常用句式加上不同句式的不同特征生成特征向量,由于作者总结的句式不能涵盖所有的情况,这个对方法的性能有影响,在conclusion里作者也做了分析说明。
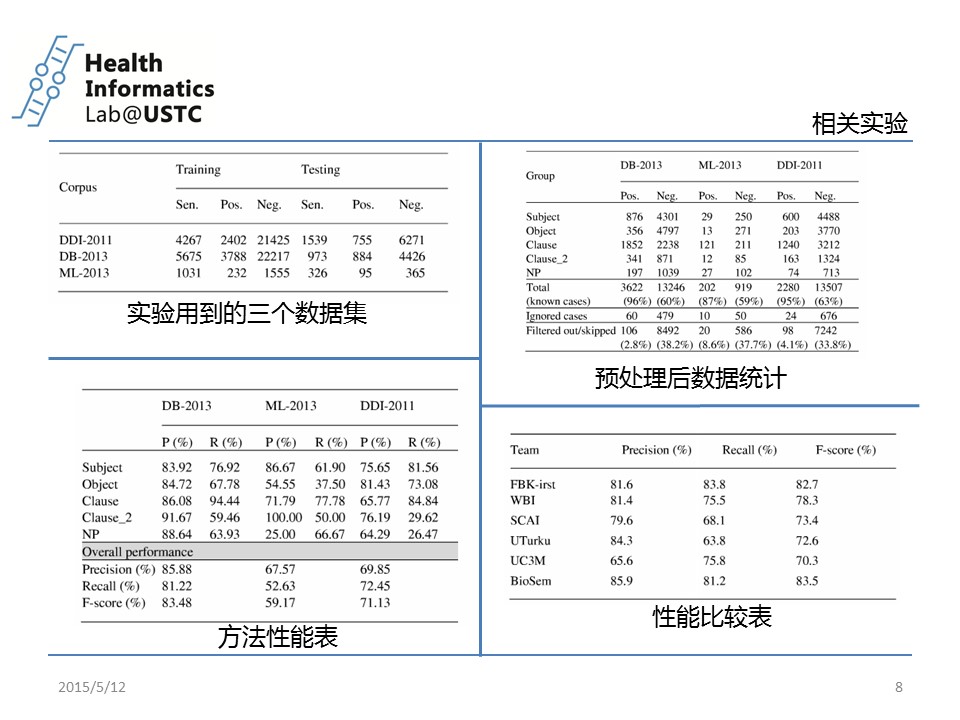
第八页:性能分析
列出了一些性能分析,这里就简单贴几个,具体可以看文章。
结束语
总结下自己的感受。
1. 看文献要抓重点,一看就明白。
2. 生物医学文本挖掘入门快,想做东西也快,做的人也不多,自己还要多努力。
这篇关于[生物医学文本挖掘]利用文本特征用于提取文献中药物之间的关系的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!