本文主要是介绍Universal Model:基于clip驱动的器官分割和肿瘤检测通用模型,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
论文:https://arxiv.org/abs/2301.00785
我看这篇主要是看看MRI的多模态融合方法的,所以会略一些东西,感兴趣细节的就翻原文好嘞
摘要
越来越多的公共数据集在自动器官分割和肿瘤检测方面显示出显著的影响。然而,由于每个数据集的大小和部分标记问题,以及对不同类型肿瘤的有限研究,所得到的模型通常仅限于分割特定的器官/肿瘤,而忽略了解剖结构的语义,也不能扩展到新的领域。为了解决这些问题,我们提出了CLIP驱动的通用模型,该模型将从对比语言图像预训练(CLIP)中学习到的文本嵌入结合到分割模型中。这种基于clip的标签编码捕获解剖关系,使模型能够学习结构化特征嵌入并分割25个器官和6种肿瘤。所提出的模型是从14个数据集的集合中开发出来的,使用总共3410个CT扫描进行训练,然后对来自3个额外数据集的6162个外部CT扫描进行评估。我们在医学细分十项全能(MSD)公共排行榜上排名第一,并在Beyond The Cranial Vault(BTCV)上取得了最先进的成果。此外,与特定数据集的模型相比,通用模型的计算效率更高(快6倍),可以更好地推广到不同地点的CT扫描,并且在新任务上显示出更强的迁移学习性能。
背景
挑战
1.标签不一致
(i)索引不一致。同一器官可标记为不同的指标。例如,胃在BTCV中被标记为“7”,但在word中被标记为“5”。
(ii)名称不一致。如果多个标签指的是相同的解剖结构,命名可能会令人困惑。例如,AMOS22中的“后腔静脉”和BTCV中的“下腔静脉”。
(iii)背景不一致。如胰腺ct与msd -脾合并时,在msd -脾中胰腺被标记为背景,而本应被标记为前景。
(iv)器官重叠。各种器官之间有重叠。例如,“肝血管”是“肝”的一部分,“肾肿瘤”是“肾”的一个子卷。(v)数据重叠。一些CT扫描在公共数据集之间重叠,但有不同的注释。例如,KiTS是腹横切t -1K的一部分,肾肿瘤在KiTS而不是腹横切t -1K中标注。第二,标签正交性。
2. 标签正交性。
大多数使用单热标签训练的分割方法[91]忽略了类之间的语义关系。给定肝脏[1,0,0]、肝肿瘤[0,1,0]和胰腺[0,0,1]的单热标签,肝↔肝肿瘤和肝↔胰腺之间没有语义上的区别。一种可能的解决方案是使用few-hot标签[64],肝脏、肝肿瘤和胰腺可以用[1,0,0]、[1,1,0]和[0,0,1]进行编码。虽然很少有热标签可以表明肝脏肿瘤是肝脏的一部分,但器官之间的关系仍然是正交的。(?)
为了解决上述问题,clip驱动的通用模型引入了文本嵌入,并采用了带二值分割掩码的掩码反向传播机制。具体来说,我们维护从公共数据集集合派生的修订标签分类法,并在图像预处理期间为每个类生成二值分割掩码。在架构设计上,我们借鉴了Guo等人[20]的灵感,用CLIP1预训练的文本编码器生成的文本嵌入代替了一个或几个热点标签。图1展示了CLIP嵌入如何呈现器官与肿瘤之间的关系。
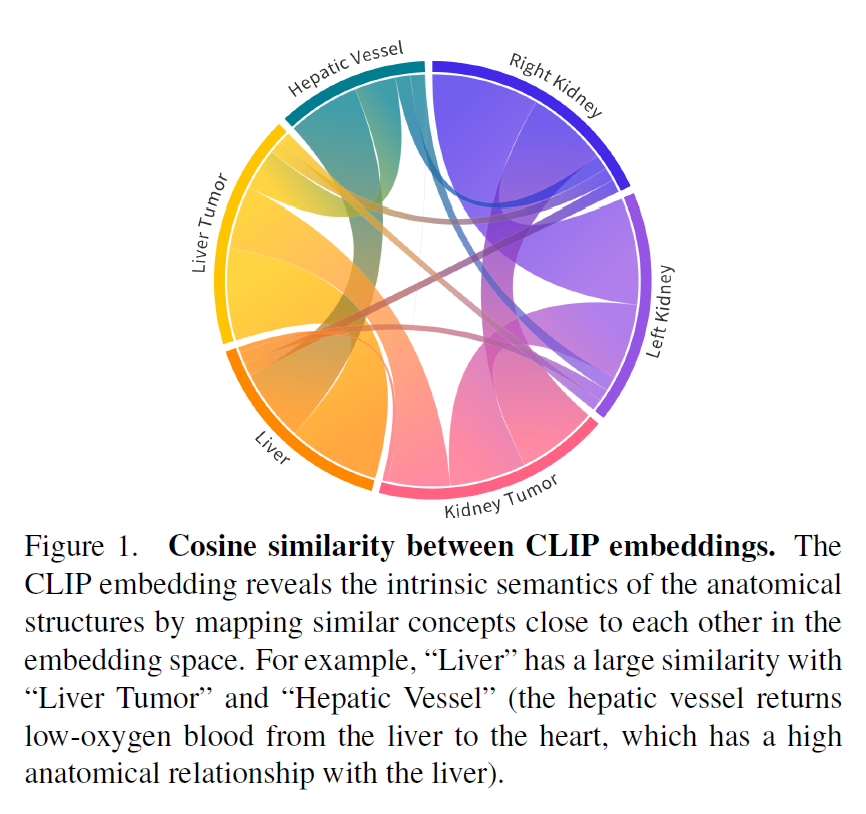
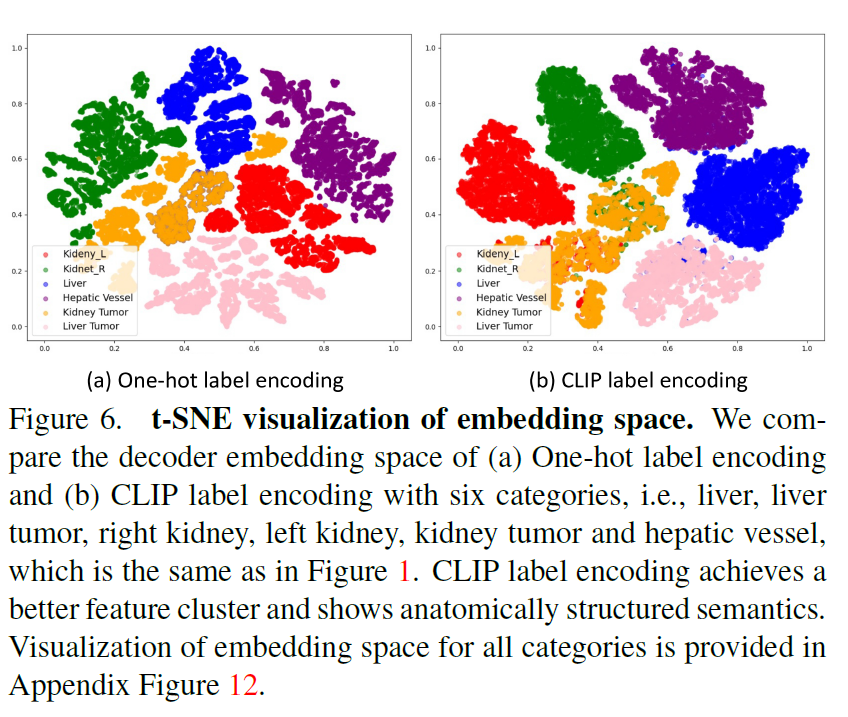
这种基于clip的标签编码增强了通用模型特征嵌入的解剖结构,如图6所示。最后,我们只计算具有可用标签的类的损失。
优点
1. 腹部器官分割性能高。此外,六个器官可以用通用模型注释,具有与人类相似的观察者内变异性。
2. 预测假阳性比现有模型少,同时保持肿瘤检测的高灵敏度。
3.在计算上比特定于数据集的模型更高效,测试速度提高了6倍。
4. 通用模型框架可以扩展到各种骨干网,如cnn和Transformers。器官分割和肿瘤检测的性能可以推广到来自各种医院的CT扫描,而无需额外的调整和适应。
6. 为众多下游任务提供了一个有效的基础模型,显示了跨多种疾病、器官和数据集的任务的强大可转移性。
相关工作
Partial label problem
公开可用的腹部成像数据集中在不同的器官和肿瘤上[36,45,44,32],例如,用于4个器官分割的腹大数据集[45],用于16个器官分割的WORD数据集[44]和用于104个解剖结构分割的TotalSegmentor数据集[79]。当在这些数据集的组合上训练AI模型时,由于它们的标签分类不一致,就会出现部分标签问题。为了利用部分标签,已经研究了几种方法[96,18,91,92],旨在建立一个可以进行器官分割[41,12]和肿瘤检测[2,103,83,42,50,81,47]的单一模型。这些研究有以下局限性。(1)由于数据集组装的规模较小2,组装数据集的潜力并不令人信服。它们的性能类似于特定于数据集的模型,并且没有在官方基准上进行评估。(2)由于单热标签,器官与肿瘤的语义关系被丢弃。表1显示,引入CLIP嵌入是我们提出的框架的一个重要因素。
Organ segmentation and tumor detection
略
CLIP in medical imaging
我们是第一个将CLIP嵌入引入体素级语义理解医学任务(即分割)的团队之一,其中我们强调了解剖结构之间语义关系的重要性。
Medical universal models
据我们所知,还有更多的努力正在进行中[58,93]。因此,我们致力于通过积极维护GitHub页面来审查该领域的杰出研究↓
https://github.com/ljwztc/CLIP-Driven-Universal-Model/blob/main/documents/awesome.md
去看了看确实是该领域的一个小集合↑
方法
背景
problem definition
设M和N分别为待合并数据集的总数和组合数据集中的数据点。给定数据集D = {(X1,Y1), (X2,Y2),…, (XN,YN)},总共有K个唯一类。对于∀n ∈ [1,N],如果存在∀k ∈ [1,K] 个类在Xi中标签在Yi内,D是个全标记(fully labeled)数据集;否则,D是部分(partially labeled)标记的数据集。
Previous solution
针对部分标签问题,提出了两组解决方案。给定一个数据点Xn, n∈[1,n],目标是使用装配数据集DA = {D1,D2,…,DM},如果用Xn表示,模型可以预测所有K个类。

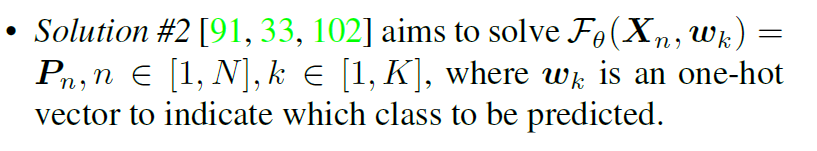
两种解决方法效果差不多但是2更高效
然而,这两种解决方案都依赖于一个热门标签,有两个共同的局限性。
首先,他们忽略了器官和肿瘤之间的语义和解剖关系。
其次,它们不适用于肿瘤的各种亚型的分割。为了解决这些限制,我们将解决方案#2中的工作修改为CLIP嵌入,并在以下部分中进行深入介绍。
CLIP-Driven Universal Model
clip驱动的通用模型的总体框架(参见图2)有一个文本分支和一个视觉分支。文本分支首先使用适当的医学提示为每个器官和肿瘤生成CLIP嵌入(表1),然后视觉分支同时使用CT扫描和CLIP嵌入来预测分割掩码3。
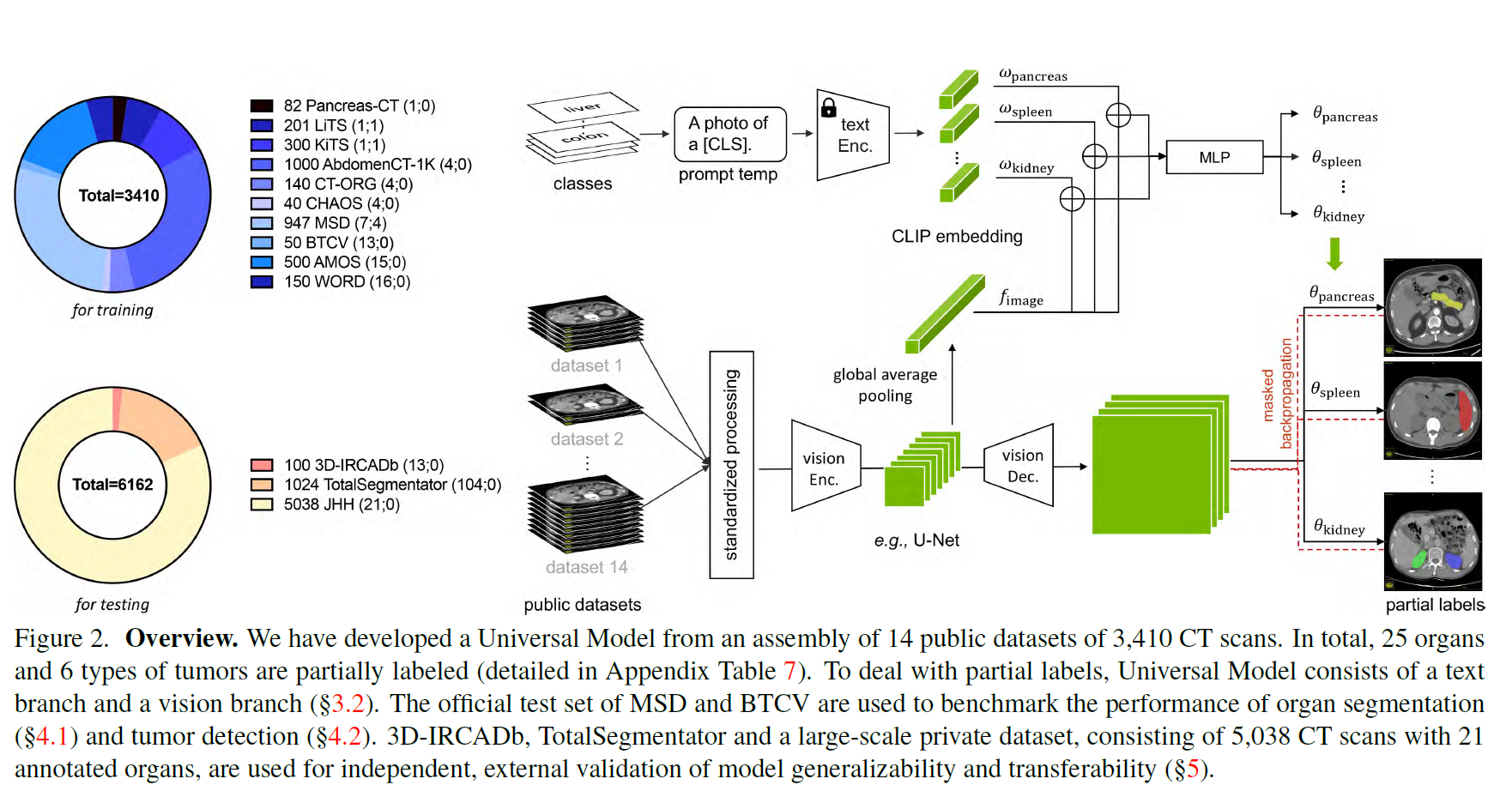
文本分支
设wk为第k类的CLIP嵌入,由CLIP中预训练的文本编码器和医疗提示(例如,“[CLS]的计算机断层扫描”,其中[CLS]是具体的类名)生成。
我们首先将CLIP嵌入(wk)和全局图像特征(f)连接(?)起来,然后将其输入到多层感知器(MLP),即基于文本的控制器[71],以生成参数(θk),即θk = MLP(wk⊕f),其中⊕为连接(?)。
尽管CLIP嵌入明显优于独热标签[91],但我们认为医学提示模板的选择至关重要。表1展示了三个提示模板的有效性。此外,CLIP嵌入的引入通过利用器官和肿瘤之间的语义关系来解决标签正交性问题(如图1所示)。

居然CLIP V3效果会比BioBERT好,,,我还准备用BioBERT做呢,不过不知道CLIP训练起来会怎么样.....不过这也是在多器官上的(后面有解释),,,单器官倒也真说不定

图像分支
我们使用各向同性间距和均匀强度尺度预处理CT扫描,以减少不同数据集之间的域间隙。标准化和归一化的CT扫描然后由视觉编码器处理。
设F为视觉编码器提取的图像特征。为了处理F,我们使用了三个具有1 × 1 × 1核的顺序卷积层,即文本驱动的分割器。前两层有8个通道,最后一层有1个通道,对应[CLS]k类。
类[CLS]k的预测计算为Pk = Sigmoid ((F∗θk1)∗θk2)∗θk3),其中θk = {θk1, θk2, θk3}在文本分支中计算,并且∗表示卷积。对于每个类[CLS]k,我们生成预测Pk∈R1×D×W×H,表示每个类的前景以一种方式与所有方式(即Sigmoid而不是Softmax)
好悲伤,,,发现它方法中并没有讲什么encode MRI数据并且和文本融合...容我一会翻一下代码补上
Masked back-propagation
为了解决标签不一致问题,我们提出了掩码反向传播技术。利用BCE损失函数进行监督。我们mask了这些不包含在Y中的类的损失项,只反向传播准确的监督来更新整个框架。掩码反向传播解决了部分标签不一致问题。具体来说,部分标记的数据集注释了一些其他器官作为背景,导致现有训练方案的失效(解决方案#1)。
实验
数据集和评估
总共收集了14个公开的3410个CT扫描数据集进行训练。另外两个公共数据集和一个私有数据集用于测试。数据集细节和预处理见附录§B。评估Dice Similarity Coefficient(DSC)和归一化表面距离Normalized Surface Distance(NSD)用于器官/肿瘤分割;敏感性和特异性(Sensitivity and Specificity)用于肿瘤检测。
细节设置
Universal Model 使用AdamW优化器进行训练,该优化器具有50个epoch的热身余弦调度程序(warm-up cosine scheduler)。分割实验使用每GPU 6个批处理大小,patch size为96 × 96 × 96。默认初始学习率为4e−4,动量为0.9,在多gpu(4)上的衰减为1e−5。该框架在monai0.9.05中实现。执行五重交叉验证策略。我们通过评估验证的最佳指标来选择每个折叠中的最佳模型。模型在8个NVIDIA RTX A5000卡上进行训练。
结果
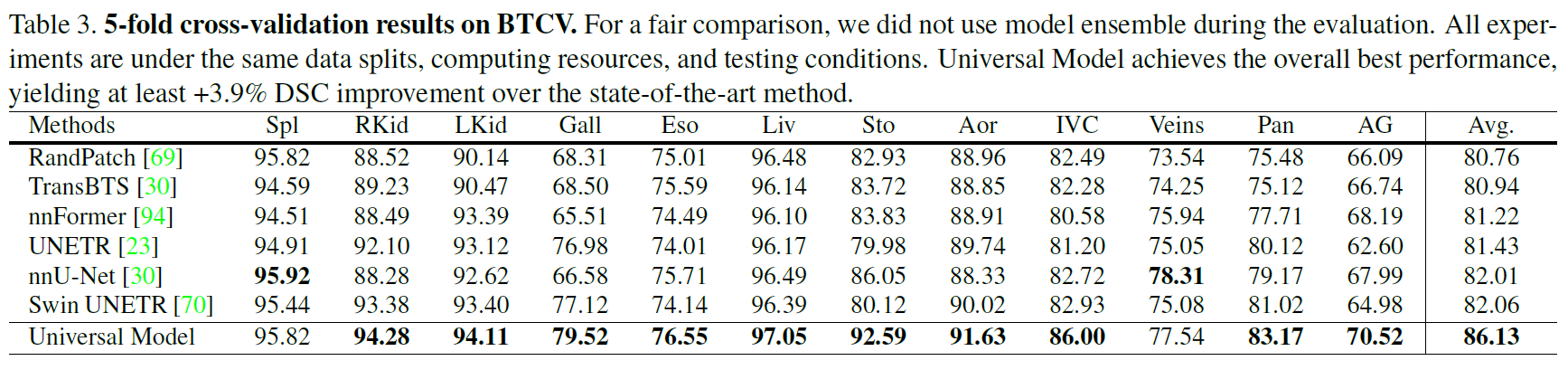
基于MSD和BTCV的器官分割
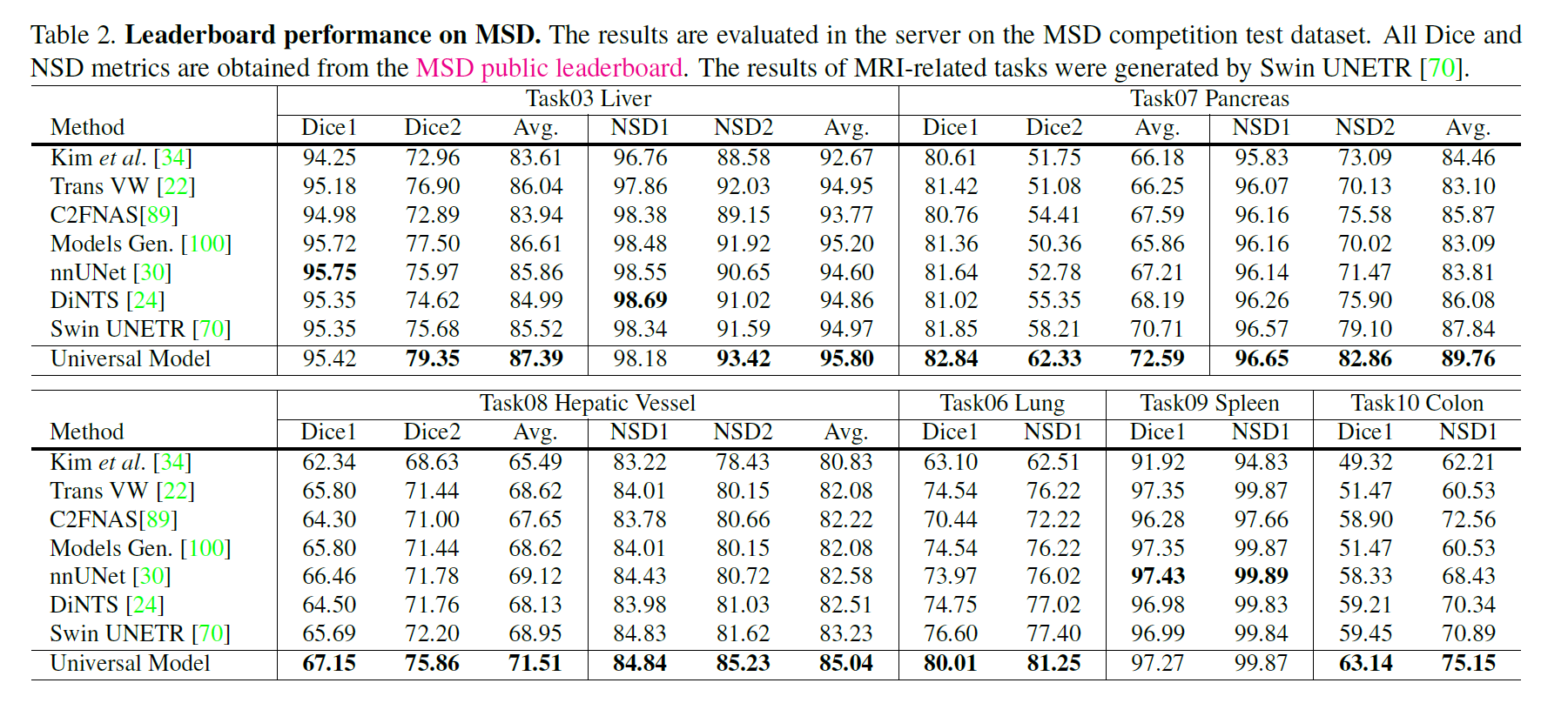

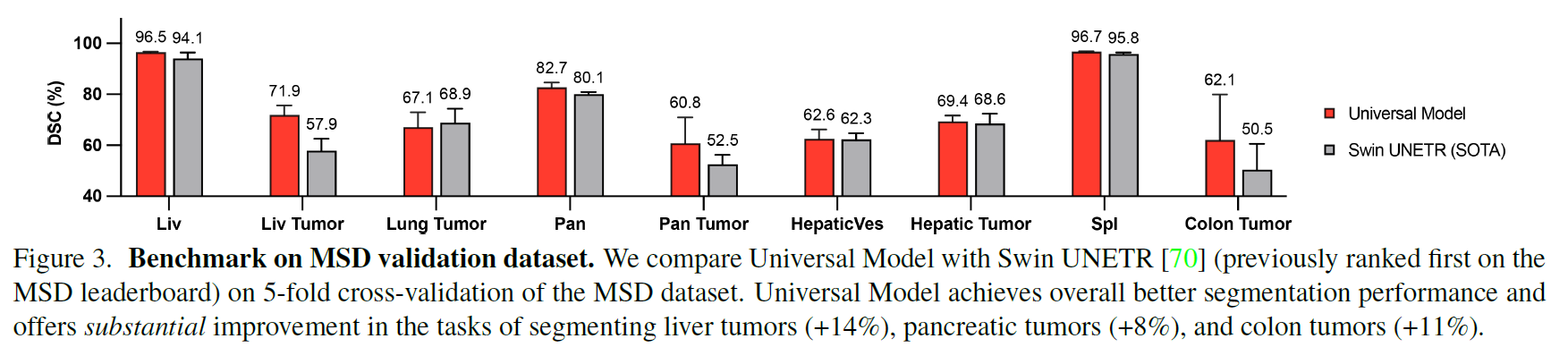
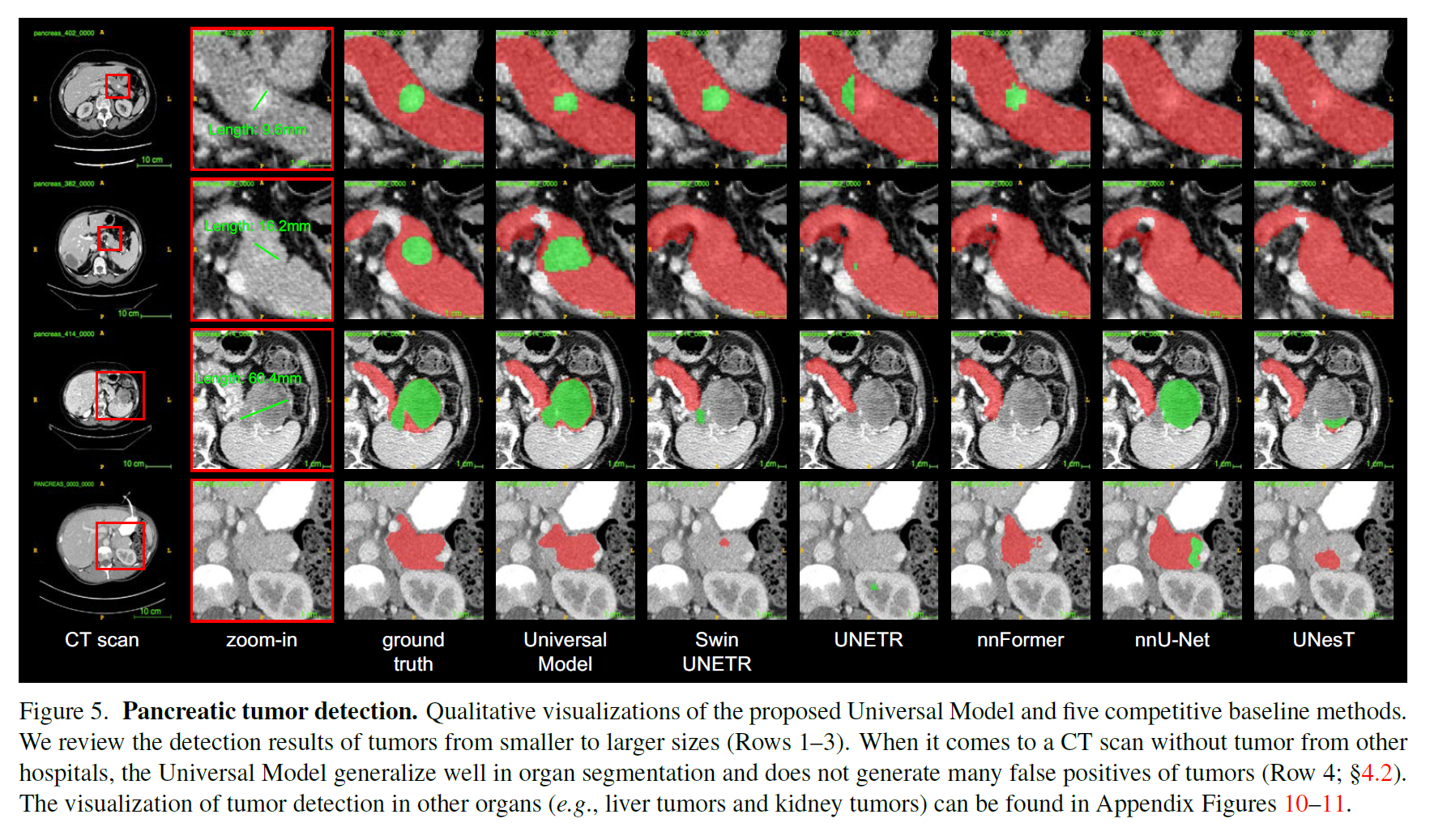
基于五个数据集的肿瘤检测
CLIP Embedding的高效性
我们在图6中进一步展示了单热编码和CLIP编码的嵌入空间的t-SNE可视化。我们可以看到CLIP编码的解码器嵌入表现出更好的特征聚类和解剖结构。
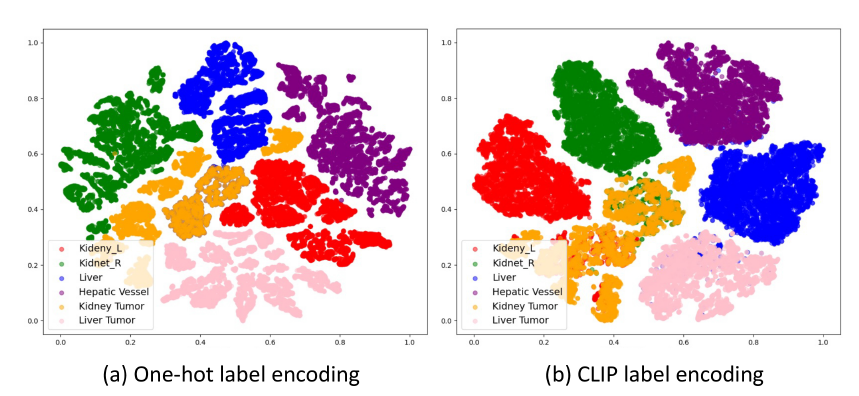
例如,右肾和左肾的特征是通用模型的嵌入空间更接近,即CLIP嵌套之间的余弦相似度高度匹配,如图1所示。这验证了基于clip的编码有助于模型捕捉解剖关系,学习结构化的特征嵌入。此外,我们还进行了包括BioLinkBERT嵌入在内的多种嵌入替代CLIP嵌入的消融研究[86],结果见附录表14。我们可以看到,与传统的单热标签(DoDNet[91])和纯文本预训练嵌入(BioLinkBERT[86])相比,基于clip的嵌入可以显著提高性能。
有趣的属性
效率
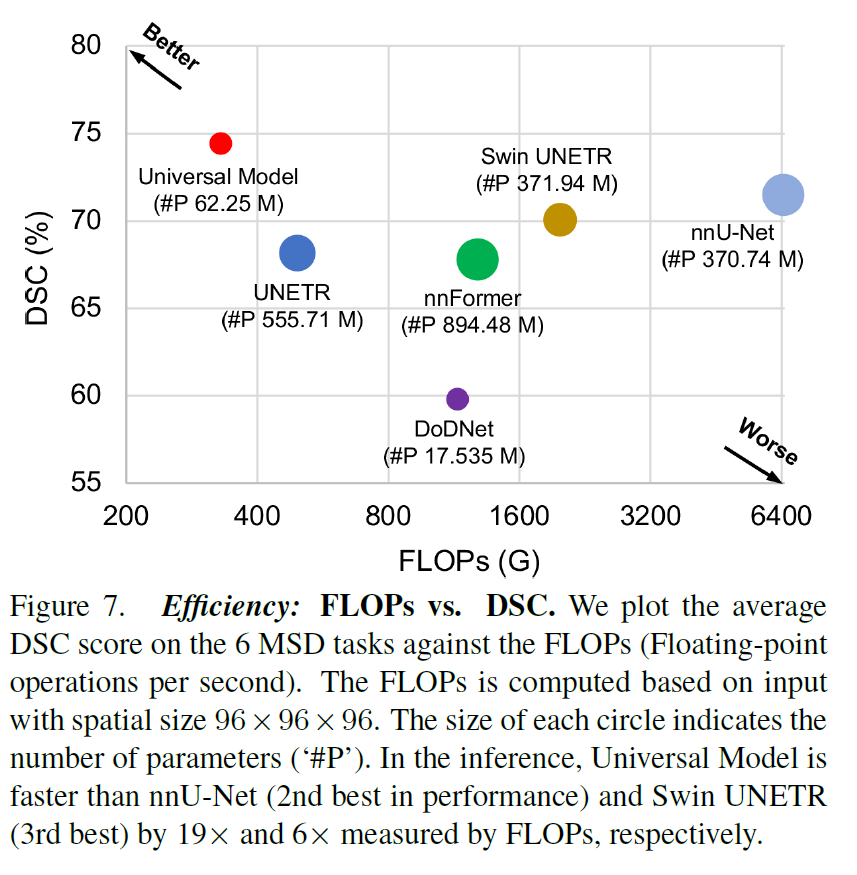
FLOPs vs. DSC。使AI模型更快在临床上具有重要意义[9,17]。使用每秒浮点运算数(FLOPS)来表示推理速度。图7给出了一个速度-性能图,显示与特定于数据集的模型相比,通用模型的计算效率更高(快6倍),同时保持平均74%的高DSC分数9。
可扩展性
灵活的backbone。拟议中的通用模型框架可以灵活地应用于其他主干。我们进一步以cnn为基础进行实验(如U-Net[61]), 25个器官和6个肿瘤的平均DSC评分为76.73%,与Swin UNETR的平均DSC评分76.11%相当,见表8
概括性
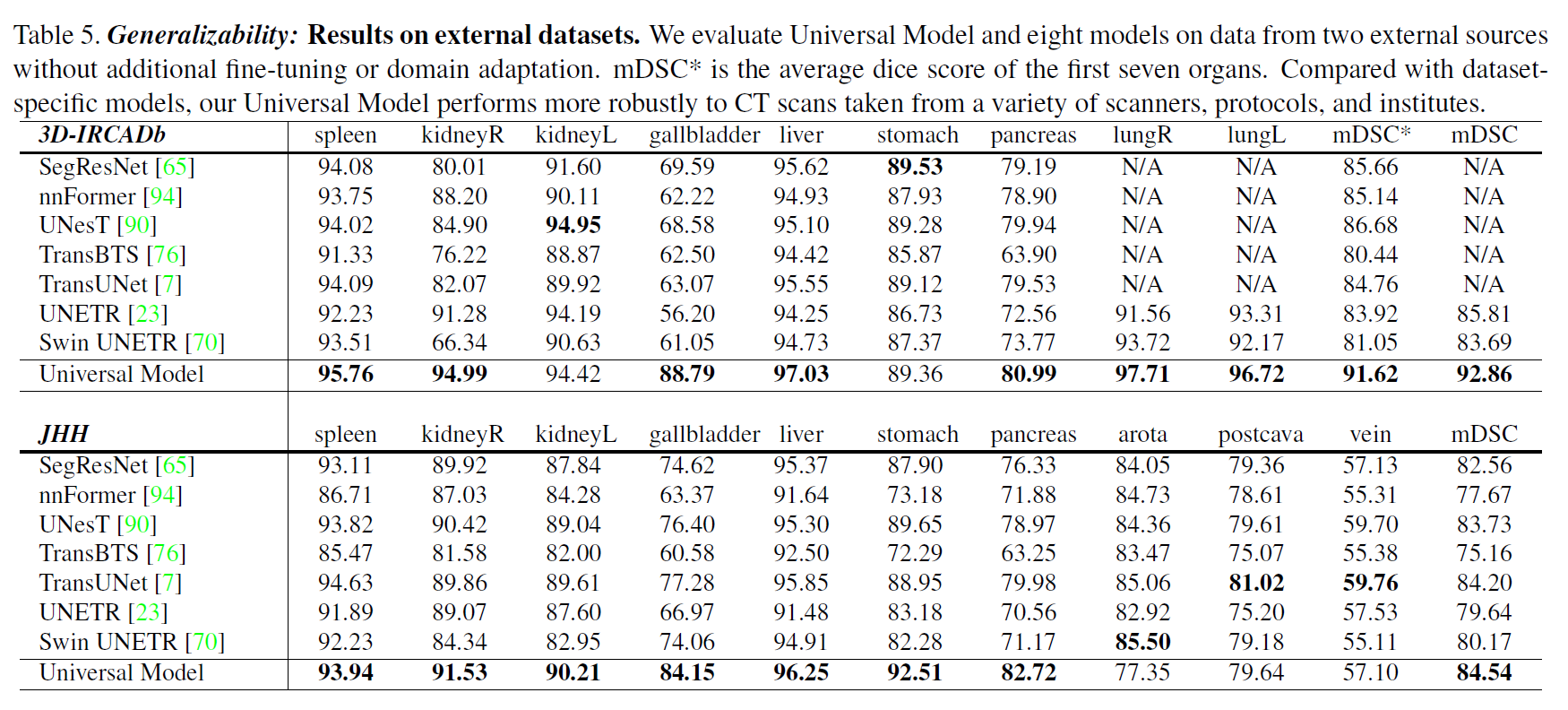
外部数据集的结果。
可转移性
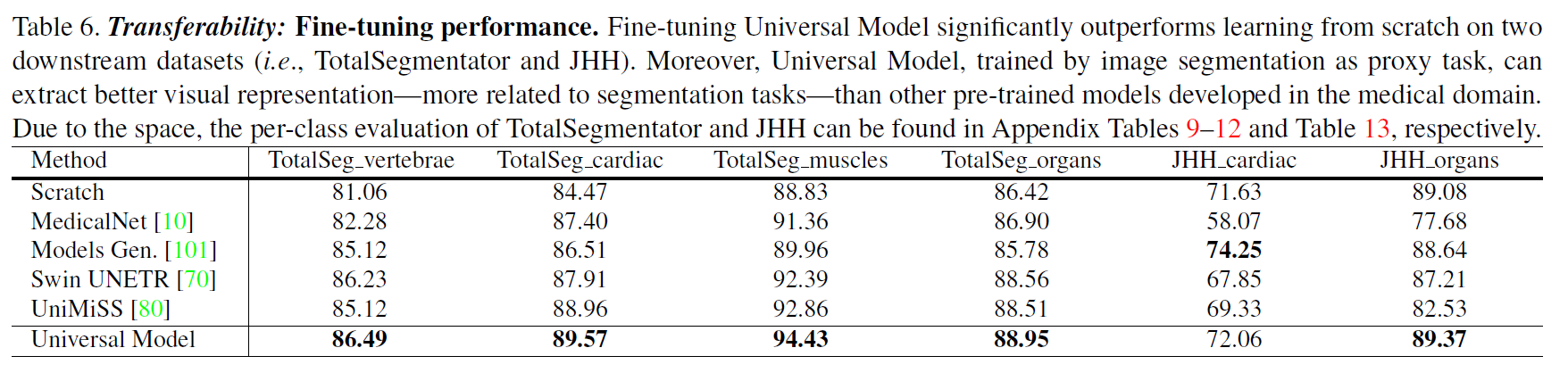
微调结果。通用模型可以作为一种强大的分割预训练模型。通过直接对装配数据集进行预训练,并对其他数据集进行微调,在TotalSegmentator数据集中的4个下游任务上,Universal Model的DSC达到了86.49%、89.57%、94.43%和88.95%,是其他预训练方法中DSC最高的(见表6)。
这篇关于Universal Model:基于clip驱动的器官分割和肿瘤检测通用模型的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!





