liver专题
3D Deeply Supervised Network for Automatic Liver Segmentation from CT Volumes
下面博主详细翻译了该篇论文,可以当做详尽的参考,并认真学习。 【参考】论文笔记:3D Deeply Supervised Network for Automatic Liver Segmentation from CT 数据集: MICCAI-SLiver07[1] 数据预处理: 作者没有讲数据预处理的过程。 CRF 轮廓精细修正: 参考上述博主博客。 参考文献: [1]Heimann,
Liver Segmentation in CT based on ResUNet with 3D Probabilistic and Geometric Post Process
一、摘要 本文提出了使用具有3D概率和几何后期处理功能的ResUNet的新型肝分割框架。 我们的语义分割模型ResUNet在U-Net的上采样和下采样部分添加了残差单元和批处理规范化层,以构建更深的网络。 为了快速收敛,我们提出了一种新的损失函数DCE,该函数由Dice损失和交叉熵损失线性组合。 我们使用连续的几个CT图像作为训练和测试的输入,以探索更多的上下文信息。 基于ResUNet的初始分割
3D Deeply Supervised Network for Automatic Liver Segmentation from CT Volumes-笔记
网络结构: 网络改进的地方是通过反卷积(核大小为3X3X3,步长为2)中间层,使得和输入大小相同,然后通过label计算loss,和最终loss一起进行梯度更新。中间反卷积层输出的loss可以防止梯消失。(深度监督DSN) 由于最终输出是通过反卷积得到的,只通过阈值生成结果,有时后边缘会不精确。所以在横断面又使用了CRF。 观察了中间层的输出:证明该论文的中间层学习到的表达相关性
肝移植笔记9 肝移植基础知识Discussion of liver transplantation-UCSF
加州大学作者: 关于肝移植的讨论由移植外科医生Kayvan Roayaie,M.D.,Ph.D.和Sandy Feng,M.D.,Ph.D.撰写,冯博士是UCSF移植学院的现任成员,也是UCSF腹部移植奖学金计划的项目主任。Roayaie博士是UCSF移植部门的前研究员和临床讲师。 https://transplantsurgery.ucsf.edu/conditions--procedure
Label-Free Liver Tumor Segmentation
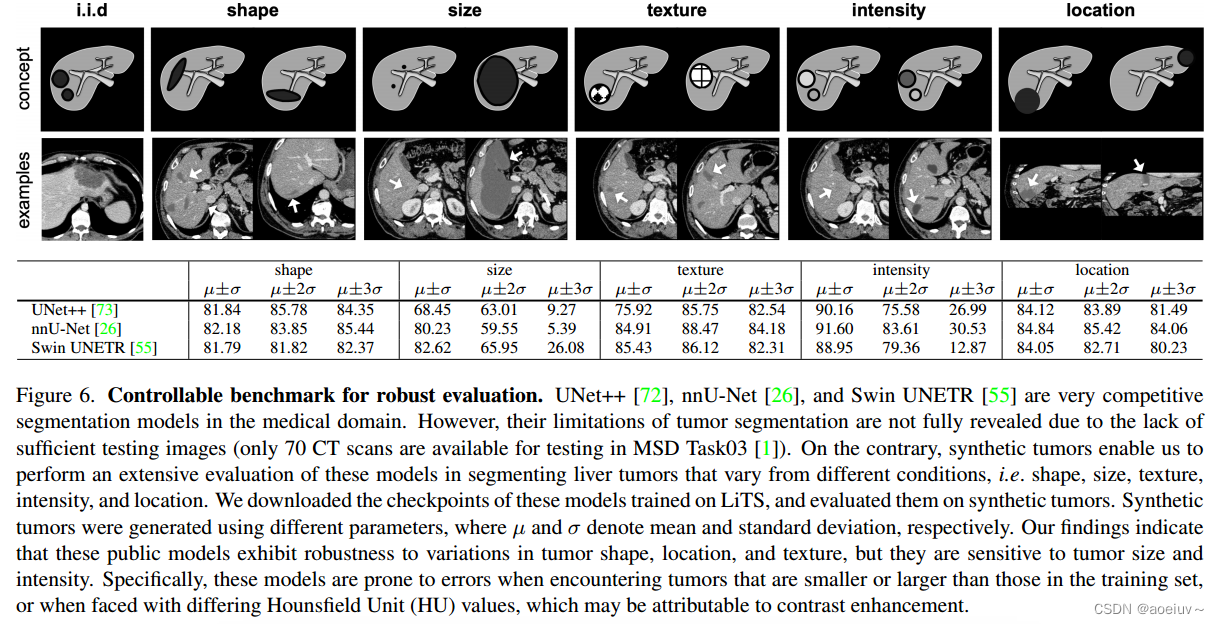
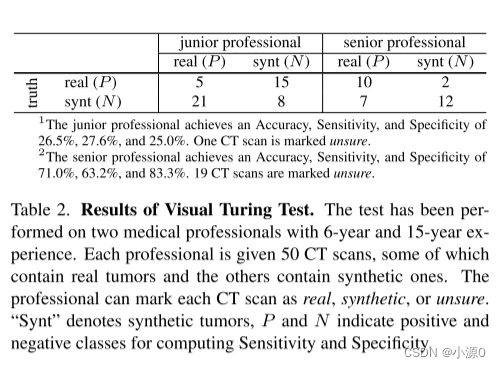
无标签肝肿瘤分割 作者单位:华中科技大学 & 香港中文大学-深圳 & 约翰霍普金斯大学 & 南京医科大学第一附属医院 发表于CVPR2023 摘要 通过在CT扫描中使用合成肿瘤,我们证明了人工智能模型可以准确地分割肝脏肿瘤,而无需人工注释。我们的合成肿瘤有两个有趣的优点:(1)形状和质地逼真,即使是医学专业人士也会将其与真实的肿瘤混淆;(II)有效地训练AI模型,该模型可以像训练真实肿瘤的模
Dense-PSP-UNet: A neural network for fast inference liver ultrasoundsegmentation
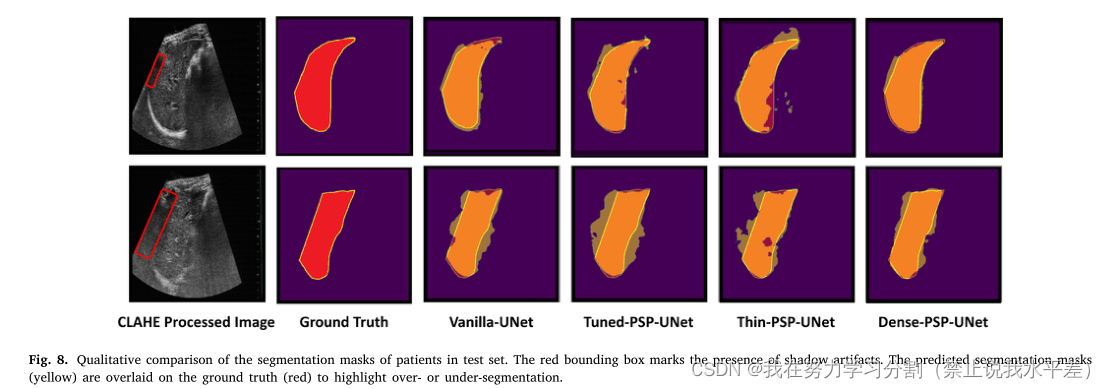
Dense-PSP-UNet:一种快速推理肝脏超声分割的神经网络 摘要:肝脏超声(US)或超声检查因其实时输出、低成本、易于使用、便携性和非侵入性而被广泛使用。实时肝脏超声分割对于诊断和分析肝脏疾病(如肝细胞癌),协助外科医生/放射科医生进行治疗程序至关重要。在本文中,我们提出了一种在调整后的神经网络骨干网中使用改进的金字塔场景解析(PSP)模块来实现实时分割而不影响分割精度的方法。考虑到美国数
阅读论文:Label-Free Liver Tumor Segmentation
论文标题:Label-Free Liver Tumor Segmentation 翻译:无标记的肝肿瘤分割 摘要 论文的目的:肿瘤合成,通过使用合成数据来改进医学图像分析和AI在肝脏肿瘤检测方面的性能 我们的主要贡献是合成了一种肿瘤生成器,它提供了以下五个优点: 1. 合成策略将医学知识嵌入可执行程序中,通过放射科医生和计算机科学家的协作,实现了逼真肿瘤的生成,无需手动标注成本。2. 整个