gene专题
基因调控网络(gene regulatory network-GRN)分析基础概念
基础背景: 染色质、转录因子和基因之间的相互作用产生了复杂的调控回路,可以表示为基因调控网络(gene regulatory nerworks,GRNs) 1. 染色质(Chromatin) 染色质是由DNA和蛋白质(主要是组蛋白)组成的复合结构。它是细胞核中DNA的主要存储形式。在真核细胞中,染色质的状态可以是高度压缩的异染色质或相对松散的常染色质,这种状态会影响基因的表达。 作
hdu 1080 ;poj 1080 Human Gene Functions
求两串字符串的匹配。类似于求最长公共子序列。不同的是:上下两串不同的匹配有不同的权值。 可以在两串加入‘-’, 开始题目理解有误,以为只能在长度较短的那串加入‘-’ 。说到底还是太水了。 这个题目的初始化需要注意一下 #include <iostream>#include<algorithm>#include<cstdio>#include<cstring>using name
hdu 1080 dp Human Gene Functions
分三种情况考虑。 dp[i][j]=max(dp[i-1][j-1]+a[s1[i]][s2[j]],max(dp[i-1][j]+a[s1[i]][4],dp[i][j-1]+a[s2[j]][4])); #include <iostream>using namespace std;int max(int a,int b){return a>b?a:b;}int main(
KEGG两类报错--①No gene can be mapped--②URL ‘https://rest.kegg.jp/link/hsa/pathway‘…
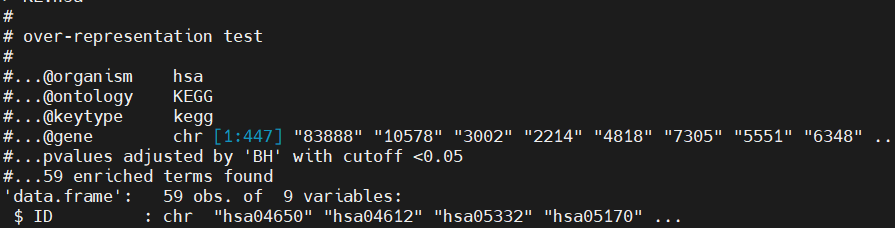
①No gene can be mapped 找到的相关链接处理办法如下: 用clusterProfiler包做KEGG富集分析遇到的问题 报错过程: 首先,我们知道进行KEGG分析之前需要转换基因名(SYMBOL->ENTREZ),更改后的基因部分如下所示: (以上可以排除基因不存在的错误因素) 出现错误 KEGG.res <- enrichKEGG(gene=sig.gen
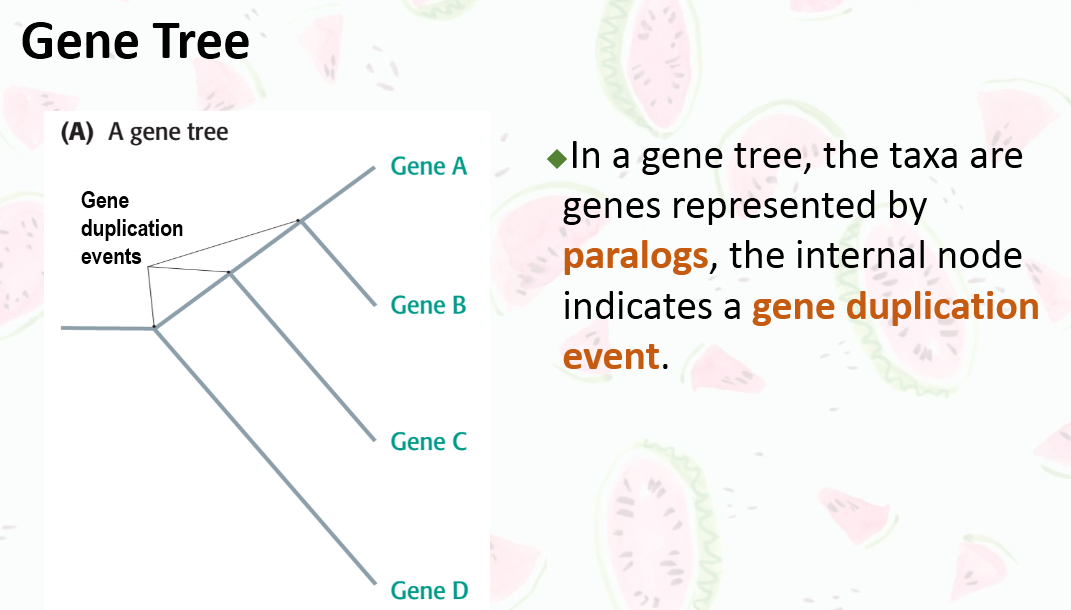
物种树(Species Tree)和基因树(Gene Tree)
认为来自不同物种的一组基因的进化关系(基因树,gene tree)与这些物种本身的进化关系(物种树,species tree) 一定相同,似乎是合理的,因为这些基因存在于这些物种的基因组中。但情况并非如此。基因树不一定与物种树相同。 基因树可以和物种树不同,一种情况如下图所示。祖先种群最先固定了A1等位基因(顶部),但随后发生了A2突变,
【Gene Expression Prediction】Part3 Deep Learning in Gene Expression Analysis
文章目录 6 第二个讲座:Deep Learning in Gene Expression Analysis6.1 Introduction6.2 D-GEX6.2.1 Connectivity map project6.2.2 Predicting gene expression from landmark genes 6.3 Deep generative models for geno
【Gene Expression Prediction】Part1 基因表达数据的获取与分析
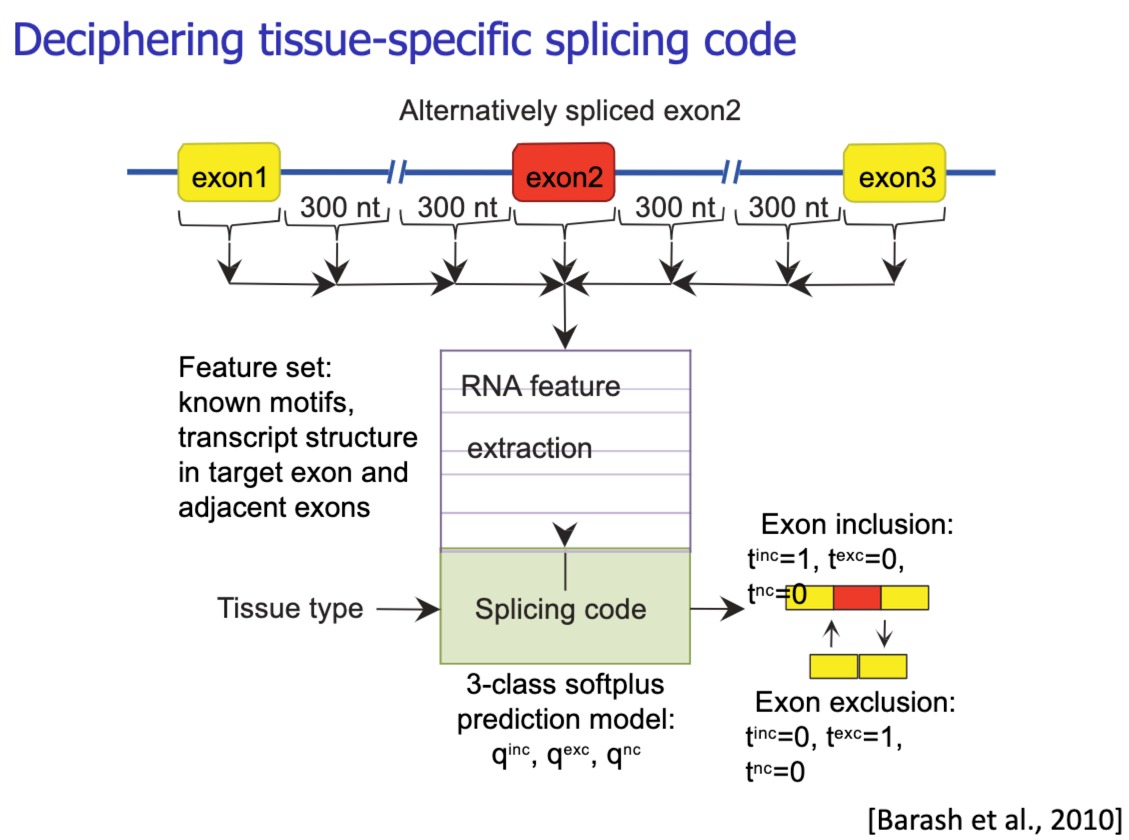
文章目录 Gene Expression Prediction1. Intro2. Up-sampling3. Compressive sensing3.5 Predicting Reporter Expression from Chromatin Features4. Predicting splicing from sequence 来自Manolis Kellis教授(MI
Lintcode 1900 · Gene Similarity [Python]
一开始当然是暴力了,结果绝对是MLE了。然后可以这么思考吧,双指针,先把两个串的数字量和字母转化为两对list。然后先对比两个数字,并减去两个数字的较小的数字量,然后判断此时,两个list数字的index对应的字母一样不,一样则说明对位的字母一样,res作为一样位置和字母的统计量,增加两个数字的较小的数字。不一样,则说明对位对比每一个对的上的。然后判断,此时两个数字减去较小数字后,哪一个为0,为0
自动控制原理与设计仿真练习(Gene.F.Franklin)
第二章:动态模型 1. 钟摆运动方程 问题描述:计算以下单摆运动在施加阶跃转矩 T e T_e Te后的角度运动方程。 采用牛顿第二定律计算: { T e r − m g sin θ ( t ) = d v ( t ) d t v ( t ) = ω ( t ) r ω ( t ) = d θ ( t ) d t 化简可得: T e − m g r sin θ ( t ) = m
SpaGCN: Integrating gene expression, spatial location and histology to identify spatial domains an
Title SpaGCN: Integrating gene expression, spatial location and histology to identify spatial domains and spatially variable genes by graph convolutional network SpaGCN是一种通过图卷积网络整合基因表达、空间位置和组织学信
2019年链圈黑马——Xrpalike Gene
2019年链圈黑马——Xrpalike Gene Xrpalike Gene系统是Xrpgeek团队历经多年的倾心研发的成果。 Xrpalike Gene系统是一个基于互联网的全球开放的支付网络。这是一个有别于SWIF的、便捷、安全、交易费用接近于零的支付模式。Xrpalike Gene系统继承了Ripple系统的技术逻辑,又延申了去中心化的扩展性,以实现不同于XRP系统的新领域运用:包含
Biomart gene_ID_mapping教程
http://www.ensembl.org/biomart/martview 1.Dataset选择 2.Filters(过滤和输入) 提交有Gene ID的文件 3.Attributes选择输出的参数(期待的输出参数) 4.Count可以看到匹配的基因数量(我原本的文件就是947个gene id) 5.Results输出结果(可导出文件)
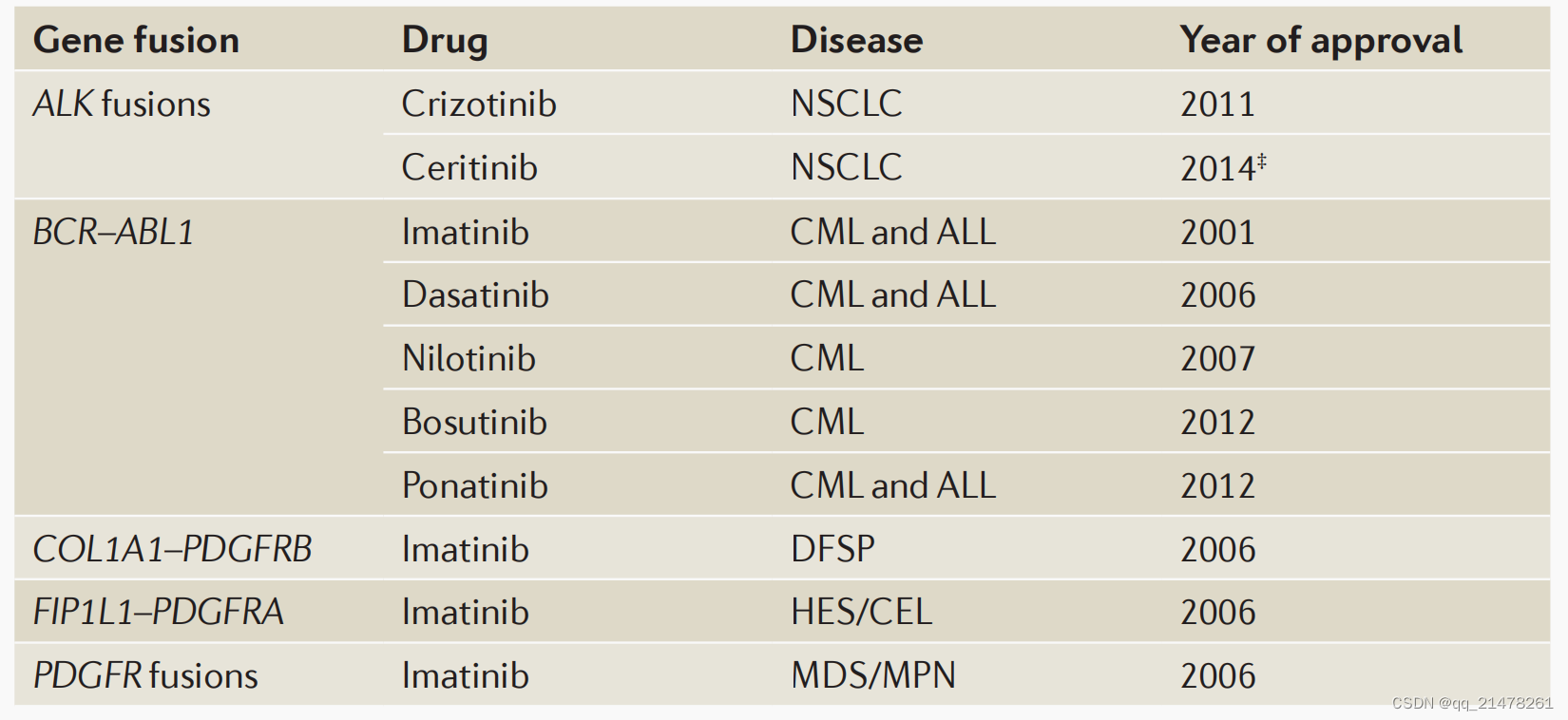
基因融合与癌症研究gene fusions in cancer
基因融合与癌症研究简历史(截止到2015年) Major discoveries from research on gene fusions and cancer 主要肿瘤亚型涉及的基因融合数量 Number of gene fusions involved in major neoplasia subtypes 恶性疾病中基因融合相关FDA批准药物 FDA-appr
Omics精进04|临床Gene Panel检测-实验生物信息学分析(无UMI标签)
本文介绍临床Gene Panel检测的实验流程及生物信息学分析流程(下图为MSK-IMPACT 468基因panel的检测流程,各产品流程也大同小异,本文参考此流程)。 本文目录 实验流程Panel探针合成与QC样本准备基因组DNA抽提与QC文库构建与QC液相杂交捕获靶向文库靶向文库高通量测序生物信息学分析流程(无UMI标签)Trimmomatic或cutadapt过滤FastQc