本文主要是介绍HE切片+qupath识别TIL和成纤维细胞文献(三阴性乳腺癌),希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
An Open-Source, Automated Tumor-Infiltrating Lymphocyte Algorithm for Prognosis in Triple-Negative Breast Cancer
An Open-Source, Automated Tumor-Infiltrating Lymphocyte Algorithm for Prognosis in Triple-Negative Breast Cancer - PubMed (nih.gov)
试验设计
尽管TIL评估进行了标准化工作,但评估的主观性和可变性阻碍了其广泛采用。使用QuPath开源软件,我们构建了一种基于H&E图像的TIL自动评估算法,并发明了一种超出人类能力的TIL评估方法。使用来自三个机构的一个发现集和四个验证集,我们发现机器读取测量的 TIL 变量将 TNBC 患者分为预后良好和不良队列,其中较高的 TIL 评分与更好的总生存期显着相关。这种开源的评估方法可以广泛使用,机器读取的 TIL 评分现在已准备好用于前瞻性测试,以证明临床实用性。
使用 QuPath 开源软件,我们为苏木精-伊红 (H&E) 染色切片上的肿瘤细胞、淋巴细胞、成纤维细胞和“其他”细胞构建了神经网络分类器。我们用五个独特的构造TIL变量分析了分类器衍生的TIL测量值。使用 171 例 TNBC 病例的回顾性集合作为发现集,以确定机器读取的 TIL 变量与患者结果的最佳关联。为了验证,我们评估了 749 名 TNBC 患者的回顾性集合,该患者由四个独立的验证子集组成。
数字图像分析
使用QuPath 开源软件平台(0.1.2 版)基于Watershed cell detection对图像中的细胞进行分割,设置如下:检测图像:苏木精 OD;要求的像素尺寸:0.5 μm;背景半径:8 μm;滤光片半径中值:0 μm;西格玛:1.5μm;最小细胞面积:10μm2;最大细胞面积:400μm2;阈值:0.1;最大背景强度:2.细胞扩增:5μm。细胞分割的质量控制由两名病理学家(DR 和 BA)进行。
细胞分类:将检测到的细胞分类为肿瘤细胞、免疫细胞 (TIL)、成纤维细胞和其他细胞。使用神经网络作为具有八个隐藏层(最大迭代次数:100)的机器学习方法。为了帮助算法进行准确的分类,我们还在半径为 25 μm 和 50 μm 处添加了平滑的物体特征,以补充单个细胞的现有测量值。

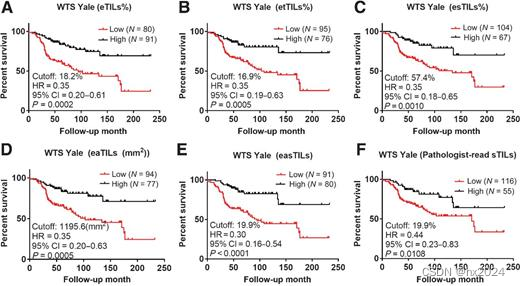
乳腺癌注释区域示意图和 H&E 图以及 QuPath TIL 变量的定义。A:标注的乳腺癌肿瘤区域。B: 每个 QuPath TIL 测量变量的计算公式,包括 eTILs%= 100 * [TILs 数量/(肿瘤细胞数量 + TILs 数量)];etTILs%= 100 * (TILs 数量/总细胞数量); esTILs=100*[TILs数量/(总细胞数量-肿瘤细胞数量)];eaTILs(mm2)= TILs数量/分析的肿瘤区域面积总和(mm2),easTILs=100*[TIL面积总和(mm2)/基质面积(mm2)]。高倍视图显示仅用苏木精和伊红染色的乳腺癌组织(C)或(D)与细胞分类掩膜重叠。标尺条代表 50 微米。细胞标注为肿瘤细胞(红色)、TIL(紫色)、成纤维细胞(绿色)和其他背景细胞(黄色)。
量化分析

算法训练流程图,发展到算法验证。A在上传 H&E 图像后首先定义估计污渍载体。然后使用标准化的分水岭细胞检测参数进行细胞分割。接下来,使用神经网络训练分类器,其中包含肿瘤细胞、TIL、成纤维细胞和其他细胞类型,每种类型都有颜色编码。构建了一个临时分类器,并将其应用于分类器训练集中的其余图像。经过多轮细胞分类审查和校正后,经过训练的分类器(CNN11)在形态学上被判断为在大多数图像上至少准确率为95%后,就会被锁定。B,应用经过训练的分类器产生了 TIL 测量值,其计算公式为以下 TIL 变量:eTILs%、etTILs%、esTILs%、eaTILs (mm2) 和 easTIL。在 WTS Yale(发现集)中使用 X-tile 软件确定的最佳切点确定了 TIL 变量与患者结果之间的关联。随后,所有 TIL 变量都在验证集中进行了测试,包括 TMA Yale1、TMA Yale2、WTS TCGA 和 WTS Sweden。C,解释如何在基于 H&E 图像的全组织图像中执行 TIL 定量的工作流程。肿瘤区域定义的步骤之后是估计染色载体以归一化 H&E 颜色。然后,使用标准化的分水岭细胞检测参数进行细胞分割,并使用经过训练的分类器进行细胞分类。最后,将TIL测量结果分析为构造变量。
相关指标计算:
-
1. eTILs% = 100 * [# TILs/(# 肿瘤细胞 + # TILs)];代表TIL在肿瘤细胞上的比例。
-
2. etTILs% = 100 *(# 的 TILs/# 的总细胞数);表示 TIL 在所有检测到的细胞中的比例。
-
3. esTILs = 100 * [# TILs/(# 总细胞 − # 肿瘤细胞)];代表 TIL 在基质细胞中的比例。
-
4. eaTILs (mm2) = # TILs/分析肿瘤区域面积之和 (mm2);表示肿瘤区域上 TIL 的密度。
-
5. easTILs = 100 * [TIL面积(mm2)/基质面积(mm2)之和];模仿病理学家读取的国际 TIL 工作组变量。
-
结果展示:
代码相关
作者提供:CNN11算法,Welcome to the Rimm Lab < Rimm Lab
该团队新文献:Image analysis-based tumor infiltrating lymphocytes measurement predicts breast cancer pathologic complete response in SWOG S0800 neoadjuvant chemotherapy trial
在github上提供了算法:CNN11 algorithm is deposited on GitHub: https://github.com/Yalaibai/Automated_QuPath_TIL_-Classifier_for-TNBC.git
相似研究文献:
An open source automated tumor infiltrating lymphocyte algorithm for prognosis in melanoma
Our TIL scoring algorithm for HE images of Melanoma has been deposited on GitHub: GitHub - acsbal/Automated-TIL-scoring-QuPath-Classifier-for-Melanoma: QuPath TIL classifier for Melanoma on HE slides. The algorithm can be used in QuPath platform. The QuPath software may be downloaded at QuPath.
参考文献:
1:An Open-Source, Automated Tumor-Infiltrating Lymphocyte Algorithm for Prognosis in Triple-Negative Breast Cancer
2:Image analysis-based tumor infiltrating lymphocytes measurement predicts breast cancer pathologic complete response in SWOG S0800 neoadjuvant chemotherapy trial
3:An open source automated tumor infiltrating lymphocyte algorithm for prognosis in melanoma
补充:
TCGA病理图像深度学习的肿瘤淋巴细胞浸润(文献)_tcga image-CSDN博客
HPAanalyze下载病理IHC然后qupath半定量分析(补全)-CSDN博客
这篇关于HE切片+qupath识别TIL和成纤维细胞文献(三阴性乳腺癌)的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!






