本文主要是介绍Geatpy数据结构,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
大部分数据存储在numpy的array数组中,默认array就是存储“矩阵”。
numpy的array在表示行向量时会有2中不同的结构:
一行n列的矩阵,它是二维的; //称为行矩阵
一种纯粹的一维行向量。 //称为行向量
不会使用超过二维的array;

在numpy中没有列向量的概念;
numpy在表现列向量时,实际上是二维的,但只有1列;
==可以通过print(变量.shape)输出其维度信息,以确定准确的维度;
一、基本数据结构
1.1 种群染色体
种群染色体是一个numpy的array类型的二维矩阵;一般用Chrom命名。每一行对应一个个体的一条染色体。默认Chrom的一行对应只有一条染色体;
种群的规模用Nind命名,种群的染色体长度用Lind命名,Chrom结构:

需要使用多种群时,可以使用多个chrom来存储各个种群的染色体;
1.2 种群表现型
也是numpy的array。一般用Phen命名。是种群染色体矩阵Chrom解码得到的基因表现型,每一行对应一个个体,一列代表一个决策变量。用Nvar表示变量的个数,种群表现型Phen的结构:

1.3 目标函数值
采用numpy的array类型矩阵存储种群的目标函数值。命名为objV,每一行代表一个个体,每一列对应一个目标函数,对于单目标函数,objV只有一列;
二元函数值矩阵:
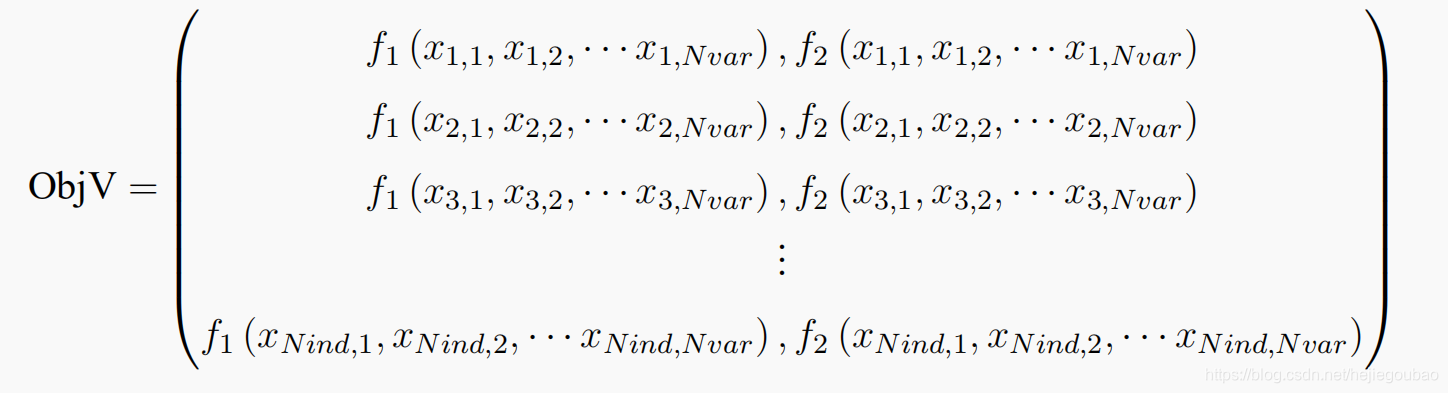
1.4个体适应度
采用列向量来存储种群个体适应度。命名为FitnV

1.5 违反约束程度
矩阵CV(Constraint Violation Value)来存储种群个体违反各个约束的程度。每一行代表种群的每一个个体;每一列代表一个约束条件,

CV矩阵的某个元素若小于或等于0,表示该元素对应的个体满足对应的约束条件。若大于0,表示违反约束条件,值越大,违反程度越高。
处理约束的两种方法:
- 罚函数法;
- 可行性法则;(需用到
CV矩阵)
1.6 译码矩阵
俗称区域描述器,用来描述种群染色体的特征,包括染色体中的每一元素所表达的决策变量的范围、是否包含范围的边界、采用二进制还是格雷码、是否使用对数刻度、染色体解码后所代表的决策变量是连续型还是离散型;
采用Geatpy提供的面向对象的进化算法框架时,译码矩阵可以与一个存储着种群染色体编码方式的字符串Encoding配合使用。
三种Encoding:
BG(二进制/格雷码)RI(实整数编码,即实数和整数的混合编码)p(排列编码,染色体每一位都是互异的)
RI和P都不需要解码,染色体上的每一位本身代表决策变量的真实值。这两种编码方式可统称为实数编码;
1.对于Encoding = 'BG'的种群,使用8行n列的矩阵FieldD作为译码矩阵,n代码染色体决策变量的个数,FieldD结构:
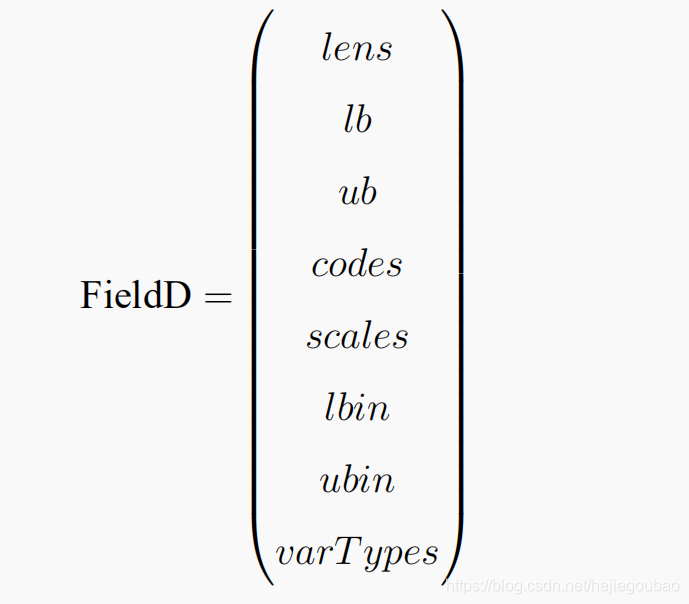
各变量均为长度等于决策变量个数的行向量;
lens包含染色体中的每一个子染色体的长度,sum(lens)等于染色体的长度;
lb和ub代表决策变量的上界和下界;
codes指明用的是二进制编码还是格雷编码;code[i] = 0表示第i各变量用的是二进制编码;codes[i] = 1表示使用格雷编码;
scales指明子串用的是算术刻度还是对数刻度。scales[i] = 0为算术刻度,1为对数刻度。对数刻度用于变量的范围较大而且不确定的情况,对于大范围的参数边界,对数刻度让搜索可用较少的位数,从而减少了遗传算法的计算量。(使用对数刻度,取值范围不能包括0);
lbin和ubin指明变量是否包含其范围的边界。0不包含,1包含;
varTypes指明决策变量的类型,0为对应位置决策变量是连续型变量,1为离散型;


2.对于实值编码的种群,使用3行n列的矩阵FeildDR来作为译码矩阵,n为染色体所表达的控制变量个数。结构为:

排列编码P的译码矩阵:要求FieldDR的第一行所有元素都相等,第二行所有元素也都相等,且第三行元素均为1(排列编码变量是离散的)。FieldDR有Lind列(即染色体长度为Lind)
要求上界 - 下界 + 1 >= Lind

创建FieldDR:

可以用内置的crtfld来方便的快速生成区域描述器;
1.7进化跟踪器
需要建立一个进化跟踪器(pop_trace)来记录种群在进化的过程中各代的最优个体,进化跟踪器的结构:
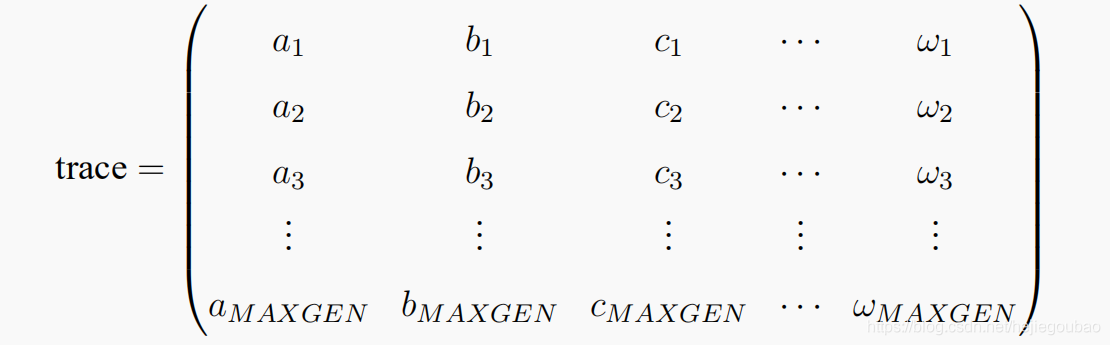
MAXGEN是种群进化的代数,每一列代表不同的指标,每一行代表一代;
另一种进化记录器是一个列表,每个元素都是拥有相同数据类型的数据。
二、种群结构
2.1 Population类
种群类(Population)是一个存储与种群个体相关信息的类。
基本属性:
size:int——种群规模,
ChromNum:int——染色体数目;
Encoding:str——染色体编码方式;
Field:array——译码矩阵,FieldD或者FieldDR
Chrom:array——种群染色体矩阵,每一行对应一个个体的一条染色体;
Lind:int——种群染色体长度
ObjV:array——种群目标函数值矩阵
FitnV:array——种群个体适应度列向量
CV:array——种群个体违反约束条件程度的矩阵
Phen:array——种群表现型矩阵
易错注意:
最容易出错的是目标函数矩阵ObjV以及CV矩阵的生成。ObjV和CV是在Problem类的目标函数接口aimFunc()计算生成的。计算的结果必须满足:ObjV和CV都是Numpy array类型矩阵,且行数等于种群的个体数目。ObjV的每一行代表一个个体,每一列代表一个优化目标。CV矩阵每一行对应一个个体,每一列对应一个约束条件。
用shape输出变量的维度信息:

2.2 PsyPopulation
PsyPopulation提供了Population类所不支持的多染色体混合编码。
基本属性:

这篇关于Geatpy数据结构的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!