本文主要是介绍UCSF DOCK 分子对接详细案例(04)-基于RDKit描述符的分子从头设计DOCK_D3N,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
| 欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 |
|---|
前言
本文是UCSF DOCK的使用案例分享,我们将使用DOCK 6.11自带的片段库,使用DOCK/RDKit的 DOCK_D3N功能从头设计分子。
一、 软件及操作环境
操作环境:Ubuntu 22.04
软件版本:UCSF DOCK 6.11,安装可以参考这篇博文;UCSF Chimera 1.17.3,UCSF ChimeraX 1.7.1,安装可以参考这篇博文。
在标准DOCK_DN设计中,分子基于用户定义的能量网格和/或扭转环境构建。目前还没有基于关键化学信息学描述符驱动分子构建的方法。在DOCK6.11版中,用户可以偏置分子生长,以促进部分生长的分子进入下一层生长,这取决于用户定义的描述符范围。
当每个部分生长的分子被传递到下一个生长层时,分子经历类似Metropolis的标准(Simulated Annealing算法获得全局最优解的核心思想),以评估计算的描述符的值是否落入基于输入文件的用户定义的范围内。如果他们通过标准,分子将立即被发送到下一层。然而,如果分子不符合标准,它们将被拒绝。由于该标准是基于Metropolis类程序,因此这些描述符的所得分布将在给定分子系综的用户定义的上限和下限附近具有软尾。
当DOCK使用RDKit(使用gnu.rdkit或gnu.parallel.rdkit配置)编译时,称为DOCK/RDKit,DOCK/RDKit利用RDKit中的多个化学信息学描述符。描述符中显示的所有描述符都将具有“RD_”前缀。主要4项不相干的描述符:定量的类药性质QED(0~1),可合成性Syntha(1-10),TPSA(0 ~ + ),logP。
DOCK_D3N与Generic de novo design, De Novo Refinement以及Fcused De Novo Design运行的却别在于对接程序使用的是:dock6.rdkit 或者dock6.rdkit_mpi。其主要思想展示在这张图:
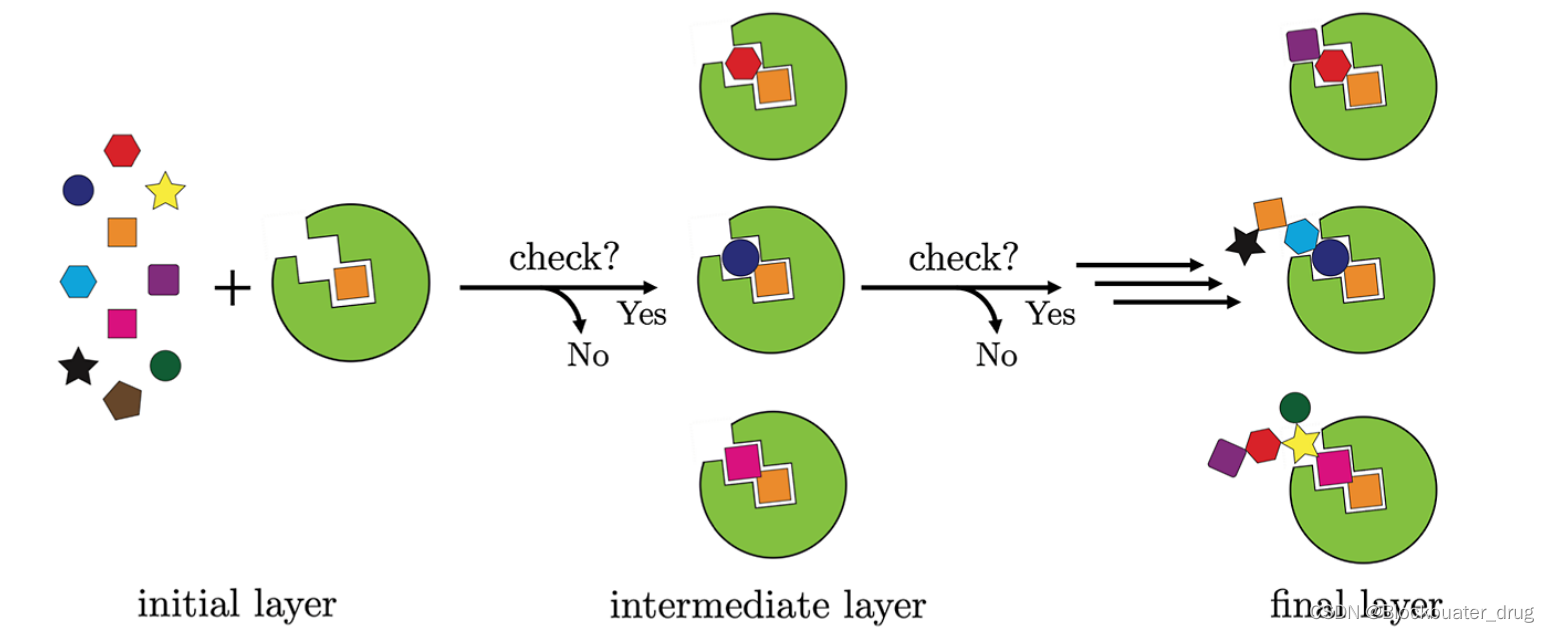
DOCK_D3N与Generic de novo design具有类似的设计流程,详细内容可参考DOCK使用手册,主要流程如下图。
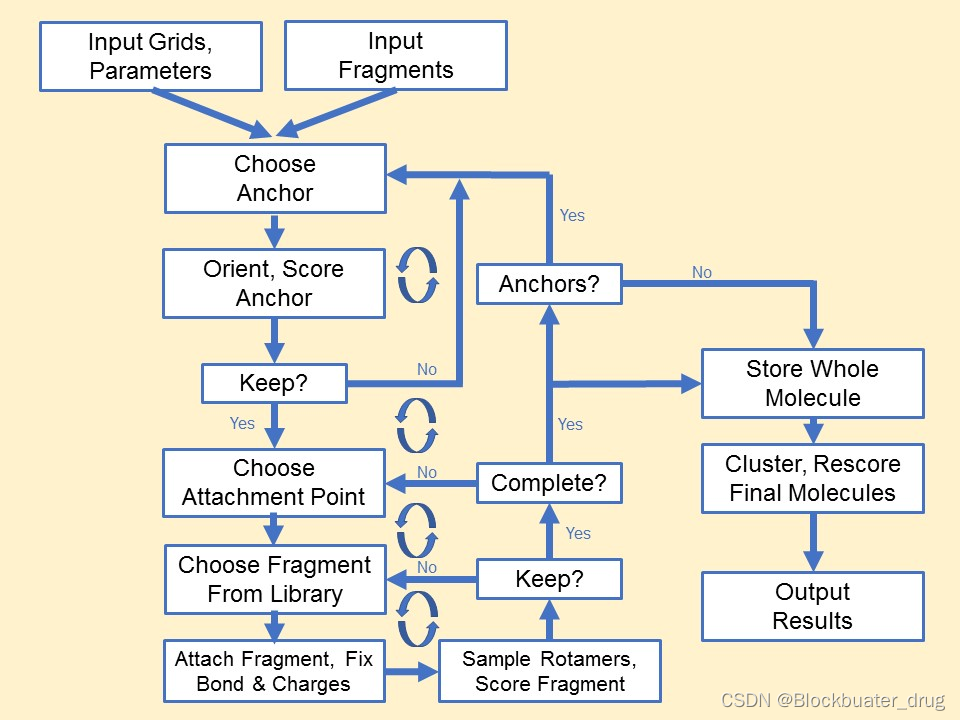
二、研究目的
NX-2127是一款具有双重活性的口服小分子靶向蛋白降解剂,可以布鲁顿氏酪氨酸激酶(BTK)蛋白。在与BTK结合的同时,NX-2127还可以招募E3泛素连接酶,使BTK蛋白“泛素化”,从而BTK被蛋白酶体降解。
在本实例中,我们将使用DOCK自带的片段库,使用DOCK/RDKit的 DOCK_D3N,基于配体-受体的结合信息,从头设计BTK抑制剂分子。
三、结构文件准备
本案例输入文件来自于前一篇案例分享:UCSF DOCK 分子对接详细案例(03)-分子从头设计de novo Design
也可以在此下载本案例需要的输入文件:下载案例(4)输入文件
新建本案例需要的目录:
cd DOCK_workdir
mkdir 012_denovo_D3N && 012_denovo_D3N
将此前建立的格点文件.bmp和.nrg文件复制到本目录:
cp ../002_surface_spheres/selected_spheres.sph ./
cp ../003_gridbox/grid.bmp ../003_gridbox/grid.nrg ./
cp ../010_denovo_refine/Chopped_ligand_for_denovo.mol2 ./
现在,012_denovo_D3N 内容如下:

四、 DOCK/RDKit中 de novo design
4.1 de novo design - refine_D3N
cd ./012_denovo_D3N
创建参数输入文件refine_D3N.in,准备输入参数:
touch refine_D3N.in
dock6.rdkit -i refine_D3N.in
输入后提示:
 参考以下的输入参数,完成引导的refine_D3N.in生成。
参考以下的输入参数,完成引导的refine_D3N.in生成。
conformer_search_type denovo
dn_fraglib_scaffold_file $DOCKHOME/parameters/fraglib_scaffold.mol2
dn_fraglib_linker_file $DOCKHOME/parameters/fraglib_linker.mol2
dn_fraglib_sidechain_file $DOCKHOME/parameters/fraglib_sidechain.mol2
dn_user_specified_anchor yes
dn_fraglib_anchor_file Chopped_ligand_for_denovo.mol2
dn_torenv_table $DOCKHOME/parameters/fraglib_torenv.dat
dn_name_identifier 8u2e_refine_D3N
dn_sampling_method graph
dn_graph_max_picks 30
dn_graph_breadth 3
dn_graph_depth 2
dn_graph_temperature 100.0
dn_pruning_conformer_score_cutoff 100.0
dn_pruning_conformer_score_scaling_factor 2.0
dn_pruning_clustering_cutoff 100.0
dn_mol_wt_cutoff_type soft
dn_upper_constraint_mol_wt 550.0
dn_lower_constraint_mol_wt 0.0
dn_mol_wt_std_dev 35.0
dn_drive_verbose no
dn_drive_clogp no
dn_drive_esol no
dn_drive_tpsa no
dn_drive_qed no
dn_drive_sa no
dn_drive_stereocenters no
dn_drive_pains no
dn_constraint_rot_bon 15
dn_constraint_formal_charge 2.0
dn_heur_unmatched_num 1
dn_heur_matched_rmsd 2.0
dn_unique_anchors 1
dn_max_grow_layers 9
dn_max_root_size 25
dn_max_layer_size 25
dn_max_current_aps 5
dn_max_scaffolds_per_layer 1
dn_write_checkpoints yes
dn_write_prune_dump yes
dn_write_orients no
dn_write_growth_trees no
dn_output_prefix output
use_internal_energy yes
internal_energy_rep_exp 12
internal_energy_cutoff 100.0
use_database_filter yes
dbfilter_max_heavy_atoms 200
dbfilter_min_heavy_atoms 0
dbfilter_max_rot_bonds 10
dbfilter_min_rot_bonds 0
dbfilter_max_hb_donors 5
dbfilter_min_hb_donors 1
dbfilter_max_hb_acceptors 10
dbfilter_min_hb_acceptors 2
dbfilter_max_molwt 500
dbfilter_min_molwt 200
dbfilter_max_formal_charge 1
dbfilter_min_formal_charge -1
dbfilter_max_stereocenters 5
dbfilter_min_stereocenters 0
dbfilter_max_spiro_centers 3
dbfilter_min_spiro_centers 0
dbfilter_max_clogp 5
dbfilter_min_clogp -3
dbfilter_max_logs 20.0
dbfilter_min_logs -20.0
dbfilter_max_tpsa 150
dbfilter_min_tpsa 50
dbfilter_max_qed 1.0
dbfilter_min_qed 0.0
dbfilter_max_sa 10.0
dbfilter_min_sa 1.0
dbfilter_max_pns 100
dbfilter_sa_fraglib_path $DOCKHOME/parameters/sa_fraglib.dat
dbfilter_PAINS_path $DOCKHOME/parameters/pains_table_2019_09_01.dat
orient_ligand no
bump_filter no
score_molecules yes
contact_score_primary no
grid_score_primary yes
grid_score_rep_rad_scale 1
grid_score_vdw_scale 1
grid_score_es_scale 1
grid_score_grid_prefix grid
minimize_ligand yes
minimize_anchor no
minimize_flexible_growth yes
use_advanced_simplex_parameters no
simplex_max_cycles 1
simplex_score_converge 0.1
simplex_cycle_converge 1.0
simplex_trans_step 1.0
simplex_rot_step 0.1
simplex_tors_step 10.0
simplex_grow_max_iterations 250
simplex_grow_tors_premin_iterations 0
simplex_random_seed 0
simplex_restraint_min no
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_de_novo.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
编写slurm运行脚本refine_D3N.sh,写入以下内容:
#! /bin/bash
#SBATCH --time=5:00:00
#SBATCH --nodes=1
#SBATCH --ntasks=10
#SBATCH --job-name=BTK_D3N
#SBATCH --output=BTK_D3Ndock6.rdkit_mpi -i refine_D3N.in -o refine_D3N.slurm.out
运行:
sbatch refine_D3N.sh
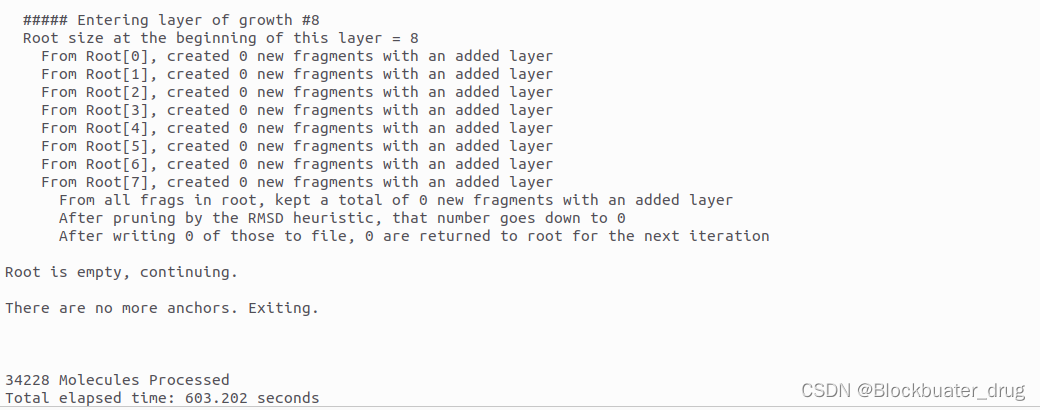
运行大约10 min。
结果文件:8u2e_refine_D3N.denovo_build.mol2,生成34288 个分子,可以通过Chimera或者ChimeraX的ViewDock查看。
4.2 对输出重新评分
通过4.1中的方式产生的分子已经通过grid score进行评分,可以进一步做rigid docking能量最小化优化,以此更加准确评价生成分子与受体之间结合能力。
生成空白的输入文件:
touch refine_D3N_min.in
运行:
dock6.rdkit -i refine_D3N_min.in
参照一下条件输入参数,最终生成运行文件。输入结束后,将继续运行以上命令。
conformer_search_type rigid
use_internal_energy yes
internal_energy_rep_exp 12
internal_energy_cutoff 100.0
ligand_atom_file 8u2e_refine_D3N.denovo_build.mol2
limit_max_ligands no
skip_molecule no
read_mol_solvation no
calculate_rmsd no
use_database_filter no
orient_ligand yes
automated_matching yes
receptor_site_file selected_spheres.sph
max_orientations 1000
critical_points no
chemical_matching no
use_ligand_spheres no
bump_filter yes
bump_grid_prefix grid
max_bumps_anchor 2
max_bumps_growth 2
score_molecules yes
contact_score_primary no
grid_score_primary yes
grid_score_rep_rad_scale 1
grid_score_vdw_scale 1
grid_score_es_scale 1
grid_score_grid_prefix grid
minimize_ligand yes
simplex_max_iterations 1000
simplex_tors_premin_iterations 0
simplex_max_cycles 1
simplex_score_converge 0.1
simplex_cycle_converge 1.0
simplex_trans_step 1.0
simplex_rot_step 0.1
simplex_tors_step 10.0
simplex_random_seed 0
simplex_restraint_min no
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_de_novo.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
ligand_outfile_prefix refine_D3N_min
write_orientations no
num_scored_conformers 1
rank_ligands no
或者,编写slurm运行脚本refine_D3N_min.sh,写入以下内容:
#! /bin/bash
#SBATCH --time=5:00:00
#SBATCH --nodes=1
#SBATCH --ntasks=10
#SBATCH --job-name=BTK_refine_D3N_min
#SBATCH --output=BTK_refine_D3N_mindock6.rdkit_mpi -i refine_D3N_min.in -o refine_D3N_min.out
slurm运行:
sbatch refine_D3N_min.sh
大约运行1 min。结果文件:refine_D3N_min_scored.mol2。
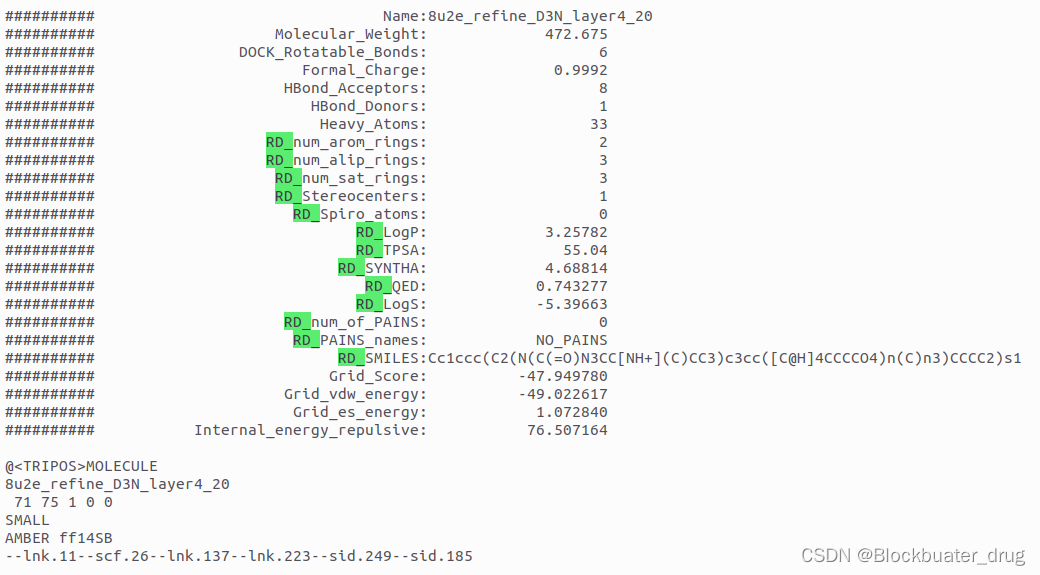 在以上演示条件下,生成获得39个分子,可以通过Chimera或者ChimeraX的ViewDock查看。
在以上演示条件下,生成获得39个分子,可以通过Chimera或者ChimeraX的ViewDock查看。
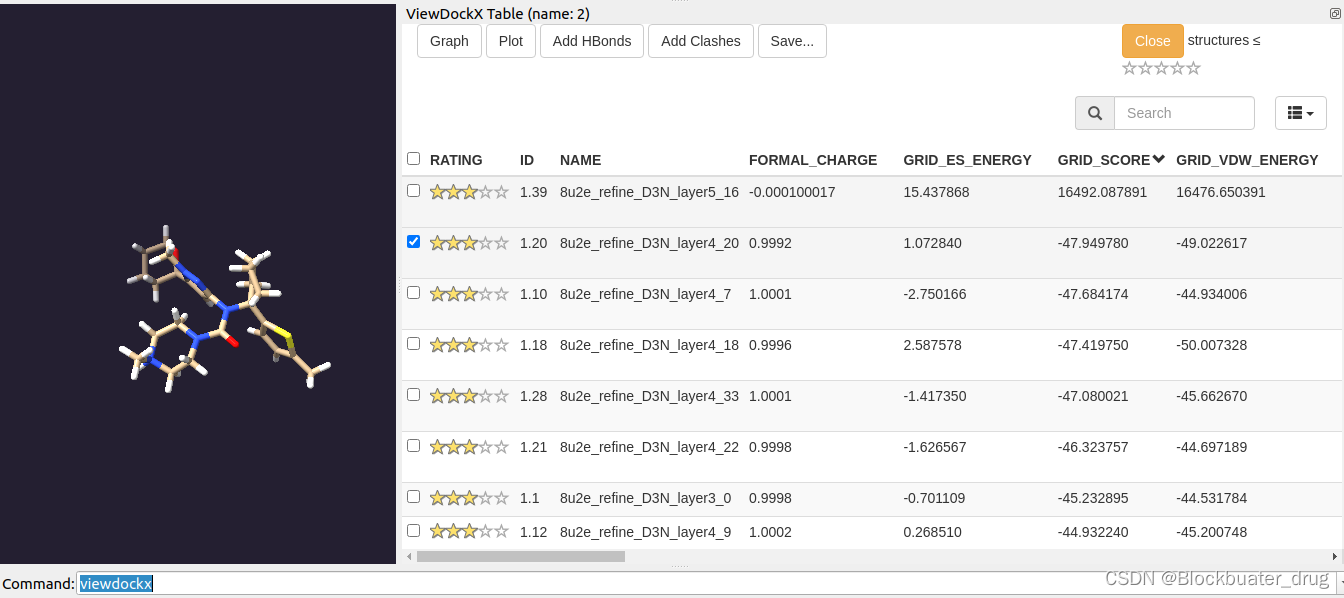
可以看到,采用自带片段库生成的一些分子与固有配体up9具有相似的空间基团连接方式;可通过进一步优化片段库得设置,得到更加多样化的新分子。
总结
在本实例中,我们使用DOCK的通用片段库,使用DOCK_D3N的方式,从头开始为我们的受体构建新的配体,这只是一个运行案例,在实际项目任务中,需要进一步优化加载的片段库、设置合适的片段生长方式、过滤条件等,不断生成接近预期的目标新分子。
参考资料
- https://ringo.ams.stonybrook.edu/index.php/Main_Page
- https://dock.compbio.ucsf.edu/DOCK_6/dock6_manual.htm
- http://dx.doi.org/10.1021/acs.jcim.3c01031
| 欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 |
|---|
这篇关于UCSF DOCK 分子对接详细案例(04)-基于RDKit描述符的分子从头设计DOCK_D3N的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!




