rdkit专题
UCSF DOCK 分子对接详细案例(04)-基于RDKit描述符的分子从头设计DOCK_D3N
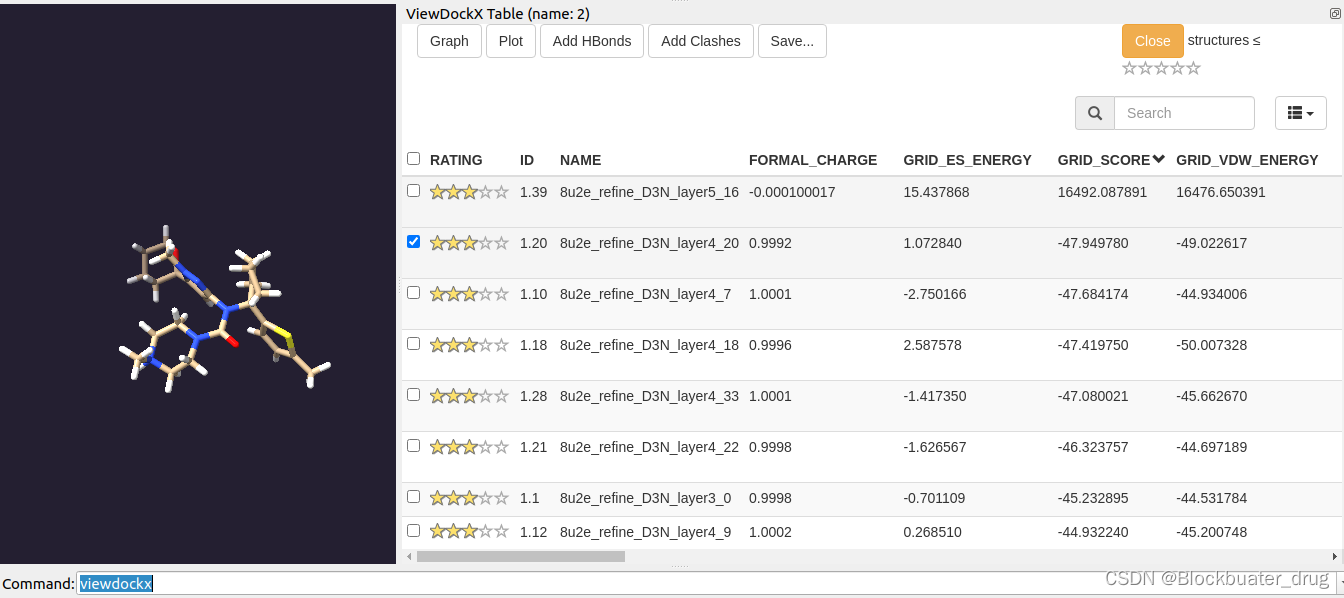
欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 文章目录 前言一、 软件及操作环境二、研究目的三、结构文件准备四、 DOCK/RDKit中 de novo design4.1 de novo design - refine_D3N4.2 对输出重新评分 总结参考资料 前言 本文是UCSF DOCK的使用案例分享,我们将使用DOCK 6.11
在Jupyter-lab中使用RDKit画分子2D图
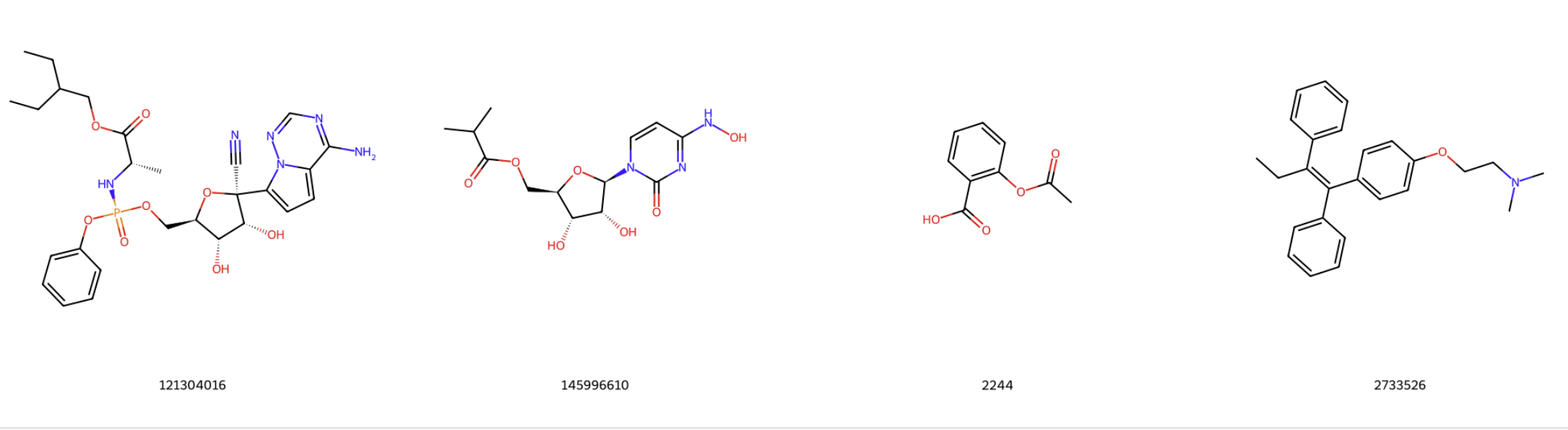
在Jupyter-lab中使用RDKit画分子2D图 在做完分子对接后,想看看筛选后的分子的结构。因此想利用Jupyter-lab来画分子的2D图。 1. 安装Jupyter-lab与RDKit 系统:Win11已安装conda RDKit 是一个功能强大、灵活易用的化学信息学工具包,广泛应用于药物发现、化学生物学、材料科学等领域。RDKit 提供了丰富的化学信息可视化功能,用户可以通过R
RDKit:基于分子文件输出分子结构
#!/usr/bin/python3from rdkit import Chemfrom rdkit.Chem import Drawspl = Chem.SDMolSupplier('molecules.sdf') #读入分子库mols = []for mol in spl:mols.append(mol)# 获取前十个分子mols = mols[:10]#输出分子结构img =
关于Conda中Rdkit报错ImportError:无libboost_python动态库问题解决方法--安装至指定目录
关于Conda中Rdkit报错ImportError:无libboost_python动态库问题解决方法--安装至指定目录 问题解析解决方法1. boost包下载2. boost包编译安装 参考博客 问题解析 libboost_python36.so.1.73.0报错信息解析: 缺少的包名为boost该库支持语言为python3.6boost包版本为1.73.0 报错原因有