本文主要是介绍Paper - CombFold: Predicting structures of large protein assemblies 环境配置,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
欢迎关注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://spike.blog.csdn.net/article/details/136153329
CombFold:
- GitHub:https://github.com/dina-lab3D/CombFold
- Paper:predicting structures of large protein assemblies using a combinatorial assembly algorithm and AlphaFold2
参考: Paper - CombFold: predicting structures of large protein assemblies 论文简读
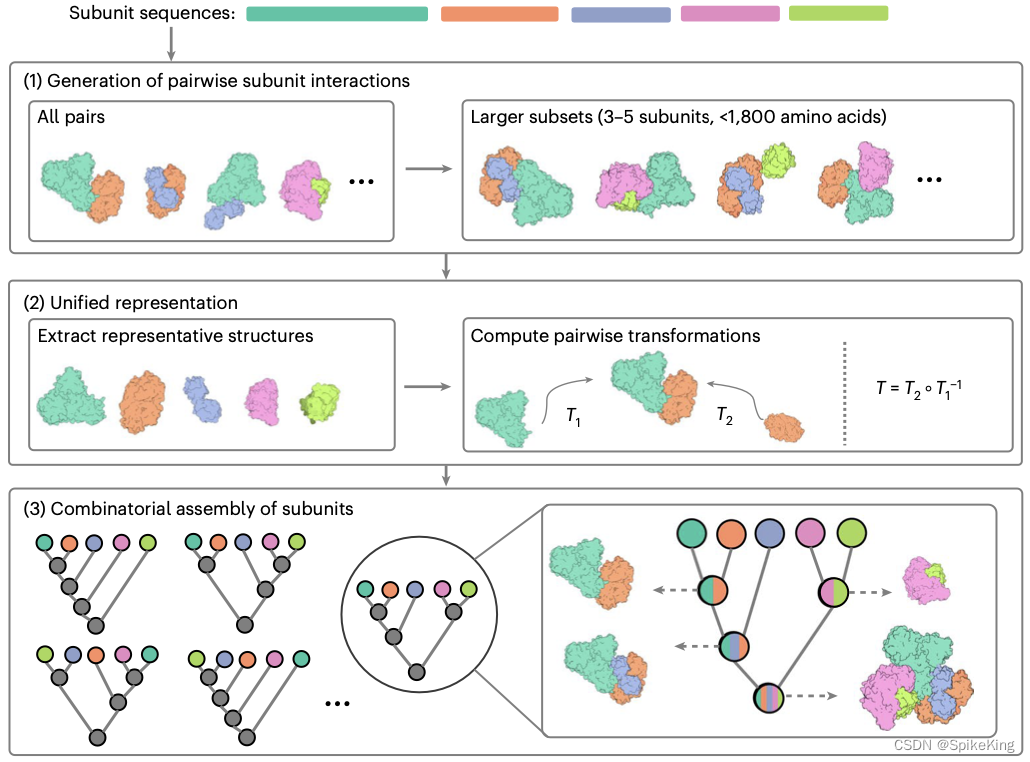
CombFold 流程,从复合物中链的序列开始,预测大型蛋白质复合物的结构 (最多包含18000个氨基酸和32个亚基)。该流程使用 AlphaFold-Multimer (AFM) 来预测 可能的亚基复合物 的结构,这些亚复合物是目标复合物的亚基的组合。CombFold 组合装配算法,将这些结构组装成一个单一的大型复合物。
该流程有 4 个阶段:
- 定义复合物中的亚基;
- 使用 AFM 预测所有亚基配对的结构
- [可选] 使用 AFM 预测更大的亚基组的结构
- 在所有生成的结构上运行组合装配算法 (关键)

论文图示:
1. 配置 Conda 环境
CombFold 工程配置:
apt-get update
apt-get install libboost-all-dev
# mac: brew install boost conda create -y -n combfold python=3.9pip install numpy
pip install scipy
pip install biopython
pip install py3Dmolgit clone https://github.com/dina-lab3D/CombFold.git
cd CombFold/CombinatorialAssembler
make
pip -q install,-q表示–quiet
Conda 环境参考:PyTorch - 高效快速配置 Conda + PyTorch 环境 (解决 segment fault )
Boost 编译错误:
libs_gamb/CIF.cc:7:10: fatal error: boost/algorithm/string.hpp: No such file or directory#include <boost/algorithm/string.hpp>
解决方案,参考 StackOverflow - Boost “no such file or directory”:
sudo apt-get update
sudo apt-get install libboost-all-dev
需要预先,构建 docker,端口 8901 用于 JupyterLab:
nvidia-docker run -it --name combfold -p 8901:8901 -v /pfs_beijing:/pfs_beijing -v /nfs_beijing:/nfs_beijing -v /nfs_beijing_ai:/nfs_beijing_ai glm:nvidia-pytorch-1.11.0-cu116-py3
1.2 配置 Jupyter 环境
参考:Server - 服务器配置 Conda 和 Jupiter Lab 的环境
密码 123:
argon2:$argon2id$v=19$m=10240,t=10,p=8$6YqE0s5gu4ZD6H3EEam0GQ$9IDRv7tq/8NC/ivzocosClkxrB2ia8jk5mZzuO07MRU
配置:
c.ServerApp.ip='*'
c.ServerApp.password=u'argon2:$argon2id$v=19...'
c.ServerApp.open_browser=False
c.ServerApp.port=8901
c.ServerApp.notebook_dir="/"
启动服务:
nohup jupyter lab --port=8901 --no-browser --allow-root > nohup.jupyter-docker-8901.out &
添加环境:
python -m ipykernel install --user --name combfold --display-name "combfold"
参考:
- CSDN - 解决JupyterNotebook启动时的警告:has moved from NotebookApp to ServerApp
- StackOverflow - ModuleNotFoundError: No module named ‘notebook.auth’
这篇关于Paper - CombFold: Predicting structures of large protein assemblies 环境配置的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!





