本文主要是介绍Nature子刊 | snATAC-seq 揭示斑马鱼胚胎早期发育过程中的单核染色质景观,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
2023年7月19日,一篇题为“Single-nucleus chromatin landscapes during zebrafish early embryogenesis”的研究论文在scientific data(IF=9.8)上发表,该研究使用华大智造单细胞测序平台进行snATAC-seq,建立了斑马鱼胚胎发育第一天的染色质可及性数据集。此外,通过整合snATAC-seq和scRNA-seq数据,描述了整个发育时间点染色质可及性和基因表达的动态变化,从而验证了染色质景观数据的准确性。

01 研究背景
脊椎动物胚胎发生是一个值得注意的过程,在这个过程中,不同谱系的细胞类型在短时间内出现。理解这一过程的一个巨大挑战是缺乏动态的染色质可及性信息来关联细胞命运决定层次中的顺式调控元件(CREs)和基因表达。
02 技术路线
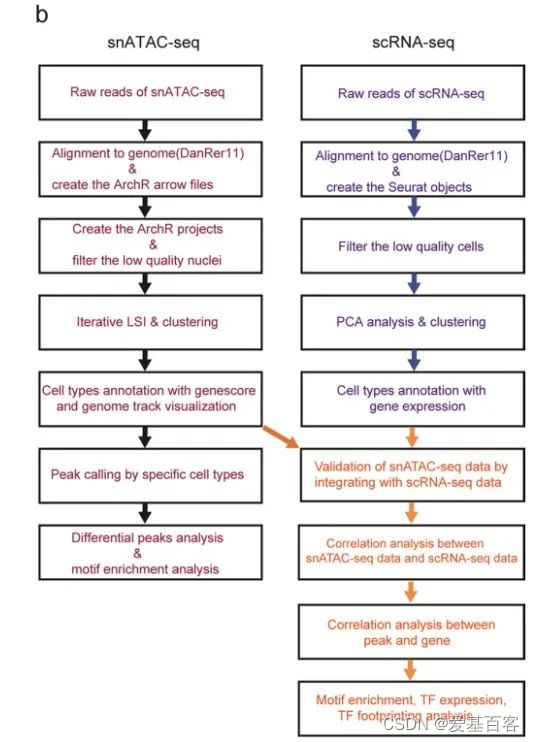
03 研究结果
取AB野生型杂交斑马鱼胚胎,在囊胚期(3.3 hpf)、原肠期(5.25 hpf、6 hpf、10 hpf)和体节期(12 hpf、18 hpf、24 hpf)进行snATAC-seq分析(图1)。为了数据的可靠性,作者在每个发育阶段设置了2-4个生物重复,用于snATAC-seq文库的构建。
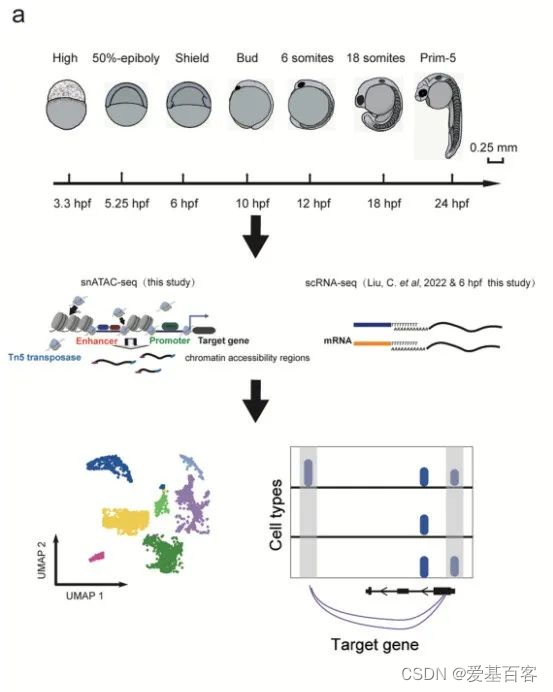
图1. 斑马鱼胚胎不同发育阶段的实验概要(上)以及snATAC-seq和scRNA-seq流程图(下)
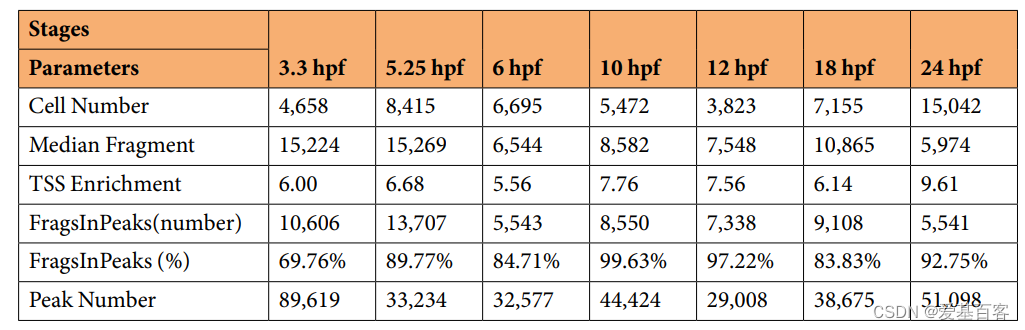
为了进行质量控制(QC),进行分析以过滤低质量数据,计算转录起始位点(TSS)富集分数和每个发育阶段每个细胞核的独特片段(图2a)。在这里,由于散点图分布在每个发育时间点不同,用于去除低片段和TSS富集分数的细胞核的过滤标准在每个各自的发育时间点不同。在QC和进一步的双重去除,各时间点共获得62699和51620个高质量的核(图2b,表1),其中TSS富集分数主要分布在5-10之间,唯一片段数量主要分布在5000 - 20000个之间(图2c,表1)。同时,作者在所有发育阶段分别鉴定出29008-89619个非冗余峰(表1),并在注释的TSS周围观察到明显的的片段峰(图2d)。此外,作者还整体生成了每个时间点的每个重复的核数量(图2e)、TSS富集分数(图2f,上)和唯一片段数(图2f,下),以便进一步分析。此外,来自20个剖面比较的Pearson相关系数的热图聚类显示,同一发育时间点的重复之间具有高度相关性,这表明生物重复和技术重复的重复性很高(图2g)。
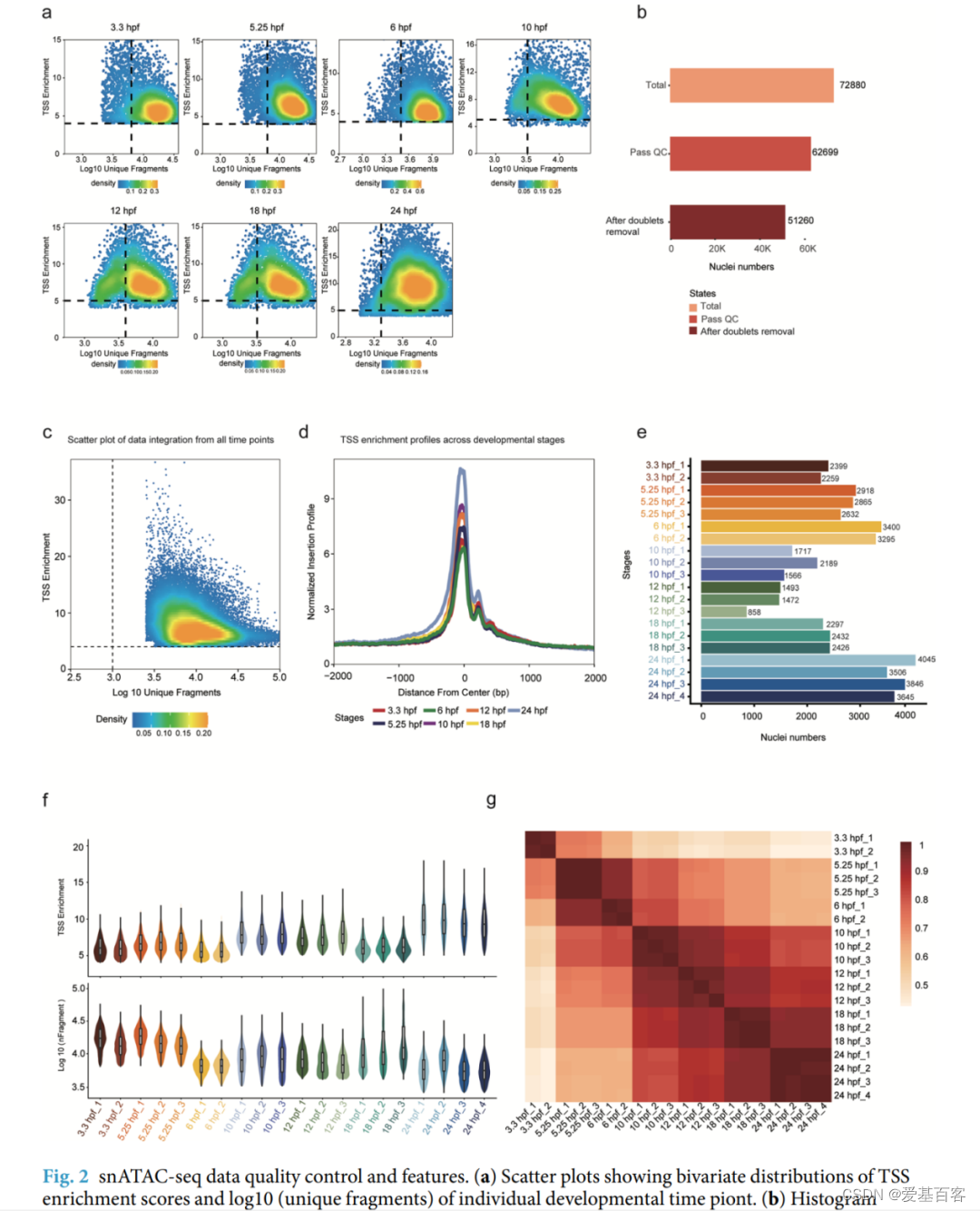
图2. snATAC-seq数据质量控制和特点。
表1. 在发育中的斑马鱼胚胎中建立的snATAC-seq图谱的QC参数概述。
随后,利用ArchR (Methods)对所有发育时间点的细胞核进行整合和聚类,以进行细胞类型特异性调控注释。作者通过将基因启动子和基因体中的片段相加来注释细胞簇来计算基因活性分数,其中两个簇由于细胞太少和缺乏簇特异性基因而被排除在外。最终,23个细胞簇被确定为候选细胞类型,包括被膜系统(包膜层(EVL)、周皮/表皮、被膜)、神经系统(神经干细胞、前脑、未成熟眼、神经龙骨)、肌肉系统、消化系统等(图3a,左)。每个簇的标记基因见补充表2。通过比较不同时间点细胞类型的比例(图3a,中),作者发现细胞类型在发育过程中逐渐增加。然后,基于对snATAC-seq数据的UMAP可视化并按发育阶段着色(图3a,右),作者观察到囊胚、原肠和体节的细胞离散地聚集在一起,并彼此分离。
为了验证染色质景观数据的准确性,作者检查了snATAC-seq数据和相应的scRNA-seq数据的一致性(图1a,b)。作者对先前发表的scRNA-seq数据集(包括3.3 hpf、5.25 hpf、10 hpf、12 hpf、18 hpf和24 hpf)结合本研究中获得的6 hpf胚胎的未发表的scRNA-seq数据进行了无监督聚类分析,并确定了30个簇(簇28因细胞太少和缺乏簇特异性基因而被排除)(图3b)。每个簇的差异表达基因可在补充表3中找到。此外,通过整合这两个数据集,作者观察到snATAC-seq数据的基因活性评分与scRNA-seq数据的基因表达值之间具有良好的相关性(图3c),并且这两个数据集之间的隶属度具有高度一致性(图3d)。
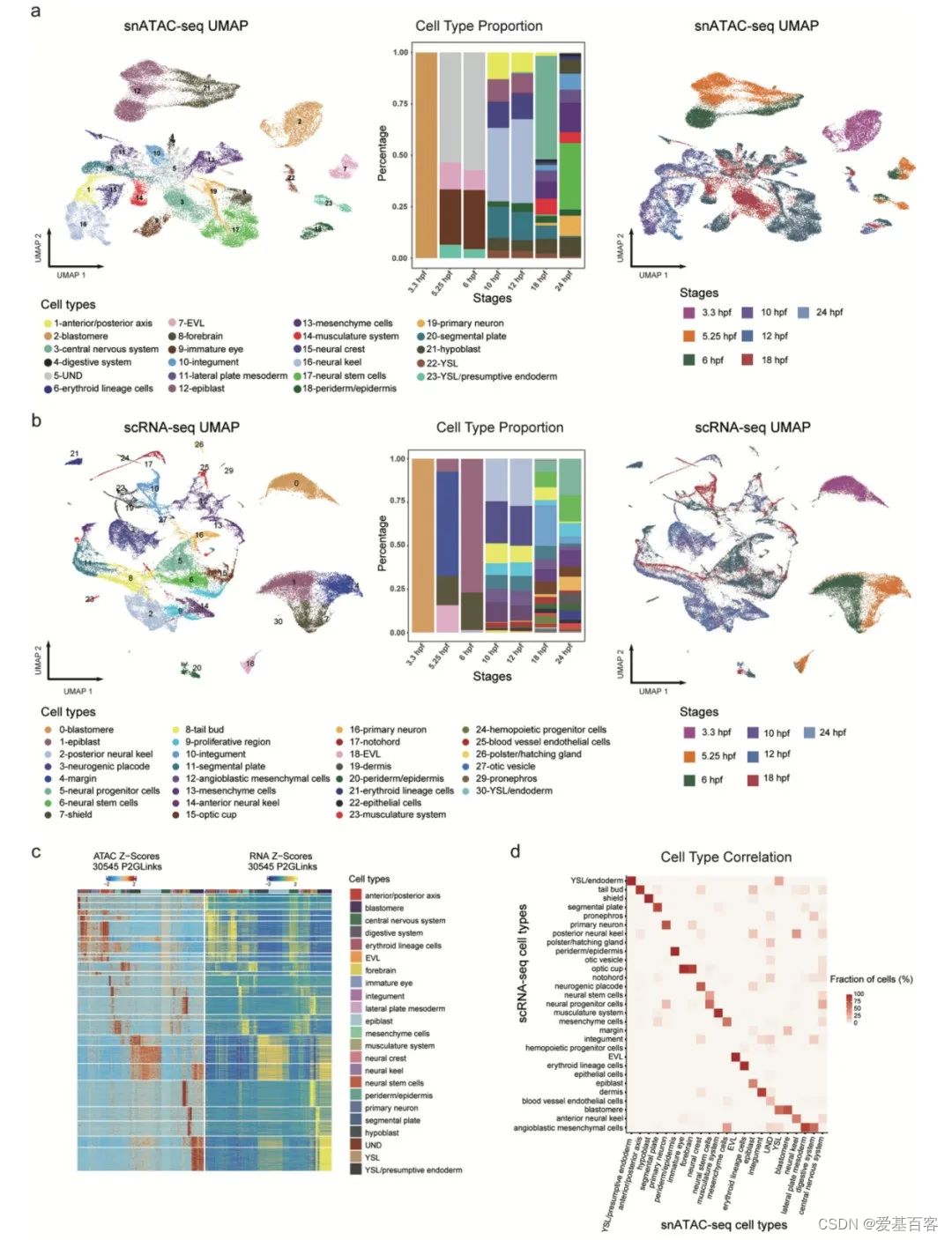
图3. 斑马鱼胚胎发育过程中染色质可及性和基因表达模式的聚类和注释
同时,作者发现染色质景观中启动子周围具有可及元件的基因(图4a),在scRNA-seq数据的相应细胞类型中也具有高表达水平(图4b),如nanog参与囊胚多能性地维持,sox32特别在卵黄合胞层(YSL)/内胚层中表达并参与内胚层形成,elavl3在初级神经元中特异表达,参与神经发生。
为了进一步剖析snATAC-seq数据中不同细胞类型的调节因子,作者将由snATAC-seq数据生成的转录因子(TF) motif富集(图4c)和TF基因活性评分(图4d)与从scRNA-seq数据获得的TF表达(图4e)进行了比较。这些分析表明,它们在相应的细胞类型中具有良好的一致性,如体细胞中的myf5,EVL中的grh1,红系细胞中的tal1,尾芽和前后轴中的cdx4。
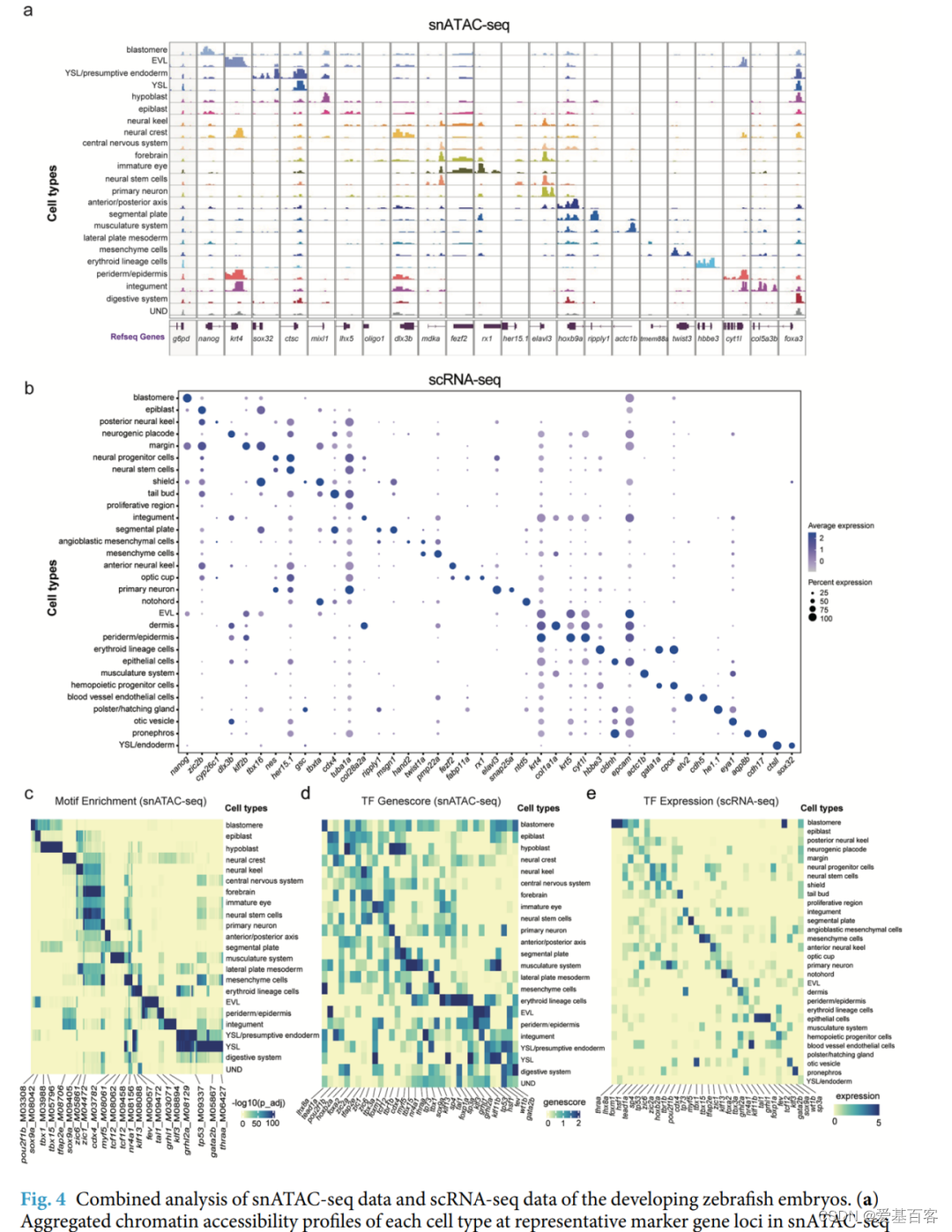
图4. 斑马鱼胚胎发育过程中snATAC-seq数据和scRNA-seq数据的联合分析
为了验证鉴定的motif,作者进行了TF足迹分析,证实了TF与DNA的结合。作者发现这些基序在相应的细胞类型中表现出活跃的TF结合(图5)。总之,作者生成了斑马鱼早期胚胎发生的染色质可及性图谱,证明了与相应的已发表的scRNA-seq数据的高度一致性。

图5. 足迹分析确定了snATAC-seq数据中具有代表性的细胞类型特定的TF活动。(上)代表细胞类型特定的TF结合motif序列标志,(下)代表特定细胞类型的TF足迹轮廓。与图4相关
04 小 结
在本研究中,作者在斑马鱼胚胎发育第一天的七个不同时间点采用snATAC-seq检测转座酶可及染色质,生成了51620个细胞核的可及性图谱,并建立了斑马鱼早期胚胎发生过程中顺式调控元件的DNA可及性(CRE)动态的资源。此外,通过在同一发育时间点整合snATAC-seq数据和scRNA-seq数据,作者系统地表征了不同细胞类型在所研究的发育时间点的染色质可及性和基因表达的动态,这两个数据集之间表现出良好的一致性。综上所述,作者的数据集为深入探索斑马鱼胚胎发育过程中的表观遗传调控机制提供了宝贵的资源。
原文链接:https://doi.org/10.1038/s41597-023-02373-y
05 公司简介
爱基百客提供领先的表观组学技术服务,目前已经积累了丰富的ATAC-seq和单细胞项目经验,助力客户发表Protein Cell、Genome Biology、Plant Communications和Nature Communicaitons等高水平期刊。

ATAC-seq和单细胞项目文章(部分)
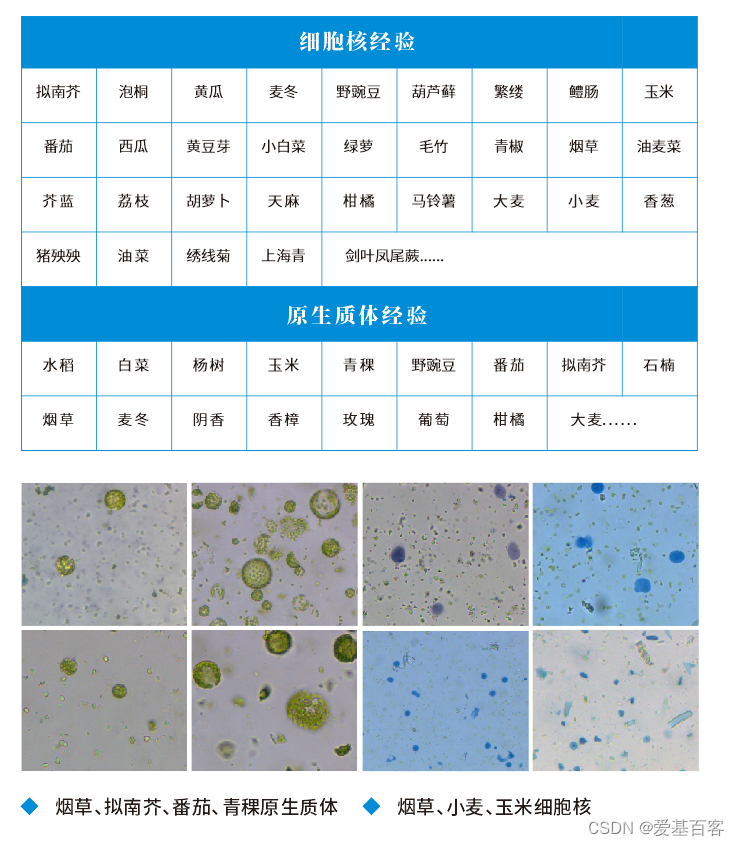
单细胞测序的难点之一在于前期样本解离,人和动物方面我们积累了包括PBMC、脂肪组织、心脏、肝脏和脑等50余种组织经验;植物已测试40+种不同植物材料的单细胞核提取以及原生质体制备,还完成水稻、拟南芥、小麦和烟草等模式以及非模式植物的单细胞科研服务。
-
单细胞悬液制备经验
人和动物:
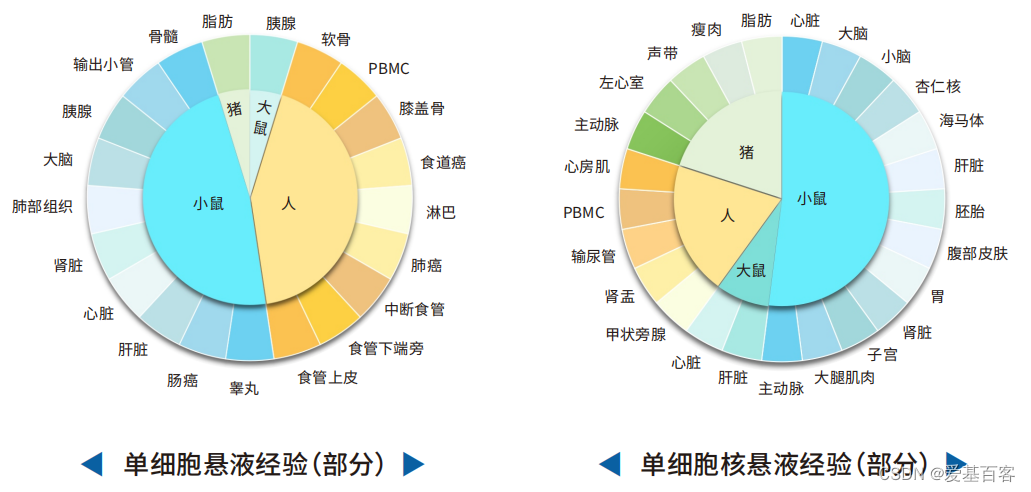
植物:
爱基百客近期喜获“华大智造DNBelab C系列高通量单细胞测序ATAC文库制备”认证服务商,作为国内首批认证的服务商,我们将继续提供优质的科研服务,以满足客户的需求。

这篇关于Nature子刊 | snATAC-seq 揭示斑马鱼胚胎早期发育过程中的单核染色质景观的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!






