本文主要是介绍pymol可视化蛋白质-蛋白质相互作用,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
一、准备工作
在淘宝花6元购买pymol并安装(可以在windows上使用!);在在线对接软件上提交两种蛋白pdb文件得到对接结果的pdb文件,下载后打开pymol,导入对接结果的pdb文件:
file open
选择文件路径
一般线上对接软件导出的对接结果pdb文件会有两个对象object:一个配体ligand和受体receptor,为了后面方便命令输入,可以修改名称简化表示
set_name old_name, new_name注意!pymol中特有的对象是“图层”(之后会用到),它初始在object上表示为(sele),但是其实其真实名称就是sele,因此修改名称时正确输入为
set_name sele, new_name由于表示化学键时,蛋白质结构最好用球棍模型(stick)来表示,给出一种高级的,只在相互作用附近展示球棍模型,大部分保留卡通模型的命令
show sticks, byres rec_name within 5 of lig_nameshow sticks, byres lig_name within 5 of rec_name其次可以微调透明度和背景
#修改透明度,透明度约接近于1,越不透明
set cartoon_transparency, 0.60#修改背景颜色
bg_color, white
注:很有启发的一篇博客
Pymol(1.8.6)作图技巧之cartoon和surface镶嵌模型_pymol怎么更改卡通形状-CSDN博客
二、接触位点可视化

1、首先将键展示出来
A find
polar contacts
to any atoms
出现新的包含键的文件
2、成键位点的标注
(1)放大:
1)点击含键文件的A zoom即可放大到分析级大小,直接用鼠标放大到一定程度会消失;
2)同时,点击右下角的F全屏,会有全新的视野用以标注接触的residue
(2)标注:先标rec或lig的一个,隐藏另一个;反之同理;最后会出现一个图层文件,包含了键和相应的成键位点
为了更加清晰,可以隐藏无用的接触面附近的stick
rec/lig对象 H sticks
然后,突触图层文件的成键情况
成键图层 S sticks
(3)显示位点编号
图层文件 Label residues(1 letter)
有些位点编号与结构相互遮挡,需要调整,这里不需要输入命令行,直接鼠标操作
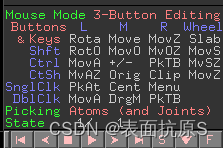
点击“3-Button Viewing”变为“3-Button Editing”,似乎点击后下次开启就自动变为Editing了,然后在长按ctrl键的前提下,鼠标长按移动可改变位点编号的位置
!如果鼠标点击了蛋白质的结构后拖动,结构会随鼠标移动发生形变!
三、生成导出图片
点击Draw/Ray即可生成图片!
参考资料:
PyMOL入门基础--分子三维结构可视化教程(上篇) - 知乎
使用 PyMOL 可视化蛋白质-蛋白质对接 - 知乎
new bing chat
这篇关于pymol可视化蛋白质-蛋白质相互作用的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!