本文主要是介绍ICLR2023 | 基于几何结构预训练的蛋白质表示学习,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
今天为大家介绍的是来自唐建老师团队的一篇关于蛋白质预训练的论文。学习有效的蛋白质表示对于预测蛋白质功能或结构等生物学任务至关重要。现有的方法通常在大量未标记的氨基酸序列上预训练蛋白质语言模型,然后在下游任务中使用一些标记数据微调模型。尽管基于序列的方法有效,但是基于蛋白质结构的预训练还未被探索,因为已知的蛋白质结构数量较少。为此,作者提出了根据蛋白质的三维结构预训练蛋白质表示的方法。在功能预测等任务上的实验结果表明,在使用了更少的预训练数据的前提下,文中提出的预训练方法表现突出。

蛋白质是细胞功能的执行单位,与众多领域息息相关。它们由一系列氨基酸线性链组成,可折叠成特定的构象。由于低成本测序技术的出现,近年来发现了大量的蛋白质序列。然而,对新蛋白质序列的功能注释仍然昂贵而耗时,与其他机器学习领域相比,蛋白质结构的数据集规模要小几个数量级。为了填补这一差距,最近的研究利用大量未标记的蛋白质序列数据来学习蛋白质的有效表示,然而这些方法没有利用可用的蛋白质结构信息,而蛋白质结构已被证明是蛋白质功能的决定因素。
由于最近深度学习为基础的高精度蛋白质结构预测方法的进展,现在可以高效地预测大量蛋白质序列的结构,预测结果也具有合理的可信度。基于此,作者开发了一个基于蛋白质结构的预训练蛋白质编码器,能够推广到各种属性预测任务。作者提出了一种简单而有效的基于结构的编码器,称为GearNet,该网络通过添加不同类型的残基边信息来编码蛋白质的空间信息,然后在蛋白质残基图上执行消息传递。受Evoformer中三角形关注设计的启发,作者提出了一种稀疏的边缘消息传递机制来增强蛋白质结构编码器,这是首次尝试在GNN中引入边消息传递来进行蛋白质结构编码。
基于结构的蛋白质编码器
作者将蛋白质的结构表示为残基级别的关系图G=(V, E, R),其中V和E分别表示节点和边的集合,而R是边的类型集合。图中有三种不同类型的有向边:顺序边、半径边和K最近邻边。其中,顺序边将根据两个端节点之间的相对顺序距离{−2, −1, 0, 1, 2}进一步分为5种不同类型的边,作者仅在顺序距离为2以内的节点之间添加顺序边。这些边的类型反映了不同的几何属性,这些属性共同构成了对蛋白质的全面特征表示。
构建完图网络之后,作者采用卷积神经网络GNN对蛋白质结构进行特征提取。除了传统的以氨基酸为节点的图网络以外(GearNet),作者还提出了一种基于边的图网络。在分子表示学习的工作中,许多几何编码器都受益于明确地建模边之间的相互作用。提出了一种带有边消息传递层的GearNet变体,称为GearNet-Edge。其主要目的是建模残基与其他顺序或空间相邻残基的不同相互作用之间的依赖关系。在此图网络中,每个节点对应于原始图中的一条边。倘若在原始图中两条边连接在同一个残基上,那么这两条边代表的节点将在GearNet-Edge网络中相连,相连边的类型由对应的两条边在原始图上的夹角决定。
基于对比学习的预训练方法
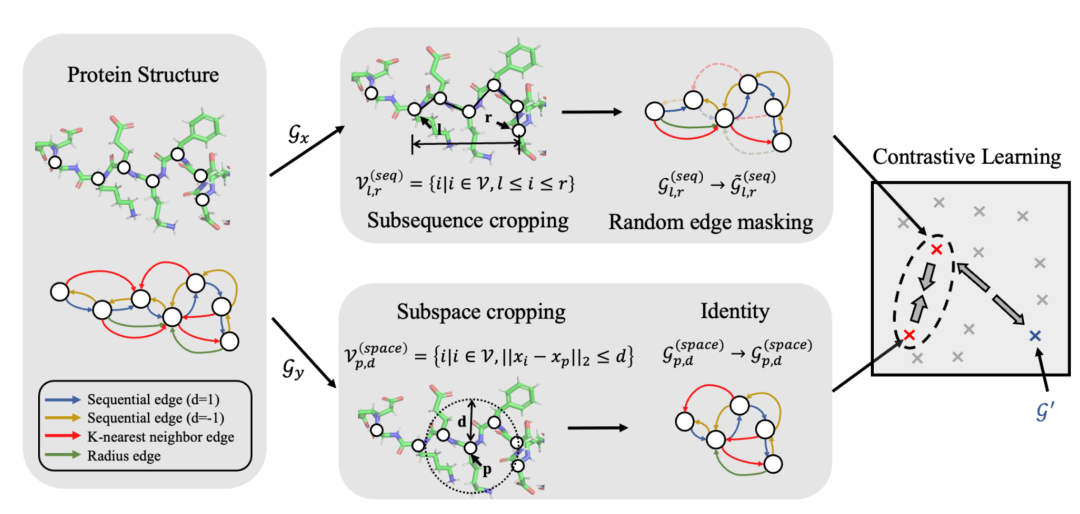
对于给定蛋白质图G,作者考虑构建两种不同的采样方案来构造视图。第一种是基于子序列裁剪的视图。此视图随机采样一个左侧残基l和一个右侧残基r,并选取所有在l和r之间的残基。这个采样方案旨在捕捉不同蛋白质中重复出现并指示它们功能的连续蛋白质子序列。然而,简单地采样蛋白质子序列不能充分利用蛋白质数据中的3D结构信息。因此,作者进一步引入了一个基于子空间裁剪方案的视图,用以发现与空间相关的结构基元,它随机采样一个残基p作为中心,并选择所有在预定义半径d内的残基。随后采用对比学习,对两种视图的特征进行比较学习,流程如上图所示。
实验结果
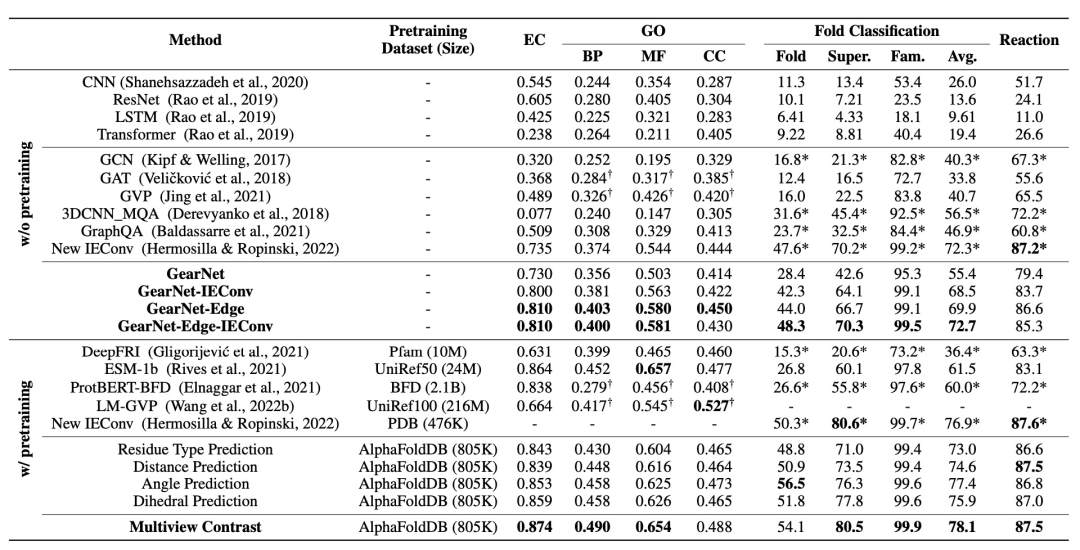
模型首先使用AlphaFold蛋白质结构数据库进行预训练,随后在四项任务上进行微调。四项任务分别为:Enzyme Commission (EC) number prediction,Gene Ontology (GO) term prediction,Fold classification,Reaction classification。实验结果如上表格所示,基于实验结果,GearNet相比于以前的工作有三大优势:
-
在没有预训练的情况下,GearNet的性能可以在大部分数据集上优于其他模型。
-
基于结构的蛋白质编码器受益于使用未标记结构进行预训练。
将上表中第三个块和最后两个块的结果进行比较,可以发现所有的预训练方法都比从头开始训练的模型表现出了显著的提升。
在这些方法中,多视图对比法在8个数据集中有7个表现最好,并在EC、GO-BP、GO-MF、Fold和Reaction任务中取得了最先进的结果。
这证明了作者预训练策略的有效性。
-
基于结构的编码器的表现优于基于序列的编码器,后者使用了更多的数据进行预训练。
最后三个实验块展示了基于序列和基于结构的预训练模型的比较。
需要注意的是,作者的模型是在不到100万个结构的数据集上进行预训练的,而所有基于序列的预训练基线都是在百万或十亿级别的序列数据库上进行预训练的。
结论
文章提出了一种基于蛋白质残基图进行关系消息传递的简单而有效的结构化编码器,引入了一种新的边消息传递机制来明确边之间的相互作用。通过多个基准任务的全面实验验证,文章中的模型在从头开始训练时效果优于以前的编码器,并且在使用更少的数据进行预训练的情况下实现堪比甚至超越最先进的基线方法的效果。作者认为此项研究是采用自监督学习方法进行蛋白质结构理解的重要一步。
参考资料
Zhang, Zuobai, et al. "Protein representation learning by geometric structure pretraining." arXiv preprint arXiv:2203.06125 (2022).
代码
https://github.com/DeepGraphLearning/GearNet
这篇关于ICLR2023 | 基于几何结构预训练的蛋白质表示学习的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







