本文主要是介绍MA-SAM:模态不可知的三维医学图像分割SAM自适应,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
论文:MA-SAM: Modality-agnostic SAM Adaptation for 3D Medical Image Segmentation | Papers With Code
代码:GitHub - cchen-cc/MA-SAM: PyTorch implementation for MA-SAM
机构:a)高级医疗计算和分析中心,麻省总医院和哈佛医学院,波士顿;b)香港中文大学计算机科学与工程系,c)伯利恒利哈伊大学计算机科学与工程系,d)哈佛大学约翰保尔森工程与应用科学学院,e) 美国佐治亚大学计算机学院
摘要
SAM是一般图像分割的基础模型,在许多自然图像分割任务中表现出令人印象深刻的zero-shot性能。然而,当应用于医学图像时,SAM的性能显着下降,主要是由于自然和医学图像域之间的巨大差异。为了有效地使SAM适应医学图像,在微调期间纳入关键的三维信息(即体积或时间知识)非常重要。同时,我们的目标是充分利用SAM在其原始2D骨干中的预训练权重。在本文中,我们引入了一个模态无关的SAM自适应框架,称为MA-SAM,它适用于各种体积和视频医疗数据。我们的方法基于parameter-efficient 的微调策略,仅更新一小部分权值增量,同时保留SAM的大部分预训练权值。通过在图像编码器的Transformer中注入一系列3D适配器,我们的方法使预训练的2D骨干能够从输入数据中提取三维信息。通过使用10个公共数据集(包括CT、MRI和外科视频数据),对我们的方法在四种医学图像分割任务中的有效性进行了全面评估。值得注意的是,在不使用任何提示的情况下,我们的方法始终优于各种最先进的3D方法,在CT多器官分割、MRI前列腺分割和手术场景分割方面的Dice分别超过nnU-Net 0.9%、2.6%和9.9%。我们的模型也显示出很强的泛化,并且在使用提示时擅长挑战肿瘤分割。
背景
SAM 模型在医学分割上效果不佳
首先,SAM的训练数据集包含大量的图像集合。在医疗应用的背景下获取类似的大规模训练数据集是极具挑战性的。虽然SAM的训练数据只包含自然图像,但它并不局限于任何特定的医学成像模式。如果SAM微调被证明对一种类型的医学成像有效,那么同样的方法很有可能也适用于其他模式。
其次,经过微调后,SAM作为预训练的大型模型可能具有鲁棒泛化的潜力
第三,SAM的提示设计为半自动分割解决肿瘤分割等困难任务提供了方便的解决方案。在这些方面,SAM提供了一个通用的基础模型,具有适应不同医学成像模式的潜力,为全自动和半自动分割提供了良好的泛化能力。
挑战
例如,考虑一个包含多个器官的腹部CT容积,即使在每个切片上提供每个器官的基本点提示,也需要付出很大的努力。
此外,在分割对象形状和位置相对规则的情况下,自动分割方法已经获得了令人鼓舞的结果,从而消除了半自动分割中提示的需要。
在SAM适应自动医学图像分割的背景下,最近的一些研究采用了参数高效迁移学习(PETL)技术,如LoRA (Hu et al., 2021)或Adapters (Houlsby et al., 2019),在自动分割中表现出良好的性能(Zhang and Liu, 2023;Wang et al., 2023a)。然而,这些方法侧重于纯二维自适应,忽略了医学图像中固有的有价值的三维信息。这包括医学体积数据中至关重要的三维空间信息和医学视频数据中的时间信息。
解决
在本文中,我们提出了一种模态无关的医学图像分割SAM自适应方法,称为MASAM,该方法能够高效地捕获医学数据中的体积或时间信息。对于图像编码器的微调,我们利用了称为FacT的参数高效迁移学习PETL技术(Jie and Deng, 2023),该技术基于tensorization decomposition来提高微调效率。
这种微调方法在很大程度上保留了预训练的权值,只更新轻量级的权值增量,保证了对象分割所需的一般知识的保留,减少了需要调整的参数数量。
为了弥合二维自然图像与体积或视频医疗数据之间的差距,我们进一步在图像编码器的每个变压器块中加入一组3D适配器,以提取有价值的三维信息。为了适应轻量级掩码解码器,我们采用了全微调,并使用简单而有效的渐进式上采样机制来修改其原始架构,以恢复预测分辨率。我们证明了我们的SAM适应框架在处理各种分割任务的多种医学成像模式上的有效性。通过对多种SOTA方法的比较,我们的自动分割方法表现出了优越的性能和显著的泛化能力。
贡献
1)提出了一种参数有效的微调方法,使SAM适应体积和视频医疗数据。我们的方法通过轻量级的3D适配器有效地将医学图像中的基本三维信息整合到2D网络骨干中。
2)证明我们的SAM适应可以应用于各种医学成像模式,包括CT、MRI和手术视频数据,用于解剖、手术场景和肿瘤分割。在不使用任何提示的情况下,我们的自动分割始终大大优于竞争对手的SOTA方法。
3)我们验证了在医学图像上进行微调后,得到的模型具有出色的泛化能力,甚至比SOTA域泛化方法表现出更好的性能。
4)我们表明,通过进一步利用提示,我们的方法在挑战肿瘤分割任务中取得了令人印象深刻的结果,在Dice得分上超过nnU-Net 38.7%。
相关方法
Vision foundation models 视觉基础模型
图文匹配:
CLIP
ALIGN
通用:
Florence(Yuan et al., 2021)
SAM
SegGPT(Wang et al., 2023c)
SEEM(Zou et al., 2023)
Parameter-efficient transfer learning 参数高效迁移学习
随着大型模型表现出的优异性能,对大型基础模型进行预训练,然后对特定下游任务进行微调的模式越来越受欢迎。这些方法只对少量(额外)参数进行微调以获得强大的性能。
PETL技术最初是在自然语言处理中提出的,可以分为三大类(Lialin et al., 2023)
加性方法:
Adapter
目的是通过引入额外的参数或层来增强现有的预训练模型,然后只对这些新引入的组件进行微调
选择性方法:
选择性方法侧重于更新模型中几个选定的有影响的层或内部结构(Gheini et al., 2021;Zaken et al., 2022)。
基于重新参数化的方法:
LoRA (Hu et al., 2021)
FacT (Jie and Deng, 2023)
利用低秩表示来最小化可训练参数的数量,在各种PETL任务中展示了鲁棒性和SOTA性能。
🤔现在最主流,效果最好的是基于重新参数化的方法
Adapting SAM in medical imaging SAM在医疗影像中的应用
SAM在医学分割中效果很差,有些任务dice差距甚至高达70%
在使用提示所观察到的改进的推动下,大多数作品在微调期间利用了SAM的提示设计(Cheng等人,2023;邓等,20023a;Dai等人,2023;Yue等人,2023)。
SAM- med2d (Cheng et al., 2023)采用了更全面的提示,包括点、边界框和Mask,为2D医学图像量身定制SAM,并进行了综合评估。
MSA (Wu et al., 2023a)采用点提示和适配器技术将医学领域知识集成到SAM模型中。
然而,为3D医疗数据的每个2D切片创建提示是一项劳动密集型工作。在自动医学图像分割中自适应SAM (Hu et al., 2023;Paranjape等人,2023)、SAMed (Zhang and Liu, 2023)和Wang等人(Wang et al., 2023a)采用LoRA进行微调,性能优于多种二维医学图像分割方法。然而,这些方法没有考虑到关键的三维体积或时间信息,而这些信息对于增强医学图像分割性能是有价值的。
感觉这篇工作的主要工作还是整了个可以3D的SAM,还是蛮有参考性的?
方法
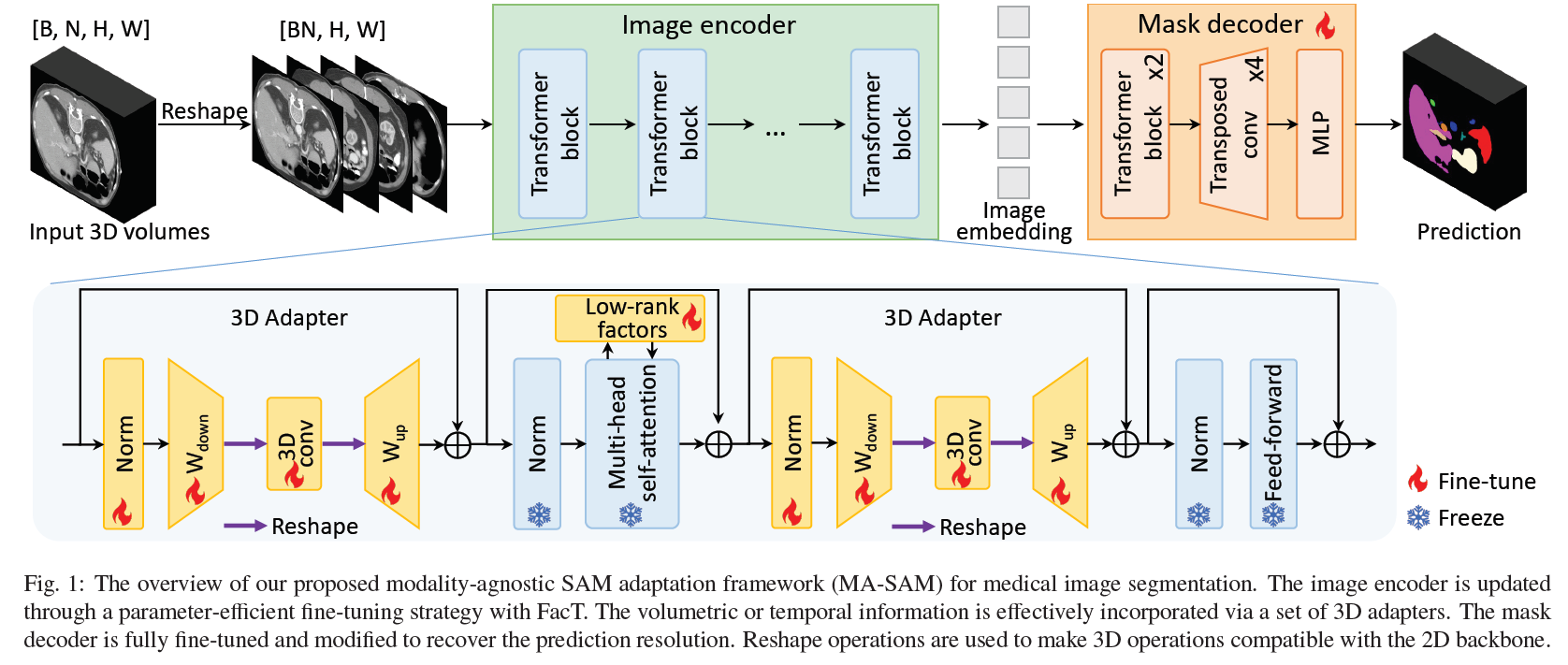
SAM概括
SAM是一种提示式分割架构,由三个主要组件组成,即图像编码器、提示编码器和掩码解码器。
图像编码器: ViT
提示编码器:提示编码器接受各种类型的提示,包括点、框或文本,并将这些输入编码为提示嵌入,以促进分割任务。
掩码解码器:设计轻巧,计算图像嵌入和提示之间的交叉关注,并利用转置卷积层和多层感知生成分割掩码。
当应用于医学图像时,由于医学图像与自然图像具有不同的纹理和物体,因此模型的性能大大降低。这突出了针对特定任务对SAM进行微调以应对此类挑战的必要性。
图像编码器的参数有效微调
为了有效地提取图像特征,SAM的图像编码器包含了相当一部分网络参数。微调所有这些权重是计算密集型的。先前的研究表明,PETL技术可以实现类似于完全微调的自适应性能,但更新的网络参数要少得多(Hu et al., 2021;Pan et al., 2022)。在这方面,我们采用了FacT (Jie and Deng, 2023),这是一种SOTA PETL技术,与其他PETL方法相比,它可以在引入较少数量的可训练参数的情况下获得相当或更好的性能。
震惊,我搜了一下FacT好新好新,是2023年aaai的
基于基于Transformer的模型在秩上趋于冗余的常见观察,FacT假设用于微调的密集权重增量矩阵ΔW可以通过一组具有跨层权重共享的低秩因子来近似。根据FacT中的张量分解,我们将每层的权值增量ΔW分解为三个因子U∈Rd×r, V∈Rd×r, Σ∈Rr×r,其中d表示ViT中的特征维数,r表示这些因子的秩,r<<d.值得注意的是,U和V这两个因子在所有层中都是共享的,而因子Σ对于每一层都是唯一的。然后可以使用以下公式计算权重增量:
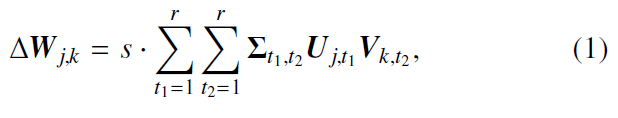
其中s为调节因子学习率的超参数。在我们的实验中,我们将s固定为1,并使用优化器调整整体学习率,以达到类似的缩放效果。将FacT权重增量应用于每个转换块中的查询和值转换,而从SAM初始化的所有其他权重保持冻结,因为根据经验,在将FacT应用于其他层时没有观察到明显的改进。随着FacT权重的增加,查询和值转换变成:

其中,wq /v为调优后的查询或值转换;
W0表示SAM预训练的权值。
没咋接触过微调,到时候琢磨琢磨好了
包含体积和时间信息
结合医学成像数据中固有的体积或时间知识,对于医学应用中SAM的成功迁移学习至关重要。为了解决这一关键挑战,我们建议在SAM架构内将一系列3D适配器集成到2DTransformer块中。这些适配器用于提取医学图像分析所需的基本体积或时间洞察力。通过整合这些适配器,我们弥合了医学成像数据固有复杂性与SAM预训练的2D主干之间的差距,使其能够有效地处理多维医学数据。

具体而言,如图1所示,每个3D适配器由归一化层、线性下投影层、三维卷积层(后为激活层)和线性上投影层组成。体积或时间信息的核心提取主要存在于三维卷积层中。下投影层的目的是将原来的d维特征降维为更紧凑的c维表示,从而控制新引入参数的数量。相反,上投影层恢复特征维度。M表示特征映射,则三维适配器可表示为↓ 其中Norm为层归一化,σ为激活函数,Wdown∈Rd×c和Wup∈Rc×d分别为线性向下和向上投影层,Conv3D为三维卷积层,核大小为3×1×1,专门提取三维信息。 所以2D的时候就是把3D adapter 拆掉吗?后面实验部分也没提到2D任务就是了
![]()
为了使3D适配器与2D SAM主干兼容,对于网络输入,我们提取了一组相邻的切片。其中,B表示批大小,N表示相邻切片数,H × W表示切片尺寸。
![]()
在输入传递到SAM骨干前,通过将相邻的切片合并到批处理维度中,对x∈RB×N×H×W进行重塑操作,将x∈R B×N×H×W转化为x∈R BN×H×W。
然后,对于特征映射,在输入到3D适配器的3D卷积层之前,将它们从[BN, H/16,W/16, c]重塑为[B, c, N, H/16,W/16]。
其中H/16和W/16表示特征映射的空间维度,由于变压器中的patch嵌入过程,特征映射被下采样了16倍。经过三维卷积运算后,特征图的形状又变回原来的形状。通过这种方法,可以有效地提取二维骨干网络中的体积或时间信息。对于每个变压器块,我们在注意层之前和之后都加入了两个3D适配器,因为这样的设计可以获得经验上更好的性能。
Adapting mask decoder 自适应掩码解码器
原始SAM中的掩码解码器仅包含两个变压器层、两个转置卷积层和一个多层感知层。考虑到其轻量级的结构,在全掩码解码器上进行全微调是可行的,可以有效地适应医学图像。在SAM图像编码器内Transformer主干的补丁嵌入过程中,每个16×16补丁作为特征向量嵌入,导致输入有16×16次下采样。SAM掩码解码器利用两个连续的转置卷积层对特征映射进行4倍的上采样,但SAM生成的最终预测的分辨率仍然比原始形状低4倍。然而,由于医学图像中的许多解剖结构或病变非常小,因此通常需要实现更高的分辨率,以确保在医学成像背景下提高识别能力(Ronneberger et al., 2015)。
为了解决这个问题,我们探索了两种方法来定制掩码解码器,以增强医学图像分割的适用性。
对于第一种方法,称为“渐进式上采样”,我们通过积分两个额外的转置卷积操作,对SAM解码器进行适度调整。每一层对特征映射进行2倍的上采样,四个转置卷积层逐渐将特征映射恢复到原始输入分辨率。
第二种方法被称为“多尺度融合”,需要创建一个类似于“u形”网络的设计(Ronneberger等人,2015)。这涉及到使用跳过连接将图像编码器的多尺度特征图与掩码解码器的相应阶段连接起来,这是一个类似于U-Net的概念。为了实现这一点,我们将图像编码器统一划分为四个阶段,通过一系列上采样和卷积操作,在每个阶段的特征映射与解码器的特征映射之间建立联系。
在我们的实验中,我们观察到渐进式上采样机制与多层特征聚合相比产生了更好的结果,显示了渐进式上采样方法的有效性和简单性。
(有点惊讶,多加专职卷积居然比类U-Net结构效果更好欸)
实验
数据集
任务1
The Beyond The Cranial Vault (BTCV)挑战数据集(Landman et al., 2015)包含30个CT体数据,其中包含13个腹部器官的手动注释。每次CT扫描包含85 ~ 198个切片,切片厚度从2.5 ~ 5.0 mm不等。所有扫描轴向尺寸为512 × 512,但面内分辨率范围为0.54 × 0.54 mm2至0.98 × 0.98 mm2。我们使用与(Tang et al., 2022a)相同的数据分割,其中包含24个用于训练的案例和6个用于测试的案例。
任务2
我们对6个MRI数据源(Liu et al., 2020)进行前列腺分割,即从nic - isbi13 (Bloch等人,2015)、I2CVB (Lema - ıtre等人,2015)和PROMISE12 (Litjens等人,2014)数据集中收集的站点A到F。每个站点的病例数分别为30、30、19、13、12、12,随机分为80%和20%进行培训和测试。这些来自不同部位的MRI扫描是通过不同的成像方案获得的,并且呈现异构的数据分布,因此通常用于以前的领域泛化研究(Liu et al., 2022)。
任务3
2018 MICCAI机器人场景分割挑战(EndoVis18)数据集(Allan et al., 2020)包括每个序列包含149、249或250帧,分辨率为1280 × 1024。该数据集包含手术场景,有12类针对各种解剖结构和机器人仪器进行了注释。数据集正式分为15个序列用于训练和4个序列用于测试。
任务4
2018年MICCAI医学分割十项全能挑战(MSD-Pancreas)数据集(Antonelli et al., 2022)中的胰腺肿瘤分割任务包含281个CT扫描胰腺和肿瘤的注释。每次扫描包括37至751片,轴向尺寸为512×512。我们遵循(Gong et al., 2023)在我们的实验中只使用肿瘤标签,并使用与他们工作中相同的数据分割。
此外,我们使用多模态腹部多器官分割挑战(AMOS 22)数据集(Ji et al., 2022)来评估模型泛化。该数据集包含来自不同患者的腹部CT和MRI数据。每次扫描都标注了15个器官,但我们关注的是与BTCV数据集重叠的12个器官。在AMOS 22的训练集和验证集中使用300个CT扫描和60个MRI扫描来进行我们的泛化评估。
数据预处理
我们分别在[- 200,250]Hounsfield Units (HU)和[- 50,200]HU范围内截断BTCV和msd -胰腺数据集的每次CT扫描强度值。每次MRI扫描的强度在第99个百分位数处截断。每个CT或MRI扫描归一化为零平均值和单位方差。对于手术视频数据,每帧归一化到[0,1]范围。我们将CT和MRI数据的轴向面以及每帧手术视频序列的所有图像调整为512 × 512。对于模型的评估,我们使用常用的度量,即Dice分数和Hausdorff Distance (HD)来分别评估像素分割精度和分割边界质量。我们还报告了EndoVis18数据集的平均相交-过并(mIoU)和msd -胰腺数据集的归一化表面距离(NSD),以与先前的研究保持一致。
实现细节
采用交叉熵损失和Dice损失相结合的混合分割损失对微调过程进行监督:Lseg = αLce + β lice。
除仅使用Dice loss的手术视频数据外,权重因子α和β分别设为0.2和0.8 (Zhang和Liu, 2023)。每五个连续切片作为网络输入。
对于数据增强,我们应用了一系列变换,包括随机旋转、翻转、擦除、剪切、缩放、平移、分隔、对比度调整、亮度修改和清晰度增强。我们的模型使用Adam优化器进行训练,批量大小为24。
如(Zhang and Liu, 2023)所述,我们采用预热训练策略,将学习率线性提高到特定值,然后在训练结束时指数降低学习率,以稳定训练。
我们采用ViT H作为图像编码器的主干,共进行了400次训练,保证了模型的有效收敛。
我们的框架是在PyTorch 2.0中使用8个NVIDIA A100 gpu实现的。
结果
已经很多项超过nn-Unet了,nb
对比模型:
基于Transformer的方法SwinUNETR (Tang et al., 2022b)是一种基于三维变压器的分层编码器模型,而nnFormer (Zhou et al., 2023a)是一种结合了局部和全局体积自关注机制的模型。我们还将我们的方法与最新的SAM自适应方法SAMed h (Zhang and Liu, 2023)和3DSAM-adapter (Gong et al., 2023)进行了比较。SAMed_h是一种用于器官分割的自动二维医学图像分割模型,而3DSAM-adapter是一种用于肿瘤分割的快速三维医学图像分割模型。
对于手术视频数据,我们将我们的方法与SOTA手术场景分割方法、NCT (Shvets等人,2018)、UNC (Ren等人,2020)和OTH (Chen等人,2018)(挑战中报道的前三种方法)、Noisy-LSTM (Wang等人,2021)(使用ConvLSTM学习时间线索)、STswinCL (Jin等人,2022)(基于变压器的捕获视频内和视频间关系的模型)和nu - net进行比较。对于所有比较实验,所有方法的数据集分割保持一致。
BTCV数据集
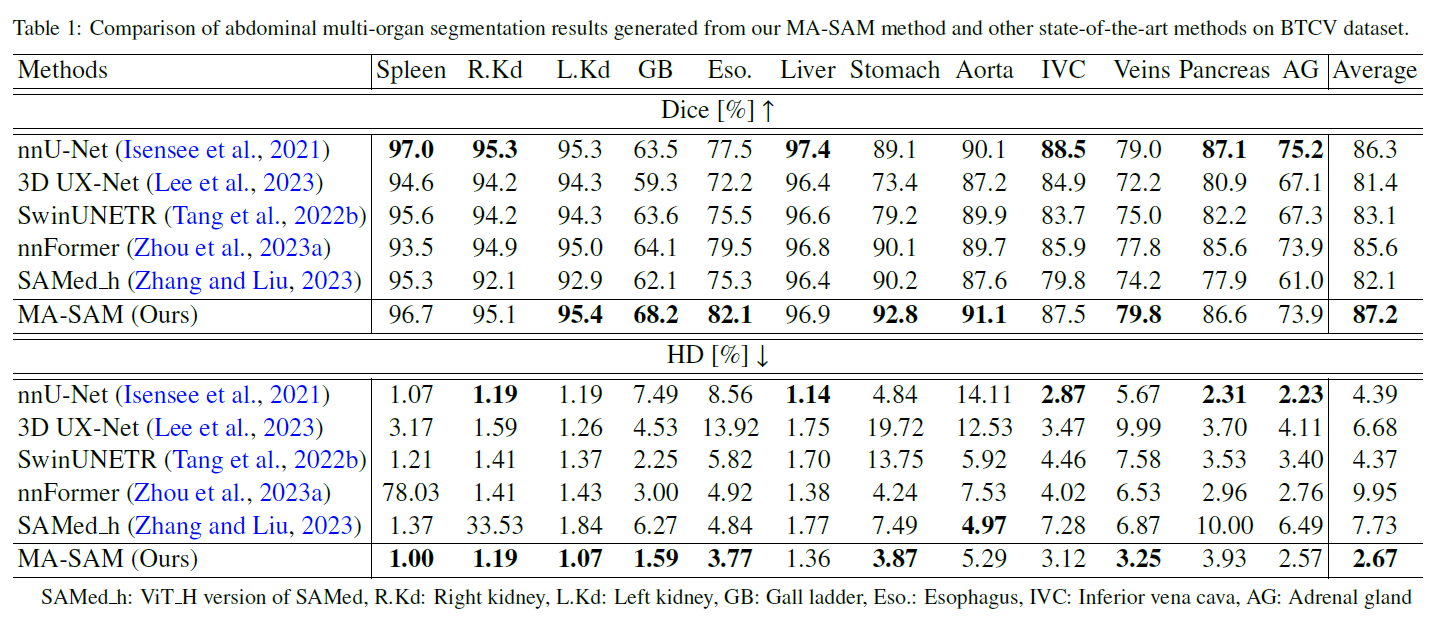
BTCV数据集上生成的分割结果的定性可视化
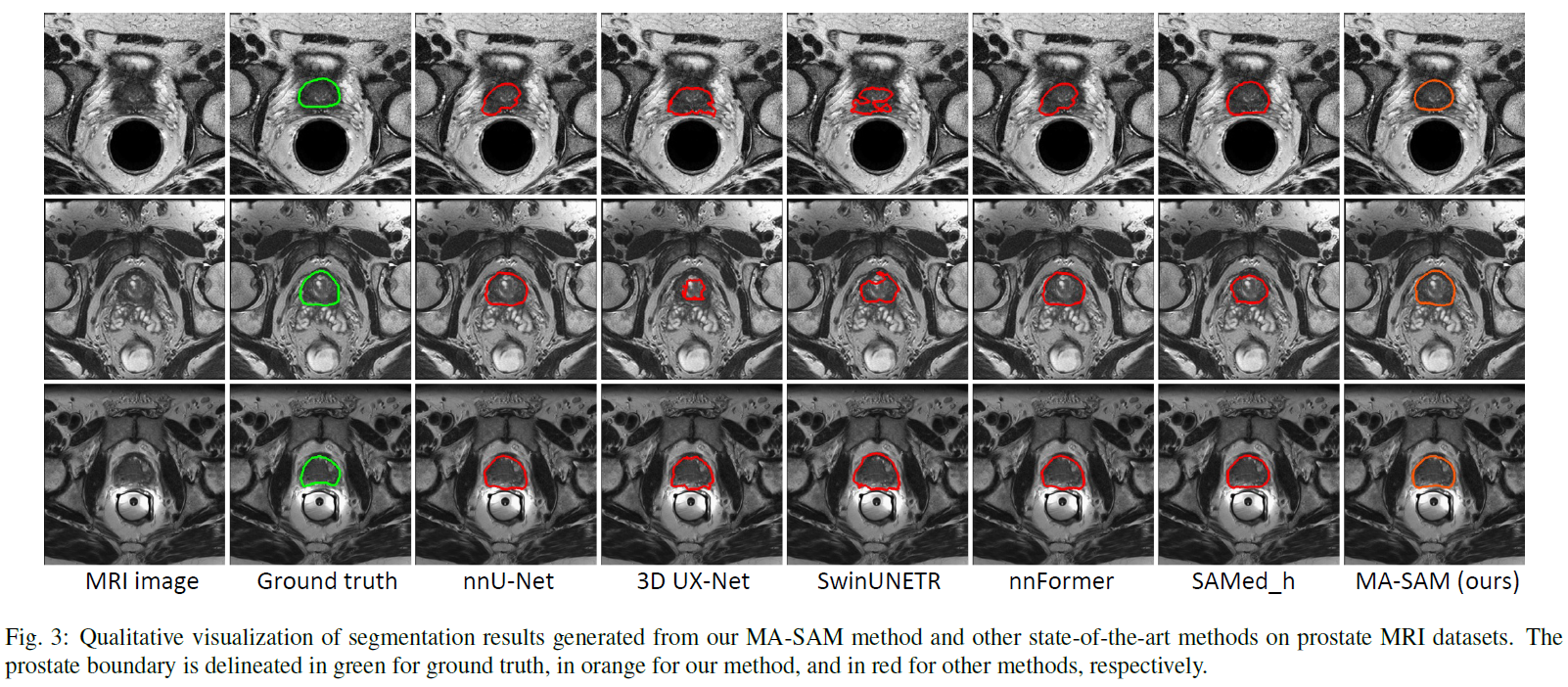
六个前列腺MRI数据集

前列腺MRI数据集上产生的分割结果的定性可视化。前列腺边界分别用绿色表示真实情况,用橙色表示我们的方法,用红色表示其他方法
Endovis18数据集上不同手术场景分割


胰腺肿瘤CT图像中不同分割方法分割
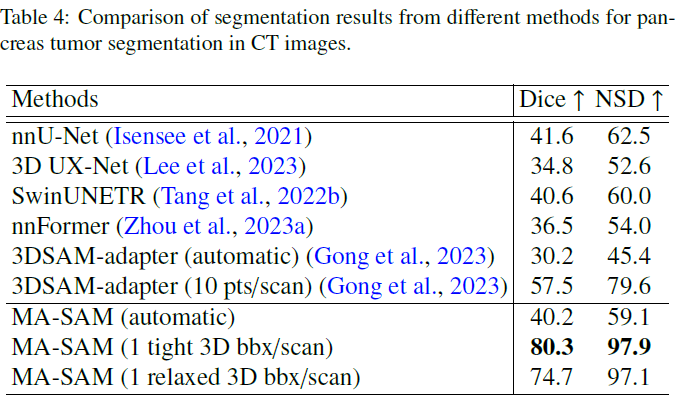
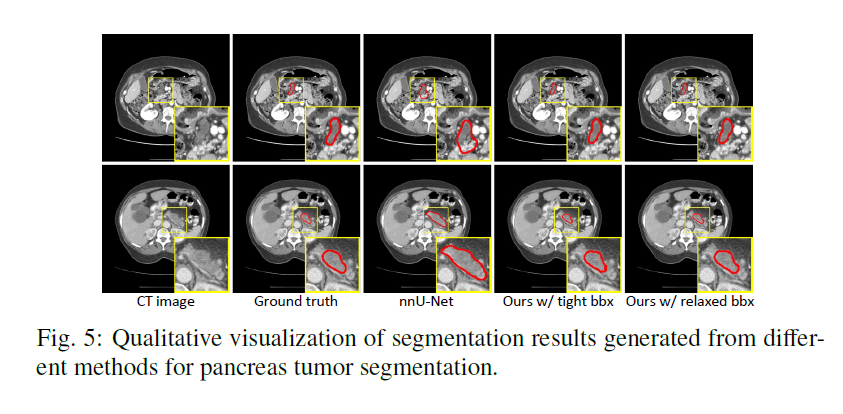
我们的方法也明显优于最近的整体3DSAM自适应方法3DSAM-adapter,在使用自动分割时,Dice改进了10%。这可以归因于我们的方法有效地将三维信息整合到模型微调中,以及大量利用SAM预训练的权重来保留一般的判别知识
由于胰腺肿瘤在CT扫描中轮廓不规则,边界不清,因此胰腺肿瘤分割提出了很大的挑战。从表4和图5可以看出,所有的自动分割模型都难以正确地描绘胰腺肿瘤区域,表现最好的模型仅获得41.6%的Dice分数。我们认为在这样一个要求苛刻的分词任务中,提示的使用变得很有价值。通过在模型中以每个体积一个紧密的3D边界框的形式添加提示,我们的方法显着将Dice分数从41.6%提高到80.35%,证明了利用提示进行肿瘤分割的有效性。然而,如果允许提供的边界框松紧度松弛5%,性能下降到74.7%,显示提示质量对分割性能的影响。
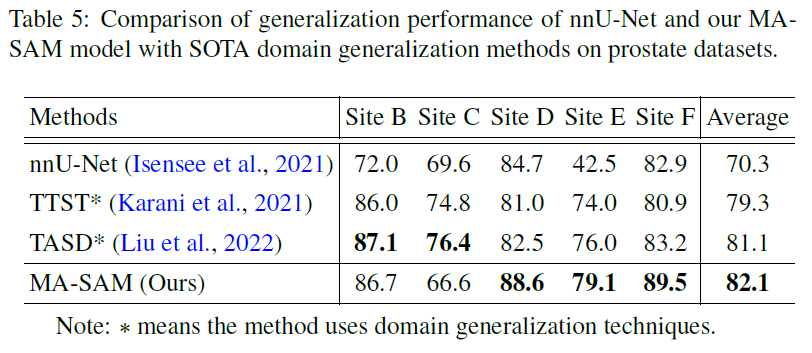
泛用性评估
AMOS2022

前列腺MRI分割 prostate MRI segmentation
消融实验
我们在BTCV数据集上进行了广泛的消融实验,以研究我们的SAM微调策略的几个关键方面:
1)我们方法中每个重要组件的有效性
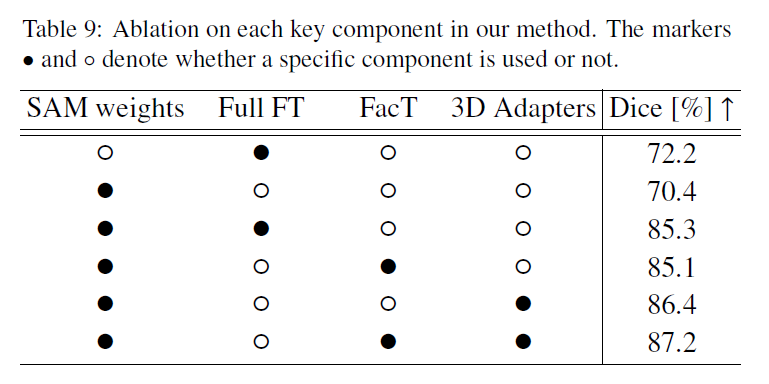
2)掩码解码器设计的影响

3) 网络主干网的影响
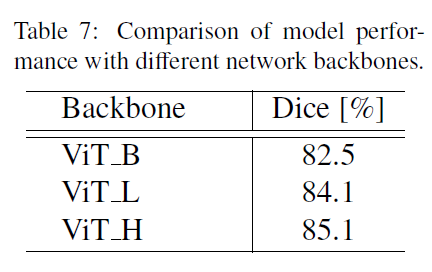
4)3D适配器的位置选择

5)参数有效微调的等级选择。
通过考虑集合{4,8,16,32,64}中的秩值,我们研究了模型的性能如何随着分解秩r的变化而变化。

讨论
基础模型,如分段任意模型(SAM),通过提供健壮的泛化和少量学习能力,已经彻底改变了智能模型的开发。SAM在自然图像任务中展示了令人印象深刻的zero-shot性能。然而,由于存在较大的域差异,直接将SAM应用于医学图像分割是无效的。为了解决这个问题,在这项工作中,我们提出了一种参数有效的微调方法,使SAM适应各种医学成像模式。我们的方法利用FacT有效地更新一小部分权重增量,并注入一组设计的3D适配器,以在微调期间提取医学图像的关键体积或时间知识。我们的方法的一般适用性和有效性已经验证了四种医学图像分割任务跨越三种成像模式。我们的模型还展示了出色的泛化能力,当使用提示时,在特别具有挑战性的肿瘤分割方面具有显着优势。
将SAM应用于医学图像的一个重要动机是它在庞大而多样的数据集上进行预训练,这在医学成像领域是很难实现的。这使得SAM的适应性普遍适用于各种医学成像方式。在医学应用中,最近有尝试预训练特定模式基础模型的努力。然而,这些模型通常局限于特定的医学成像模式,很难扩展到其他模式。例如,用胸部x线数据预先训练的模型在应用于MRI数据时可能会遇到困难。通过利用SAM的预训练权重,我们能够训练大规模分割网络,例如ViT H,用于医学图像分割,即使数据有限,例如仅使用5个成像扫描。我们的实验已经证明了增加模型大小的好处,提出了一个有趣的问题,即性能如何随着模型大小的进一步增加而发展。对于医学图像,我们可以使用更大的模型来提高准确性或增强泛化吗?探索这些可能性引起了极大的兴趣。
对于已经可以使用SOTA医学图像分割方法获得满意结果的任务,使用提示分割的意义不大。在处理具有挑战性的肿瘤分割任务时,提示被证明是特别有益和有价值的,正如我们的实验以及其他SAM微调工作所证明的那样。然而,制作有效的提示需要大量的努力。如表4所示,提示分割的性能随着提示质量的下降而下降。考虑到与手动提示符创建相关的挑战,在自动化此过程中有充分的未来探索空间。研究如何自动生成合适的提示符,以及如何在有噪声或不完整提示符的情况下训练出准确的分割模型,是非常有意义的。这将在手动提示创建具有挑战性的场景中增强提示分段的实用性。
这篇关于MA-SAM:模态不可知的三维医学图像分割SAM自适应的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







