本文主要是介绍qiita上传和分析16S rRNA数据,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
qiita(https://qiita.ucsd.edu/)是一个用于微生物组学研究的在线工具和资源库平台。

上传数据
注册并登陆
准备样品序列文件及meta信息和preparation文件
样品序列文件:后缀可以为fastq,fastq.gz,fasta,fna,不可以为fq等
meta文件:后缀可以为txt和tsv,第一列列名必须为sample_name,后面各列为样品meta信息,列名中不能出现空格(可以使用下划线_代替),样品名中不能出现“-”,列与列之间以table键分隔。
sample_name | Gender | Age | Caries |
S1 | Male | Prime | No |
S2 | Male | Youth | Yes |
S3 | Female | Prime | No |
preparation文件格式:后缀可以为txt和tsv,第一列列名必须为sample_name,后面各列为样品测序信息,run_prefix为序列文件的前缀,如S1样品对应的序列文件为S1.R1.fastq,S1.R2.fastq;platform为测序平台信息,target_gene为测序基因(如16S rRNA,WMS等);target_subfragment为测序区域;列与列之间以table键分隔。
sample_name | run_prefix | platform | target_gene | target_subfragment |
S1 | S1 | Illumina | 16S rRNA | V3-V4 |
S2 | S2 | Illumina | 16S rRNA | V3-V4 |
S3 | S3 | Illumina | 16S rRNA | V3-V4 |
创建项目并填写必要的相关信息
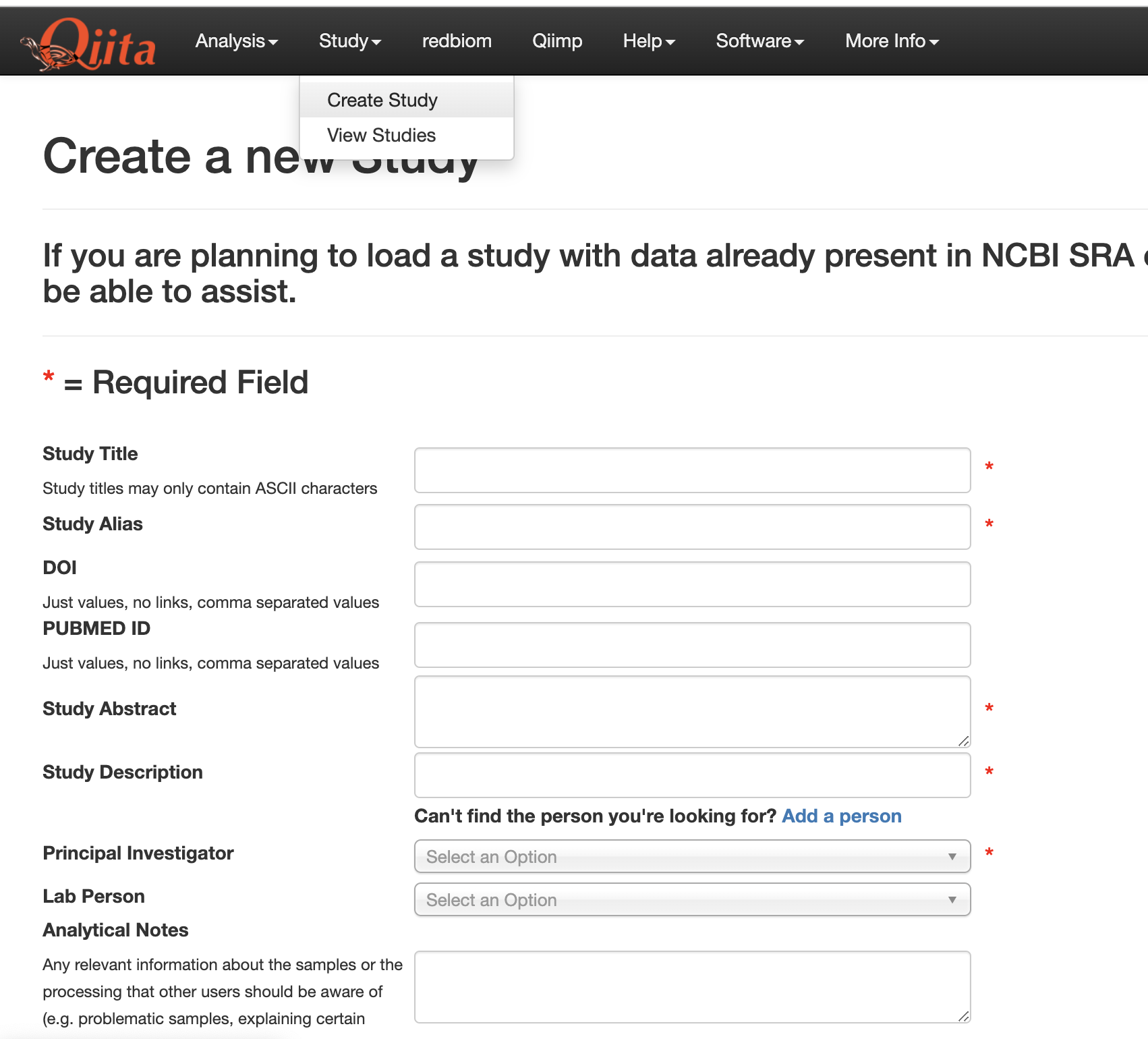
已经创建的项目可以通过点击Study-》View Studies查看
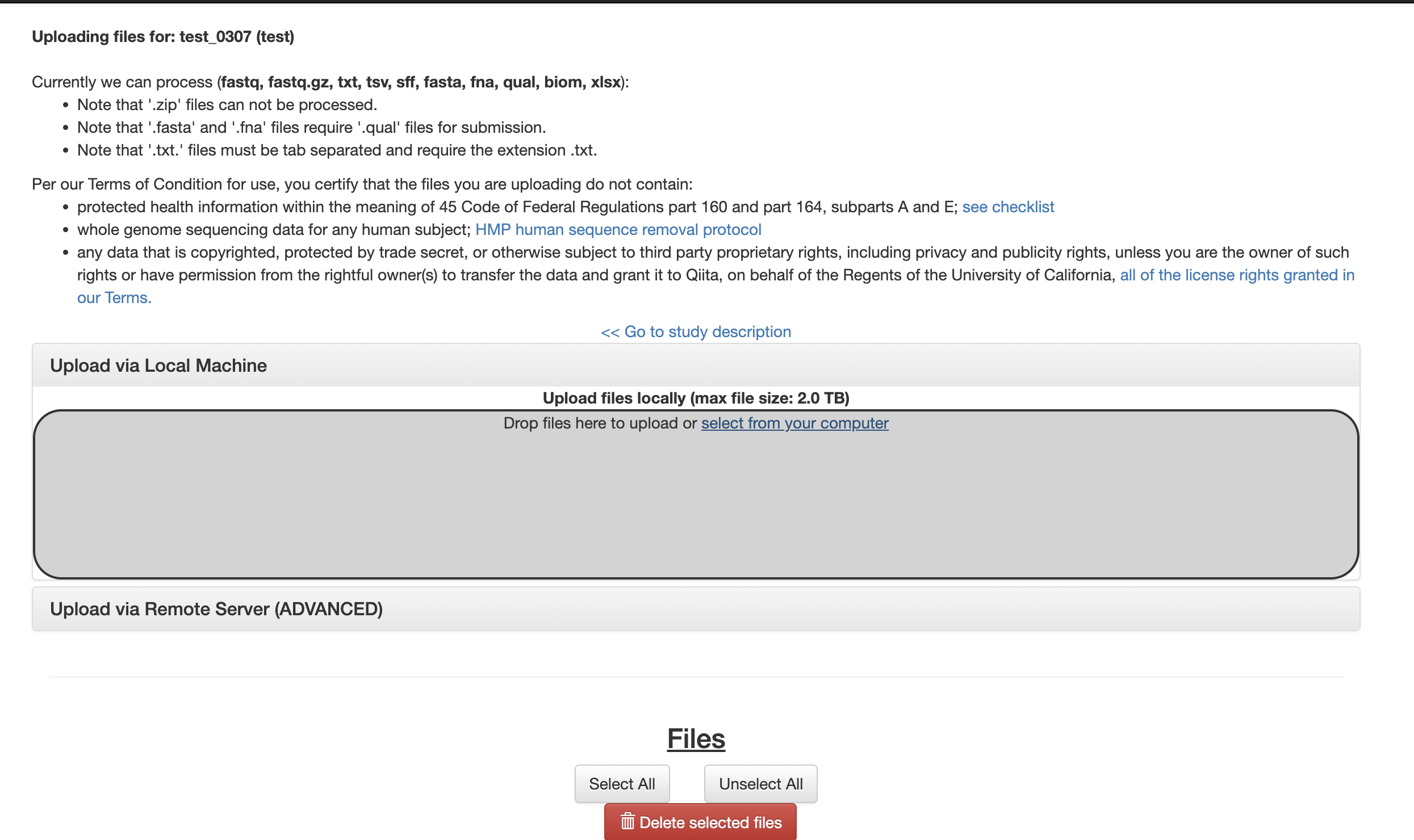
选择Upload Files,单击select from your computer上传序列文件,样品meta信息文件及preparation文件(如果要通过服务器上传文件,则需要服务器可以通过外网登陆访问,所以一般我们是用本地上传比较多)
点击<< Go to study description,返回研究介绍页面,单击左侧的Sample Information,点击Choose file选择上传的meta信息文件,data type选择16S,点击Create完成样品信息上传
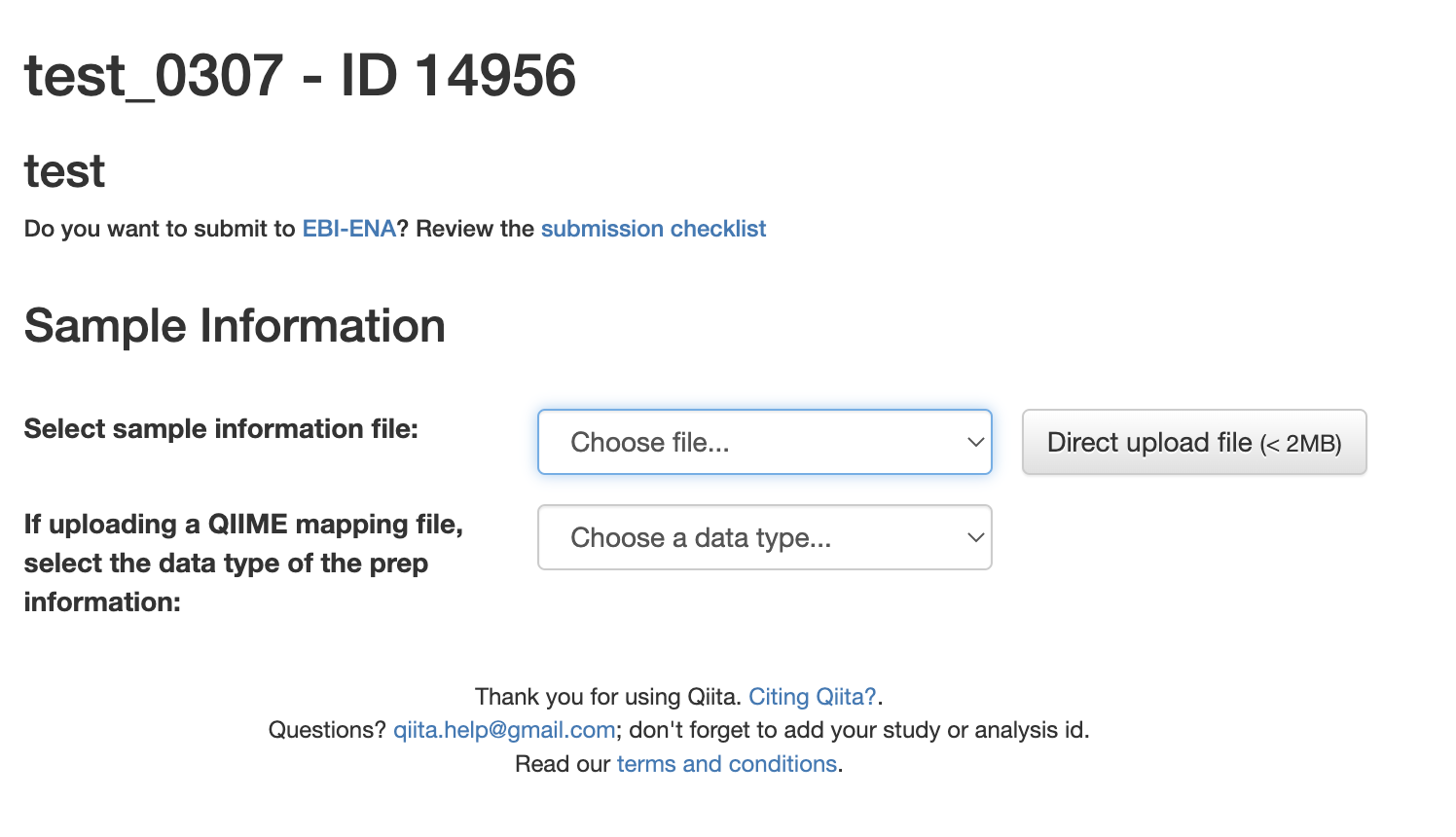
在新的页面中Updata sample information的Choose file内选择上传的preparation文件,单击右侧的Update完成样品上传
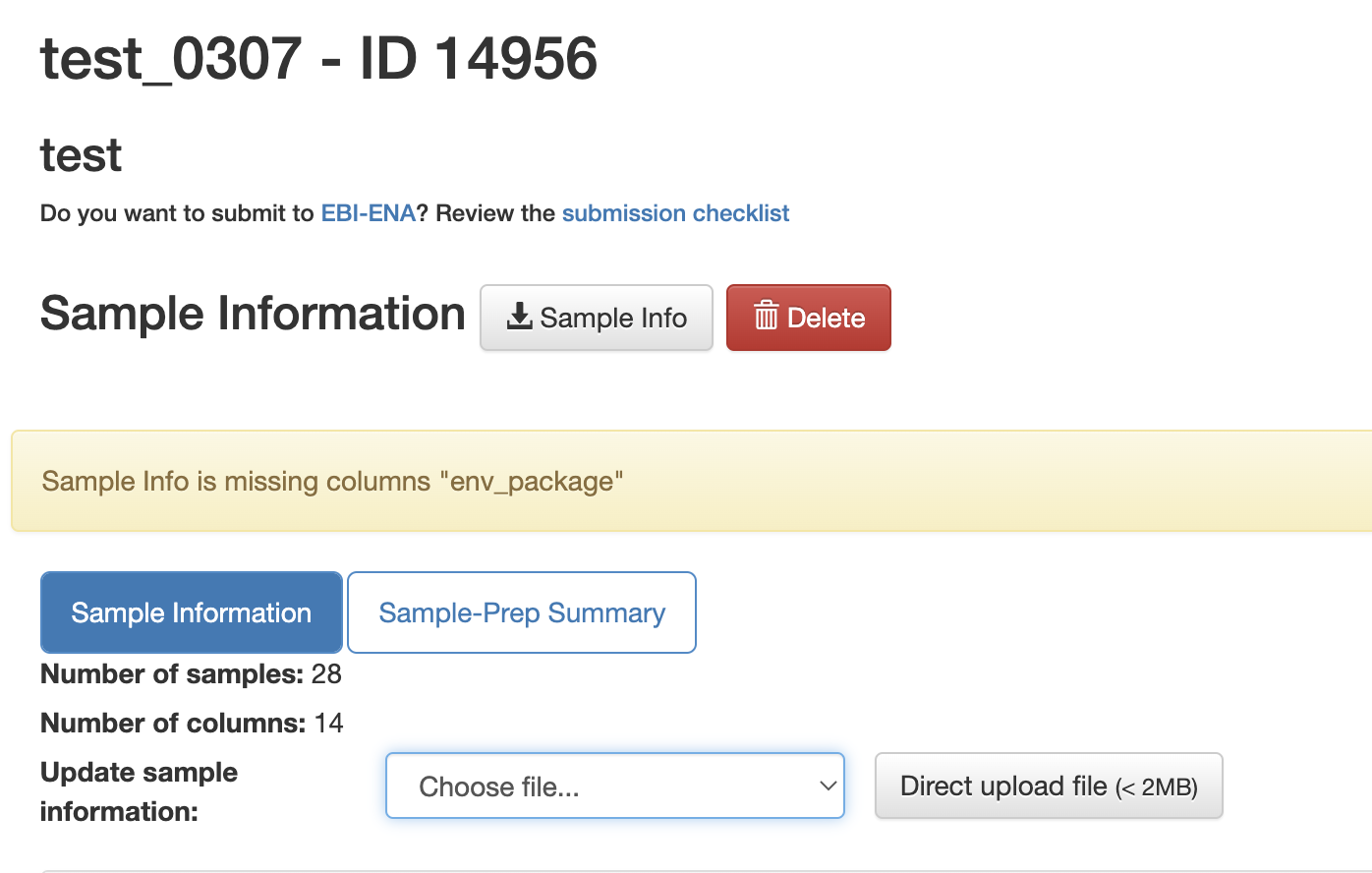
数据基础分析
点击左侧的样品信息,会出现样品分析网络界面,点击Add Default Workflow,再点击Run,可以执行默认的分析操作,包括Split Libraries FASTQ,data trimming,Deblur和Pick closed-reference OTUs操作,最终得到biom格式的feature table
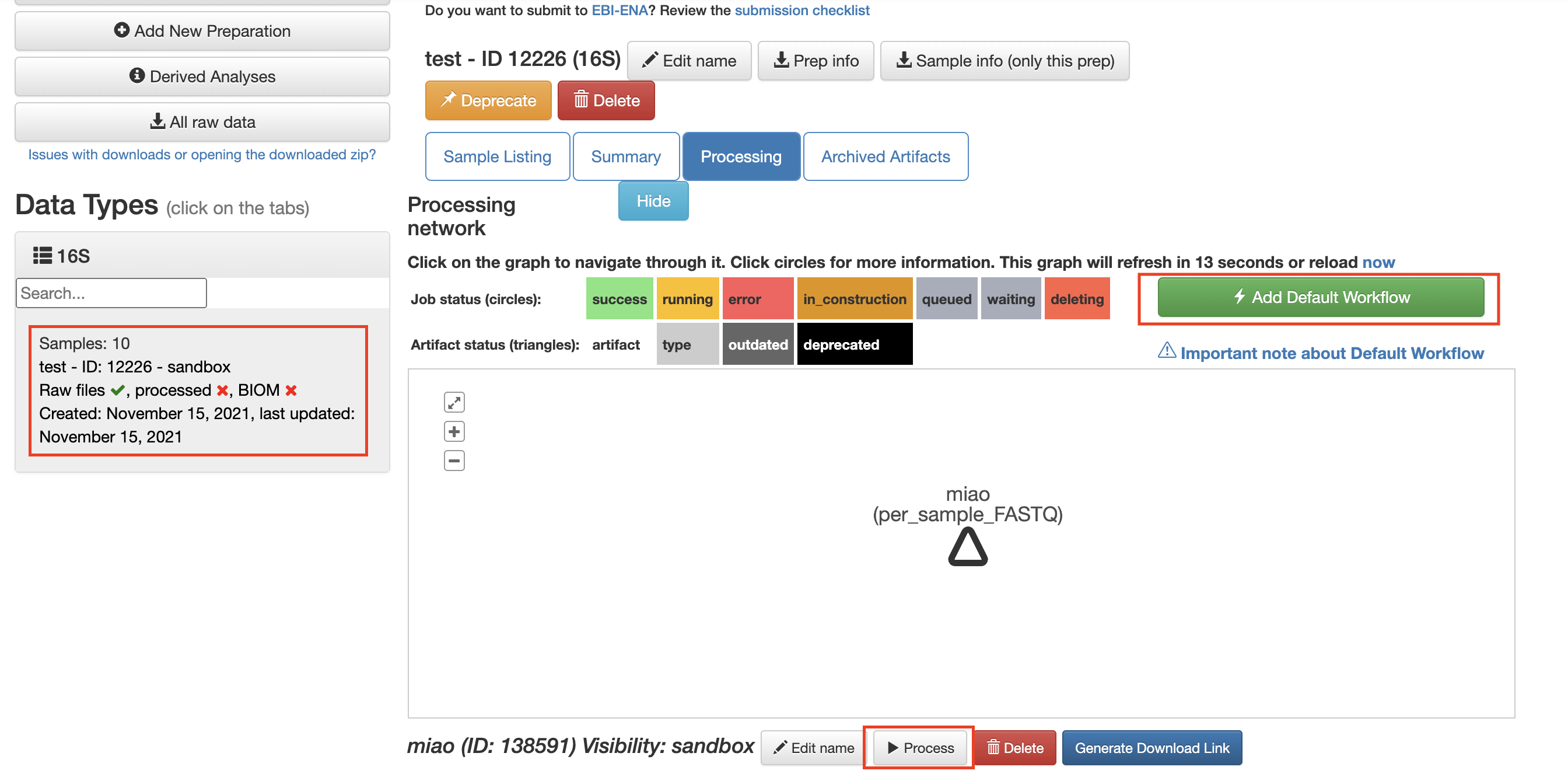
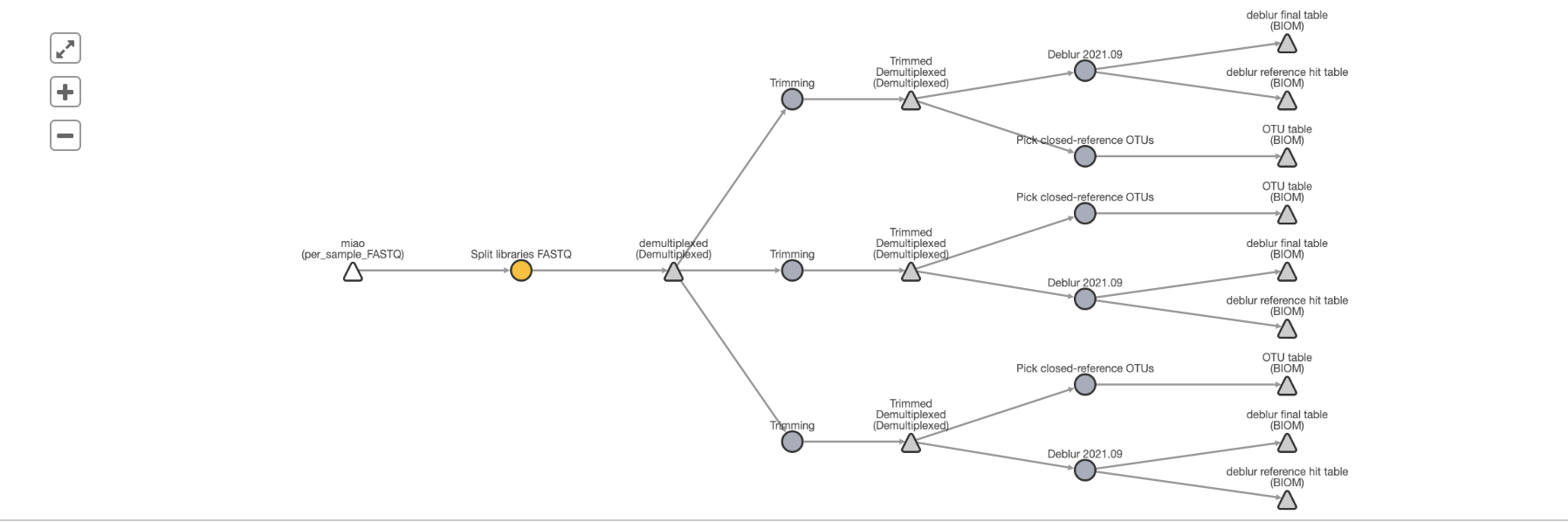
数据进一步分析
点击生成的biom文件(三角符号),点击下方的Add to Analysis按钮,添加数据(可以添加多组数据到同一个analysis中)。Deblur结果通常选择deblur reference hit table添加到analysis中。

添加完成后页面右上角从右数第二个图标会变成绿色,点击这个图标

在这里可以删除选择的样品,最后点击Create Analysis,填写相关信息后点击Create analysis按钮可以生成一个Private的数据分析项目
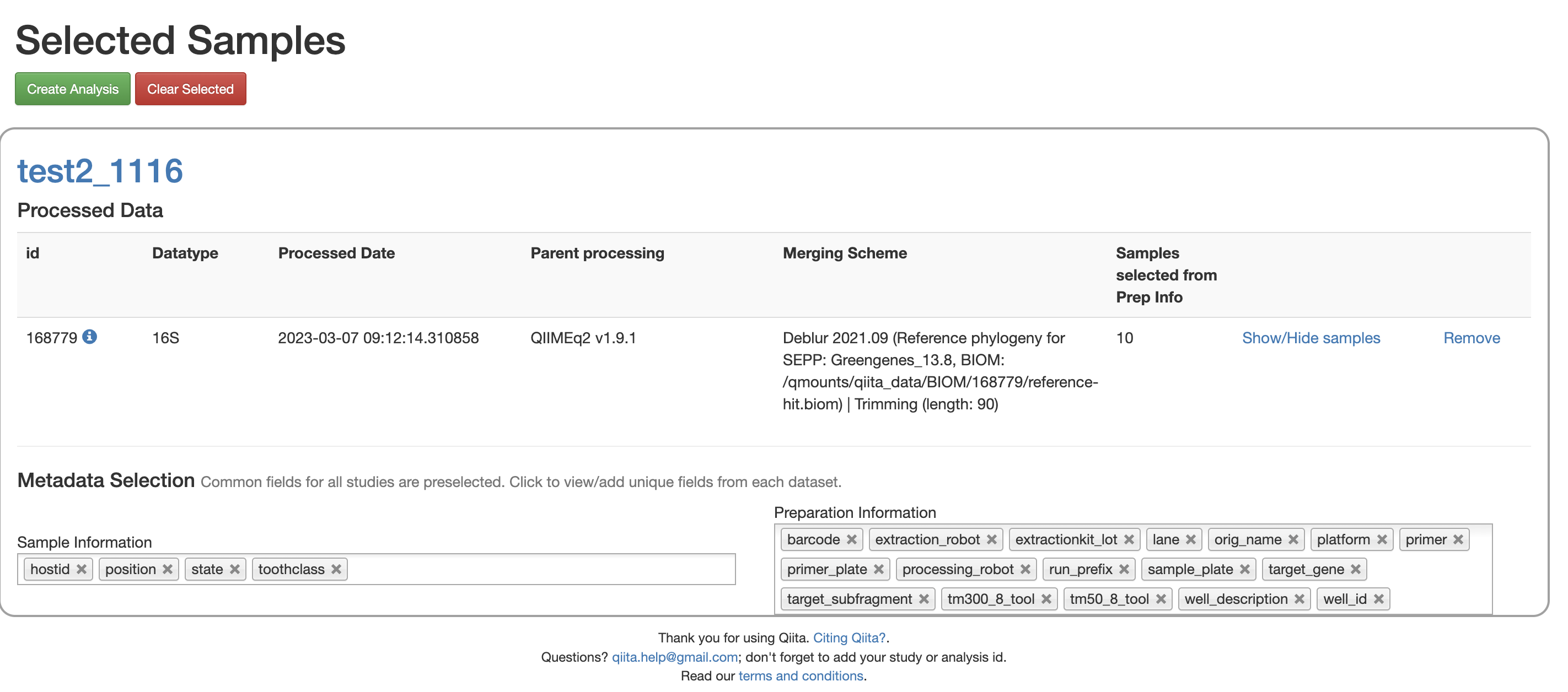
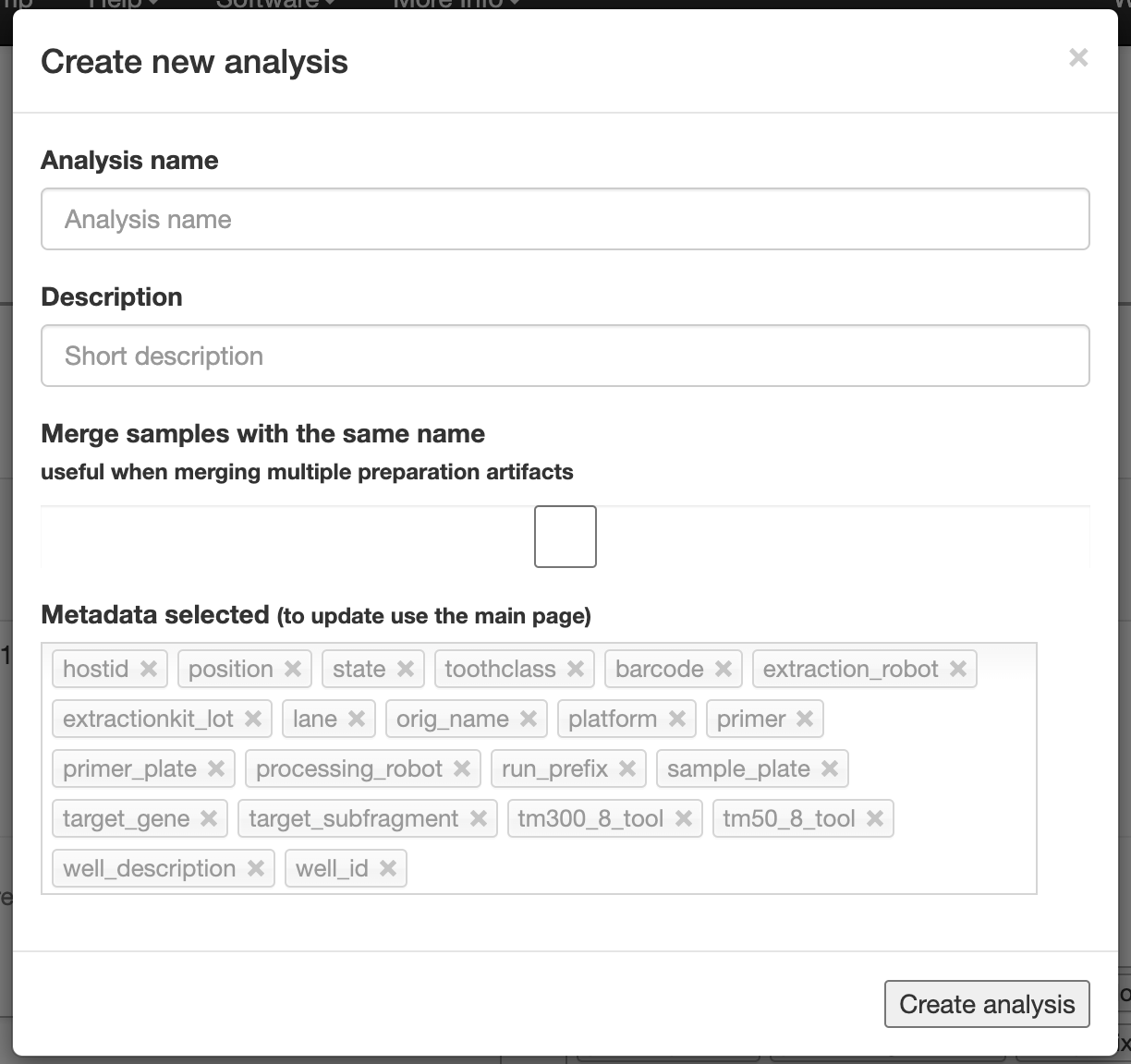
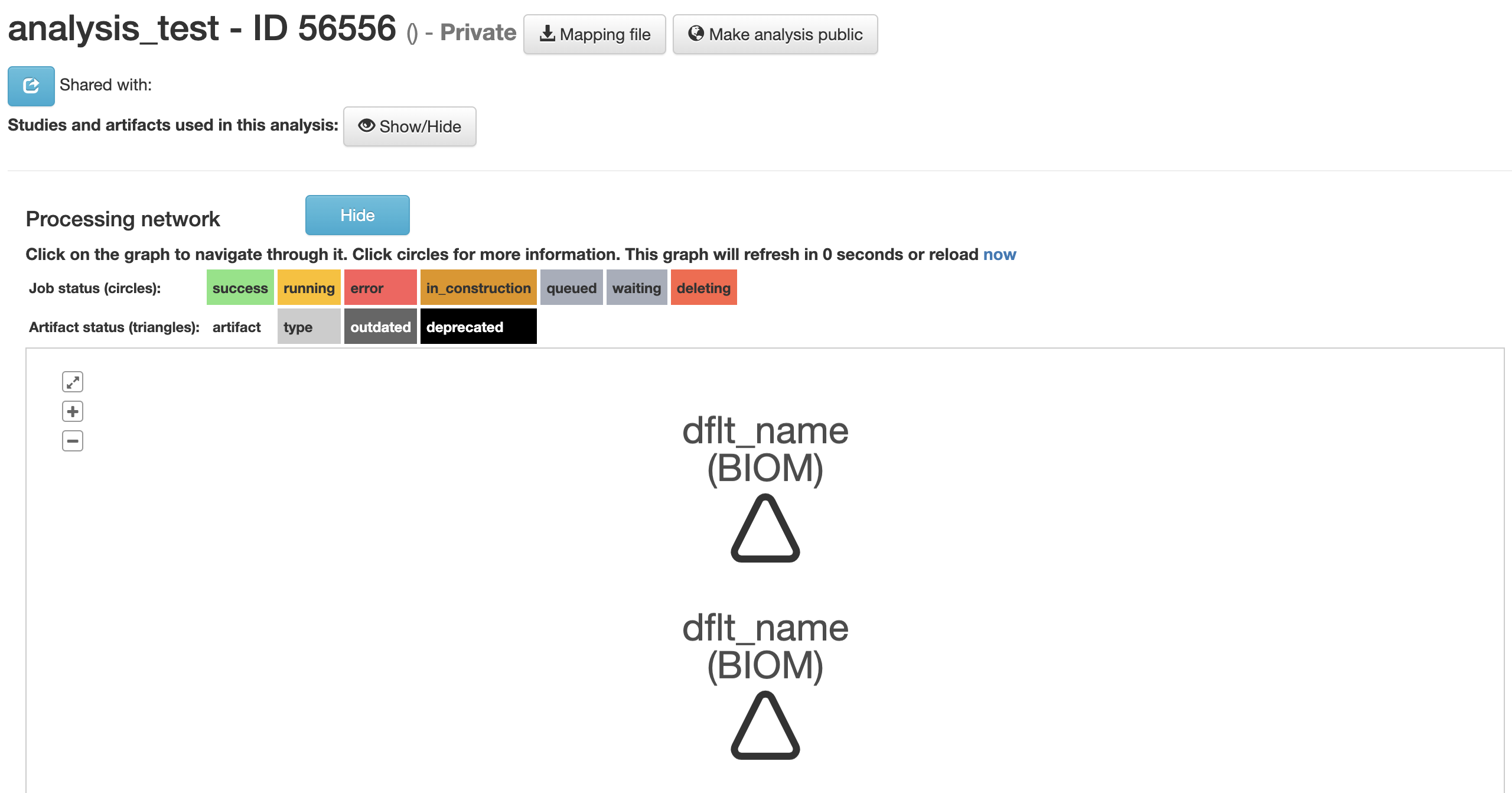
这两个三角一个包括进化树,一个不包括,如果要做涉及到进化树的alpha/beta diversity,应该选择包含进化树的那一个三角形。
已经创建的分析项目,可以通过点击主页的Analysis-》See Previous Analyses查看
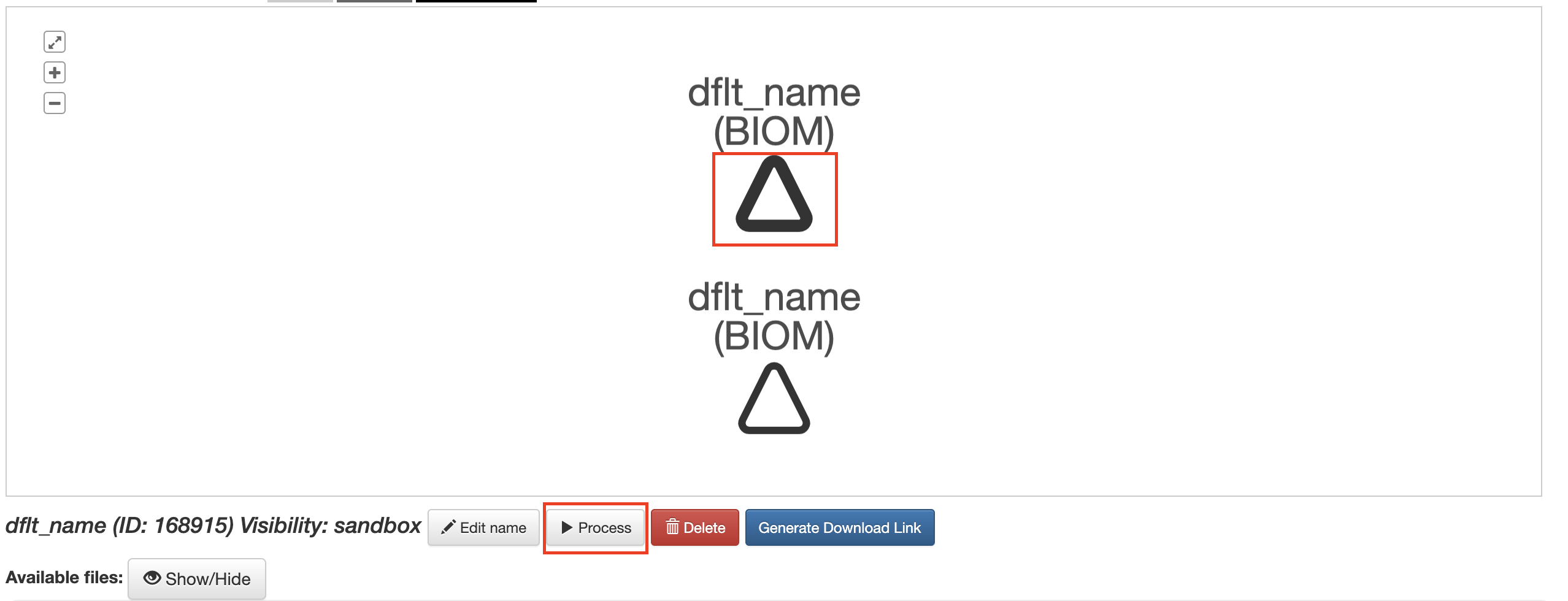
点击图中代表biom文件的三角形,点击下方的Process按钮可以选择要进行的分析操作


这篇关于qiita上传和分析16S rRNA数据的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!






