本文主要是介绍UCSC基因组浏览器用法,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
UCSC基因组浏览器用法
UCSC基因组浏览器是一个强大的在线工具,主要用于查看和分析多种生物的基因组数据。这个浏览器最初是由加利福尼亚大学圣克鲁兹分校的生物信息学家和计算生物学家开发的,旨在为科研人员提供一个易于访问和使用的界面,用于探索和研究基因组数据。
主要特点包括:
-
多物种支持:UCSC基因组浏览器包含了多种物种的基因组数据,包括人类、小鼠、斑马鱼等,用户可以选择不同的物种进行查看。
-
丰富的数据类型:它提供了包括基因组序列、基因、转录本、变异、表达量数据等多种类型的信息,方便用户进行综合分析。
-
自定义查看选项:用户可以自定义浏览器界面,选择感兴趣的数据层进行显示,如基因注释、单核苷酸多态性(SNPs)、表达谱等。
-
交互式界面:浏览器提供了一个交互式的界面,允许用户放大、缩小、滑动来查看特定区域的详细信息。
-
集成分析工具:它集成了多种分析工具,如BLAT(一种快速序列比对工具),帮助用户进行更深入的数据分析。
-
数据共享和下载:UCSC基因组浏览器允许用户上传自己的数据进行查看和分析,并支持将数据导出以供进一步使用。
UCSC基因组浏览器对于基因组学、分子生物学和相关领域的研究人员来说是一个非常有价值的资源,它不仅提供了大量的基因组数据,还提供了强大的分析和可视化工具。
扫码关注微信公众号【生信F3】获取更多生物信息学最新知识。

使用方法
轨道显示模式
UCSC Genome Browser 是以轨道为单位来展示不同基因组信息的,一共包括5种不同的展示方式:hide(隐藏)、dense、squise、pack以及full。在轨道的右侧点击右键即可进行不同的设置,不同的展示效果如下:
dense
将所有信息浓缩为一行。

squise
与 pack 类似,高度只有 pack 一半
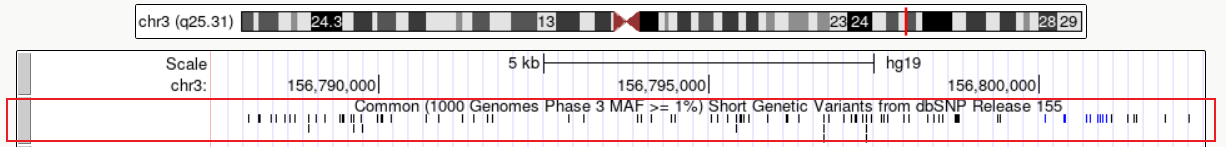
pack
每一项都单独显示,但会尽力合理安排空间
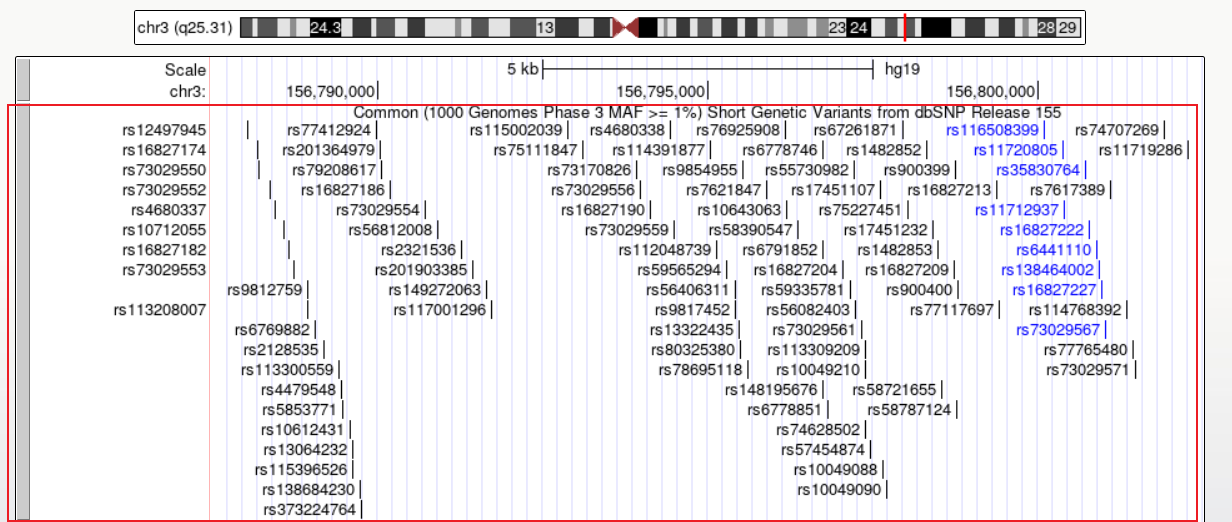
full
完全展开,显示与每个注释功能关联的 wiggle 值,从而创建类似直方图的图像
另外,每一个轨道内的信息,可以点击相对应的内容查看具体信息。比如SNP位点的详细信息。
在Genome Browser当中经常会展示多个轨道的信息。在不同的轨道之间,可以通过拖拽的方式进行排序
利用UCSC基因组浏览器可视化Roadmap数据库
Roadmap Epigenomics Project 数据库是一个旨在收集和提供人类表观基因组数据的重要资源。这个项目是由国家卫生研究院(NIH)的一个计划资助的,其主要目标是揭示人类基因组不同部分的表观遗传状态,以及这些状态如何在不同的细胞类型、组织和疾病状态下变化。这个项目对于理解复杂疾病的基因调控和细胞命运决策至关重要。
下图展示了LEKR1转录起始位点的基因组变异及其在脂肪核中的增强子活性。信号富集轨道来自UCSC基因组浏览器上的ENCODE整合分析和Roadmap表观基因组数据库。转录本来自GENCODE注释:
https://genome.ucsc.edu/cgi-bin/hgTracks?db=hg19&lastVirtModeType=default&lastVirtModeExtraState=&virtModeType=default&virtMode=0&nonVirtPosition=&position=chr3%3A156787499%2D156802500&hgsid=1793209462_pa0Aau4N17cQrLy5nU8J7Dl7qRun
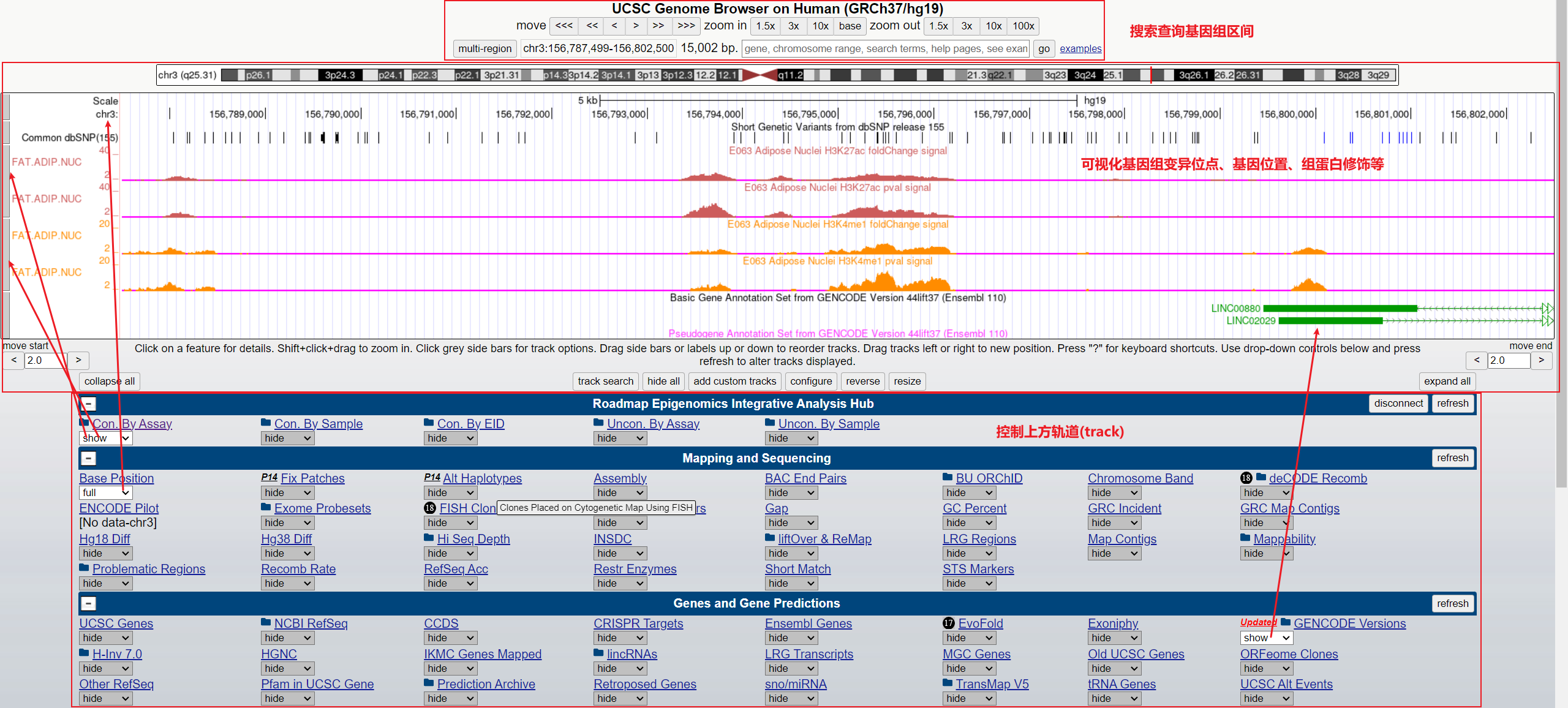
页面从上到下可分为三块:检索查询、基因组可视化、track轨道管理。实现以上可视化需要以下步骤:
1. 定位基因组区间
直接输入查询的染色体和物理位置即可,注意基因组版本号。move左右移动基因结构视图;zoom in放大碱基区域,减少展现出来的区段,宏观查看整个基因组各区域的基因密度;zoom out扩大序列区段,看到周边基因的信息,微观查看一个序列区域内的基因信息;支持用户从科研/教育的角度加入自己的注释信息

2. 设置轨道
参考以下方式设置:
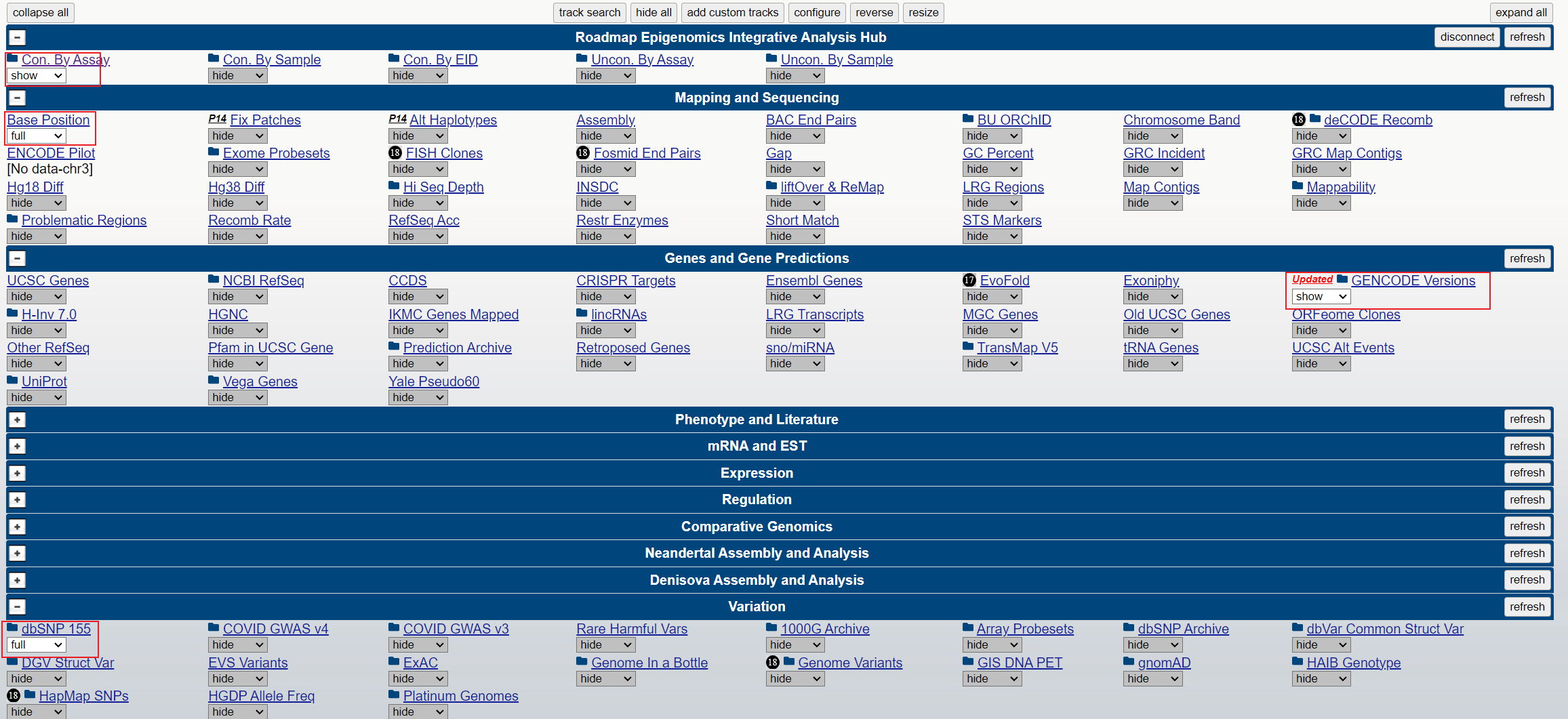
以 Roadmap 数据库为例:

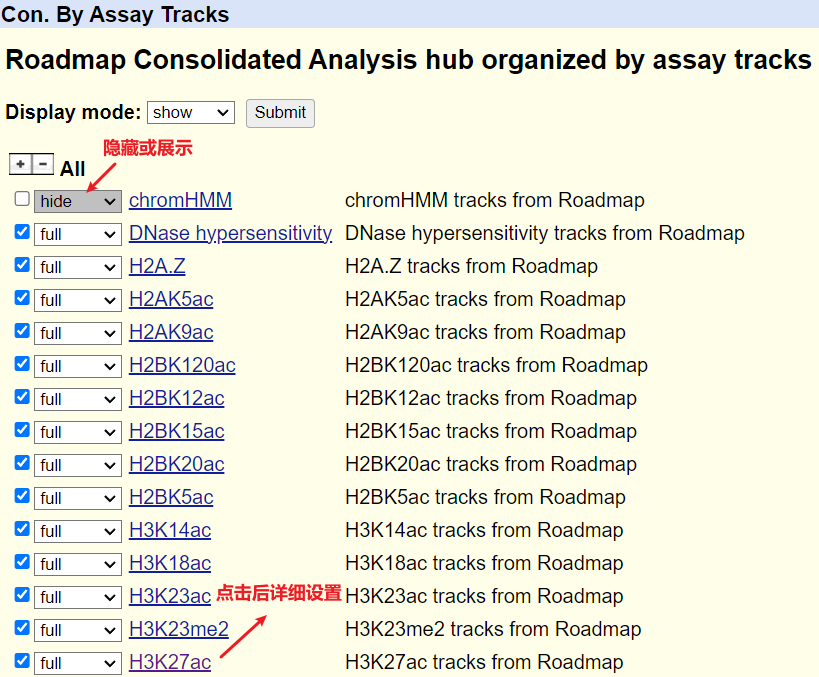
3. 详细设置
单击图标即可设置展示不同组蛋白的数据。
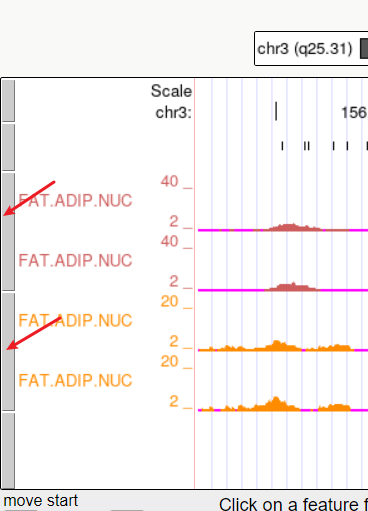
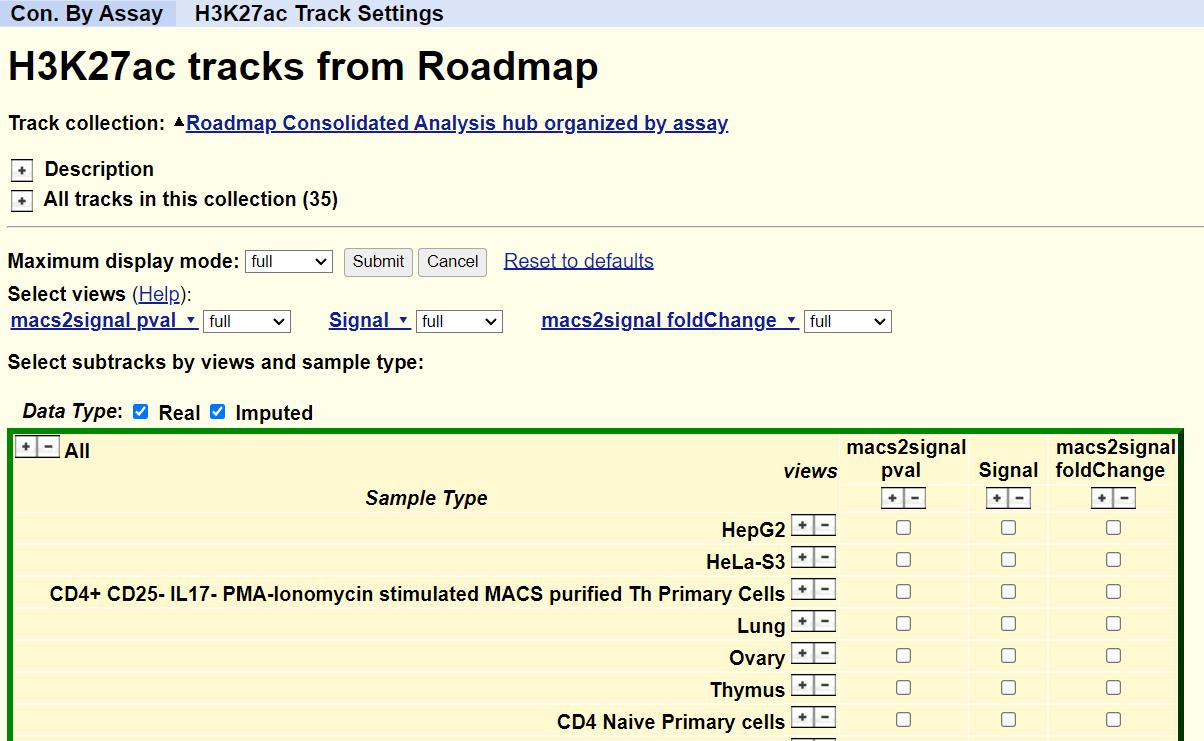
单击下图中任一track均会跳转至该track的管理页面,含有显示设置、track描述、算法、参考文献等;
扫码关注微信公众号【生信F3】获取更多生物信息学最新知识。

本文由 mdnice 多平台发布
这篇关于UCSC基因组浏览器用法的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!






