本文主要是介绍最近3篇蛋白质及其组学知识图谱Nature子刊文章解决生物学核心问题,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
第一篇文章
2022年1月31日Alberto Santos 最新发表在《自然生物技术》上的文章 ” A knowledge graph to interpret clinical proteomics data”. 针对生物医学数据数量大、种类丰富而带来的数据整合困难,该工作提出了一个开源的临床知识图谱平台CKG(Clinical Knowledge Graph), 该平台结合了统计和机器学习算法,加速了典型蛋白质组学工作流程的分析和解释。相比于其他解决方案,CKG平台显得更加友好,将一系列数据库和科学文献信息与omic数据整合到一个易于使用的工作流中,显著增强了科学研究和临床实践的能力。作者开源了相关数据和代码,可以直接迁移到自己的项目中。

将精准医疗应用到临床决策过程中,取决于整合的多组学数据的情况。但是由于生物医学数据的质量与多样性,以及跨不同生物医学数据库和出版物中的扩展性,对数据集成提出了很高的要求。作者构建了临床知识图谱(CKG),这个开源平台目前包含了2000万个节点和2.2亿个关系。图算法提供了一个灵活的数据模型,当新的数据库可用时,该模型很容易扩展到新的节点和关系。CKG结合了统计和机器学习算法,可加速蛋白质组学工作流程的分析和解释。通过一组proof-of-concept生物标志物研究,作者展示了CKG增强和丰富了蛋白质组学数据,并为临床决策提供了关键信息。
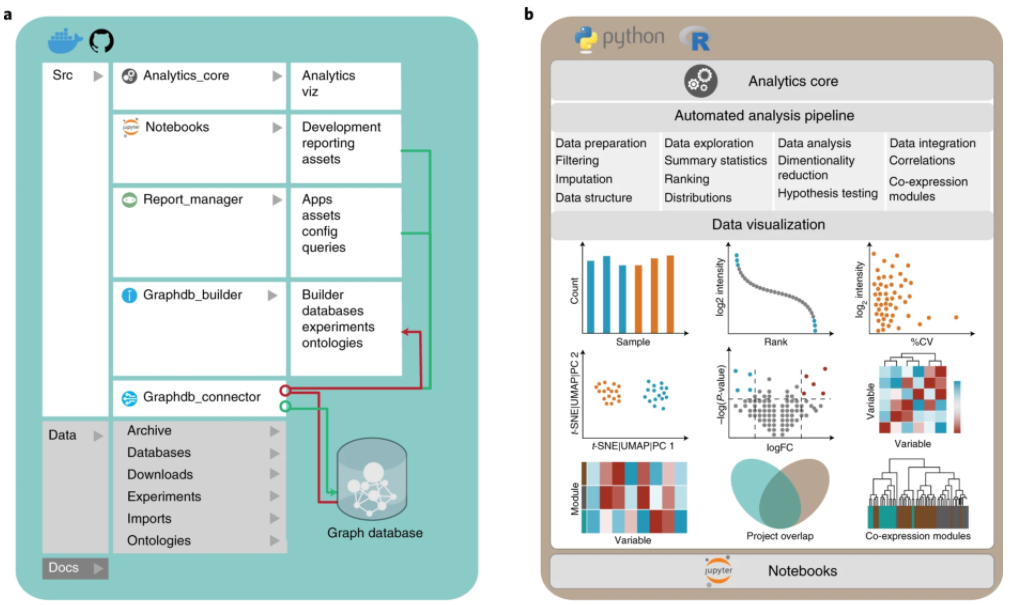
CKG建立在科学的Python库上,具有可靠、可维护性。整个系统是开源的,并取得了MIT许可。它可以在标准工作流程和基于Jupyter笔记本的互动探索中实现可重复、可再现和透明的分析。
CKG包括几个独立的功能模块:
(1)格式化和分析蛋白质组学数据(analytics_core);
(2)通过整合来自一系列可公开访问的数据库、用户进行的实验、现有本体和科学出版物的可用数据,构建一个图形数据库(graphdb_builder);
(3)连接和查询这个图形数据库(graphdb_connector);
(4)通过在线报告(report_manager)和Jupyter notebooks促进数据可视化、存储和分析(图1a,b)。这个架构无缝地协调和整合了数据以及用户提供的分析。它还促进了数据共享和可视化,以及基于详细的生物医学知识注释的统计报告的解释,产生了临床相关的结果。
最新3篇发表顶刊蛋白质组学知识图谱相关论文,开源了相关数据和代码第一篇文章2022年1月31日Alberto Santos 最新发表在《自然生物技术》上的文章 ” A knhttps://mp.weixin.qq.com/s?__biz=MzIzOTA2NTQ5NA==&mid=2247484284&idx=1&sn=6c9f4dabad93a2920129a3c5e74f95f0&chksm=e92e8385de590a930ceec855fbc20e2eedd3f4bba238f1c73362aa0a374305bc93ef97f9cca0&token=664256073&lang=zh_CN#rd
这篇关于最近3篇蛋白质及其组学知识图谱Nature子刊文章解决生物学核心问题的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







