本文主要是介绍PSP - HHblits 算法搜索 BFD 与 UniRef30 的结果分析 (bfd_uniref_hits.a3m),希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
欢迎关注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://spike.blog.csdn.net/article/details/132047940
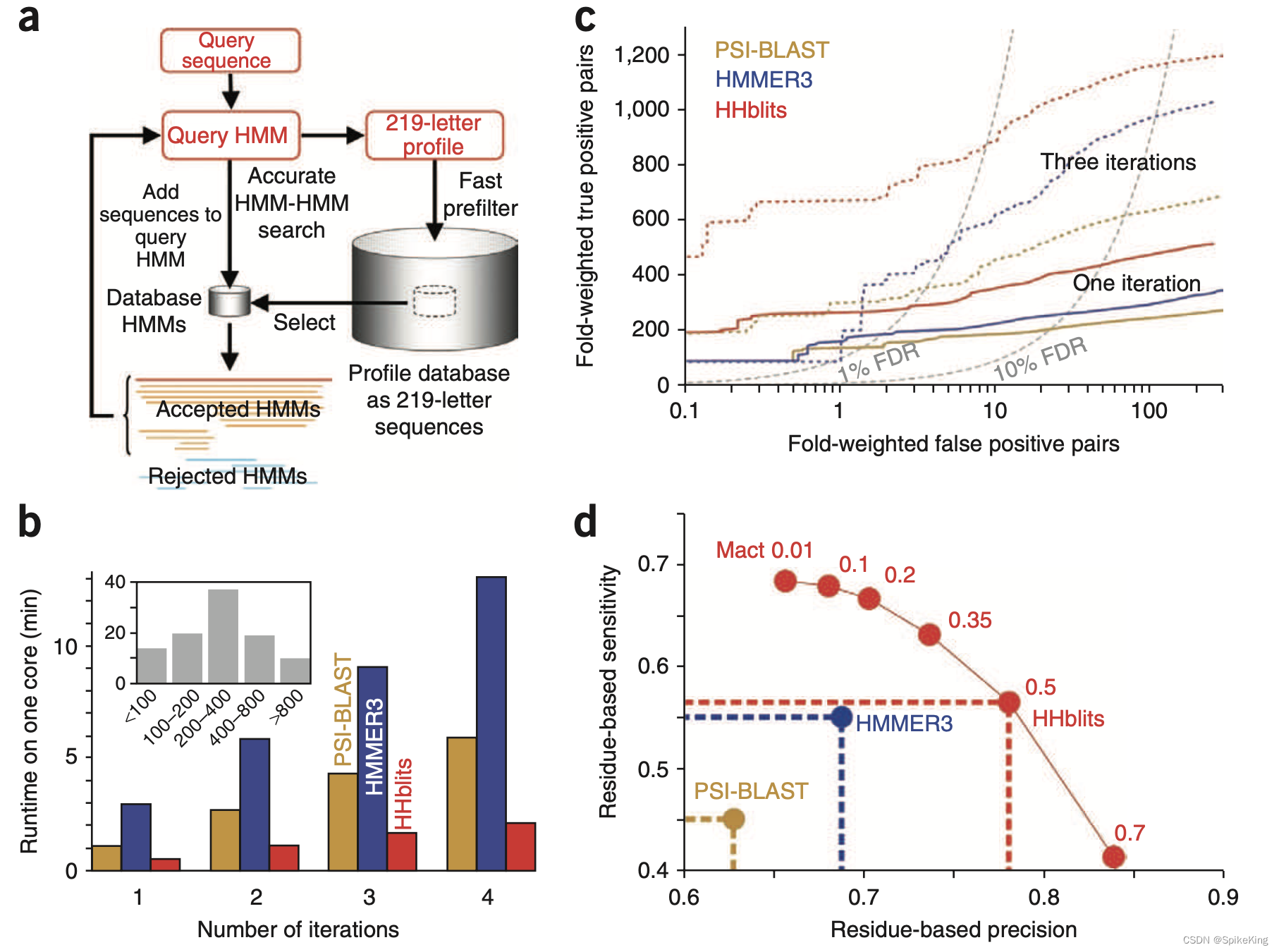
MMseqs2 与 HHblits 的算法比较:
- 蛋白质序列搜索算法 MMseqs2 与 HHblits 的搜索结果差异
- HHblits 算法搜索 BFD 与 UniRef30 的结果分析
Paper: HHblits: lightning-fast iterative protein sequence searching by HMM-HMM alignment
- 2011.12 Nature Methods
HHblits 是一种基于 HMM-HMM 对齐的迭代蛋白质序列搜索工具,可以快速、灵敏、准确地找到数据库中与查询序列相似的蛋白质序列。HHblits 的主要特点有:
- 使用隐马尔可夫模型(HMM)来表示查询序列和数据库序列,而不是使用简单的氨基酸序列。HMM 是一种统计模型,可以捕捉序列的进化变化和保守区域,从而提高序列相似性的检测能力。
- 使用一种离散化的预过滤器,可以快速地筛选出与查询序列有潜在相似性的数据库序列,从而减少了 HMM-HMM 对齐的计算量。这种预过滤器可以将搜索时间提高 2500 倍,而不损失搜索灵敏度。
- 使用迭代的方法,可以利用上一轮搜索得到的多序列比对(MSA)来构建更精确的查询 HMM,来进行下一轮搜索。这样可以不断地扩展搜索范围,找到更多的相关序列。
在 bfd_uniref_hits.a3m 中,以 UniRef100 开头的序列描述,即来源于 UniRef30 数据库,即:
>UniRef100_A0A2X3SLP5 Uncharacterized protein n=2 Tax=Streptococcus equi TaxID=1336 RepID=A0A2X3SLP5_STRSZ
-TNtsSQEVDQVAQALELMFDNNVSTSNFKKYVNNNFSDSEIAIAELELESRISNSrsefrvawnemggCIAGKIRDEFFAMISVGTIVKYAQKKAWKELALVVLKFVKANGLKTNAIIVAGQLALWAVQCG
a3m文件格式是用于表示蛋白质序列比对的文本格式,是 FASTA 格式的一种扩展。在a3m文件格式中,蛋白质序列用单个字母表示,其中,匹配用大写字母表示,删除用横线(-) 表示,插入用小写字母表示,去掉小写字母,只保留大写字母与横线(-) ,则长度与目标序列相同。
其他序列就是来源于 BFD,以 SRR 开头的数量较多,即:
>SRR5690606_14355373
-----QIEELAAQLEFLMEEALiiENGERTfdfEKIENEFgkeVKDEIKMLTVDAQVWqvqpgaitlaanqPWKDCMVGAIKDHFGvALVTAaleGGLWAYLEKKAYKEAAKLLVKF----AVGTNAVGIAGTLIYYGGKCT
...
>SoimicmetaTmtLPB_FD_contig_61_1212631_length_209_multi_1_in_0_out_0_1 # 2 # 76 # 1 # ID=3042713_1;partial=10;start_type=Edge;rbs_motif=None;rbs_spacer=None;gc_cont=0.693
---NEEIEQLAADLEFLMEEAAiydEKGKVVnfdfDLLEERFgYVLELEMLKEEIEAYnattegd--NDeiqlfswksCMISALKGHFGvALIEValtGGLWSYLEKKAYKEAAKLLLKI----GIEGNVIGLTAFLTWYSVDCI
...
>APFre7841882654_1041346.scaffolds.fasta_scaffold441692_2 # 291 # 482 # 1 # ID=441692_2;partial=01;start_type=ATG;rbs_motif=AGGAG;rbs_spacer=5-10bp;gc_cont=0.609
-EIPLEAqgisiFGANHCDGareserliahelAHQWFgnsvtakrwrhiwlhegFACYAEWLWSEHSGdrsaDEWAhhfHEKLASSPQDLLLADPGPRDMFddrvykrgalTLHVLRRTLGDENFFALLKDWTSRHRHGSAVTD------DFTGLAANYTDQSLRPLWDAWLYS--TEVPALDAESX-------------------------------------------------------------------------------------------------------------------
...
因此,使用 MMseqs2 分别搜索 BFD 和 UniRef30,再合并,与使用 HHblits 一起搜索两个库的效果相同,同时也可以计算不同搜索算法的结果差异,即:
- 测试序列来源于 CASP15 提供的官方序列。
- MMseqs2 的搜索参数,
num_iterations是 3,sensitivity是 8,即i3s8,其余默认。 - HHblits 使用 AF2 默认的搜索工具,数据库也保持一致。
即:

源码如下:
#!/usr/bin/env python
# -- coding: utf-8 --
"""
Copyright (c) 2022. All rights reserved.
Created by C. L. Wang on 2023/8/1
"""
import osfrom myutils.project_utils import read_file
from root_dir import DATA_DIRclass BfdUniref30Overlap(object):"""计算 MMseqs2 与 HHblits 搜索结果之间的重叠度"""def __init__(self):pass@staticmethoddef count_seqs(data_lines):return len(data_lines) // 2 - 1def split_hhblits(self, data_lines):"""拆分 Uniref30 与 BFD 数据"""n = len(data_lines)n = n // 2 * 2u_desc_list, u_seq_list = [], []b_desc_list, b_seq_list = [], []for i in range(2, n, 2):desc = data_lines[i][1:]seq = data_lines[i+1]seq = seq.replace("-", "")if desc.startswith("UniRef100"):desc = desc.split(" ")[0]u_desc_list.append(desc)u_seq_list.append(seq)else:items = desc.split("|")if len(items) > 1:desc = items[1]b_desc_list.append(desc)b_seq_list.append(seq)unique_u_seq = len(list(set(u_seq_list)))unique_u_desc = len(list(set(u_desc_list)))print(f"[Info] unique uniref30 desc: {unique_u_desc} / {len(u_desc_list)}")print(f"[Info] unique uniref30 seqs: {unique_u_seq} / {len(u_seq_list)}")unique_b_seq = len(list(set(b_seq_list)))unique_b_desc = len(list(set(b_desc_list)))print(f"[Info] unique bfd desc: {unique_b_desc} / {len(b_desc_list)}")print(f"[Info] unique bfd seqs: {unique_b_seq} / {len(b_seq_list)}")return u_desc_list, u_seq_list, b_desc_list, b_seq_listdef process(self, uniref30_path, bfd_path, hhblits_path):assert os.path.isfile(uniref30_path) and os.path.isfile(bfd_path) and os.path.isfile(hhblits_path)print(f"[Info] mmseqs uniref30: {uniref30_path}")print(f"[Info] mmseqs bfd: {bfd_path}")print(f"[Info] hhblits bfd and uniref30: {hhblits_path}")uniref30_lines = read_file(uniref30_path)bfd_lines = read_file(bfd_path)hhblits_lines = read_file(hhblits_path)print(f"[Info] uniref30: {self.count_seqs(uniref30_lines)}, bfd: {self.count_seqs(bfd_lines)}, "f"hhblits: {self.count_seqs(hhblits_lines)}")# ---------- 统计 MMseqs2 Uniref30 ---------- #u_desc_list, u_seq_list = [], []n = len(uniref30_lines)n = n // 2 * 2for i in range(2, n, 2):u_name = uniref30_lines[i][1:].split("\t")[0]u_seq = uniref30_lines[i+1].replace("-", "")assert u_name.startswith("UniRef100")u_desc_list.append(u_name)u_seq_list.append(u_seq)assert len(u_desc_list) == self.count_seqs(uniref30_lines)unique_u_seq = len(list(set(u_seq_list)))unique_u_desc = len(list(set(u_desc_list)))print(f"[Info] unique uniref30 desc: {unique_u_desc} / {len(u_desc_list)}")print(f"[Info] unique uniref30 seqs: {unique_u_seq} / {len(u_seq_list)}")# ---------- 统计 MMseqs2 Uniref30 ---------- ## ---------- 统计 BFD Uniref30 ---------- #b_desc_list, b_seq_list = [], []n = len(bfd_lines)n = n // 2 * 2for i in range(2, n, 2):b_name = bfd_lines[i][1:].split("\t")[0]b_seq = bfd_lines[i+1].replace("-", "")b_desc_list.append(b_name)b_seq_list.append(b_seq)assert len(b_desc_list) == self.count_seqs(bfd_lines)unique_b_seq = len(list(set(b_seq_list)))unique_b_desc = len(list(set(b_desc_list)))print(f"[Info] unique bfd desc: {unique_b_desc} / {len(b_desc_list)}")print(f"[Info] unique bfd seqs: {unique_b_seq} / {len(b_seq_list)}")# ---------- 统计 BFD Uniref30 ---------- ## ---------- 统计 HHblits BFD and Uniref30 ---------- #hu_desc_list, hu_seq_list, hb_desc_list, hb_seq_list = self.split_hhblits(hhblits_lines)assert len(hu_desc_list) + len(hb_desc_list) == self.count_seqs(hhblits_lines)# ---------- 统计 HHblits BFD and Uniref30 ---------- ## ---------- 统计 交集 ---------- #num_u_desc_is = len(set(u_desc_list).intersection(set(hu_desc_list)))print(f"[Info] uniref30 desc intersection: {num_u_desc_is}")num_u_seqs_is = len(set(u_seq_list).intersection(set(hu_seq_list)))print(f"[Info] uniref30 seqs intersection: {num_u_seqs_is}")num_b_desc_is = len(set(b_desc_list).intersection(set(hb_desc_list)))print(f"[Info] bfd desc intersection: {num_b_desc_is}")num_b_seqs_is = len(set(b_seq_list).intersection(set(hb_seq_list)))print(f"[Info] bfd seqs intersection: {num_b_seqs_is}")# ---------- 统计 交集 ---------- ## ---------- 统计 总数 ---------- #unique_mmseqs_seqs = list(set(u_seq_list + b_seq_list))unique_hhblits_seqs = list(set(hu_seq_list + hb_seq_list))print(f"[Info] unique_mmseqs_ses: {len(unique_mmseqs_seqs)}")print(f"[Info] unique_hhblis_ses: {len(unique_hhblits_seqs)}")# ---------- 统计 总数 ---------- #def main():fasta_name = "T1104-D1_A117"# fasta_name = "T1137s8-D1_A251"# fasta_name = "T1188-D1_A573"# fasta_name = "T1157s1_A1029"uniref30_path = os.path.join(DATA_DIR, "overlap", fasta_name, f"{fasta_name}-uniref30-i3s8.a3m")bfd_path = os.path.join(DATA_DIR, "overlap", fasta_name, f"{fasta_name}-bfd-i3s8.a3m")hhblits_path = os.path.join(DATA_DIR, "overlap", fasta_name, "bfd_uniref_hits.a3m")buo = BfdUniref30Overlap()buo.process(uniref30_path, bfd_path, hhblits_path)if __name__ == "__main__":main()
这篇关于PSP - HHblits 算法搜索 BFD 与 UniRef30 的结果分析 (bfd_uniref_hits.a3m)的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







